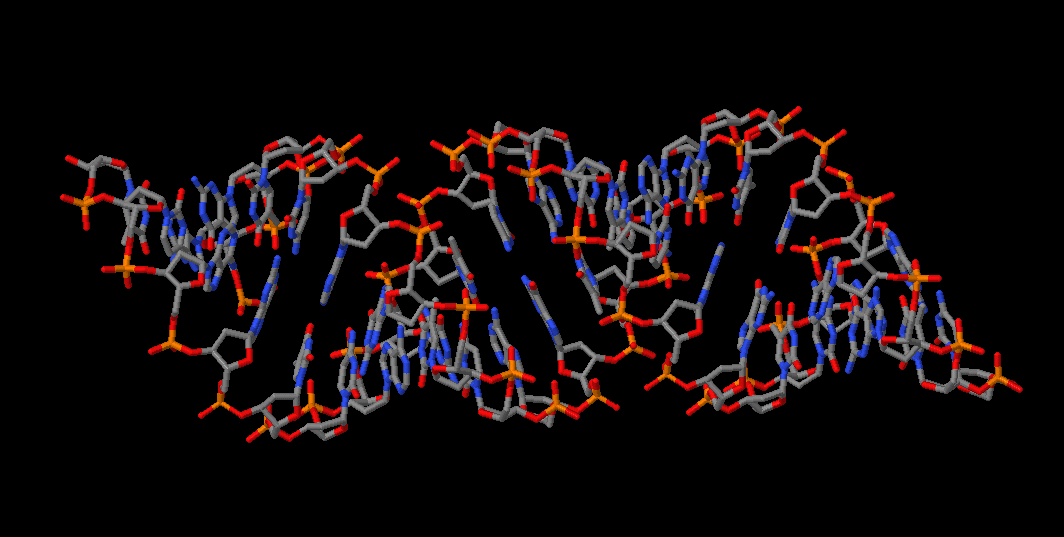
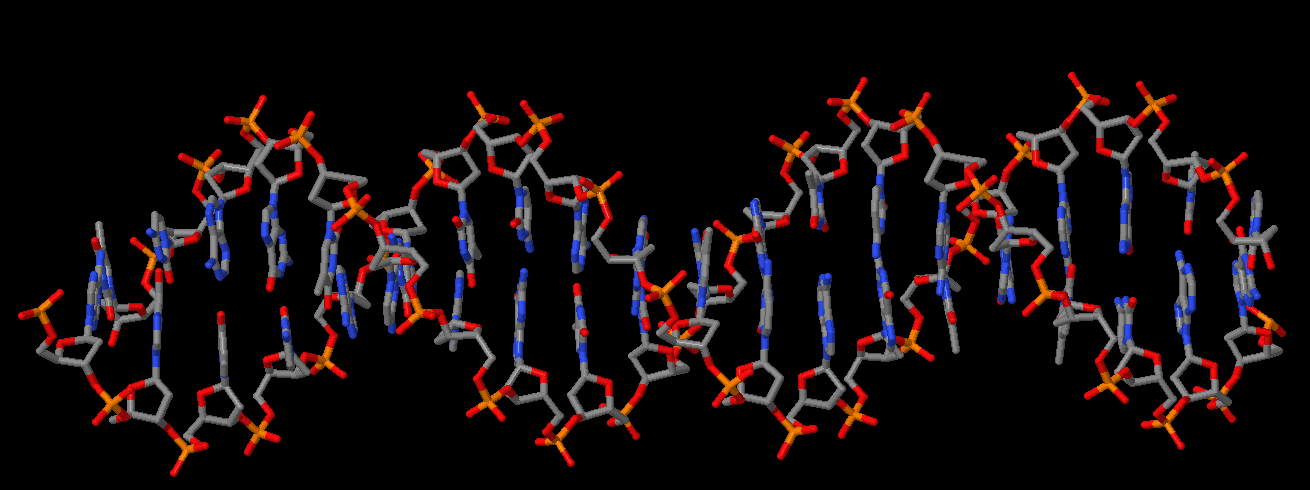
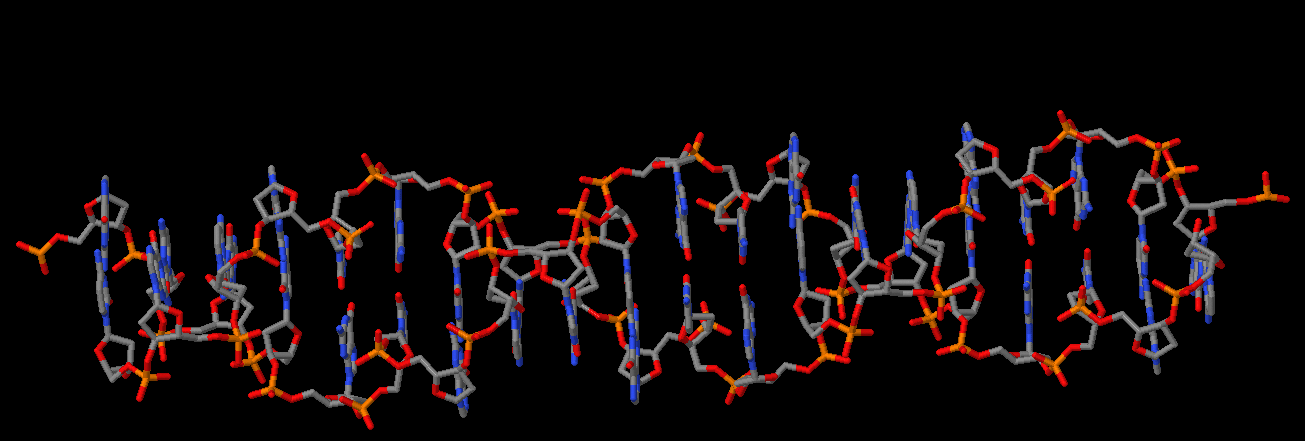
С помощью программы fiber пакета 3DNA были построены A-, B- и Z-формы ДНК, состоящие из повторяющейся последовательности GATC (У Z-формы - GC). В результате я получила файлы gatc-a.pdb, gatc-b.pdb и gatc-z.pdb. Ниже представлены изображения трёх форм ДНК в программе Jmol A, B и Z соответственно.
Формы ДНК
| A | B | Z |
 |  |  |
На примере структуры B-формы ДНК в программе Jmol я определила, какие атомы азотистого основания гуанина смотрят в сторону большой бороздки, а какие - в сторому малой бороздки.
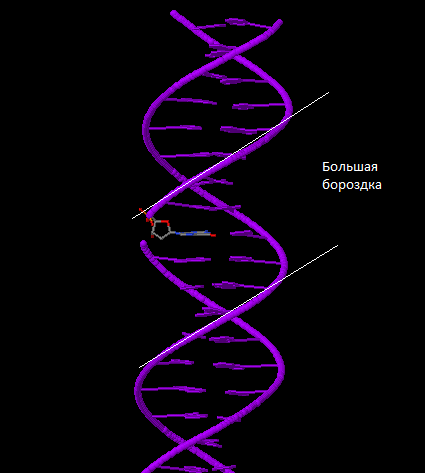 | 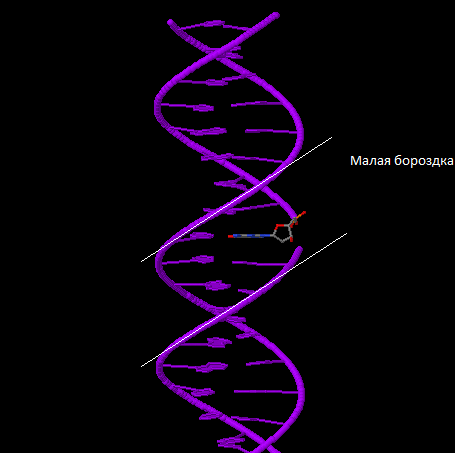 | |
С помощью программы MarvinSketch было получено следующее изображение (красным цветом отмечены атомы аденина явно смотрящие в сторону большой бороздки, а синим - в сторону малой бороздки).
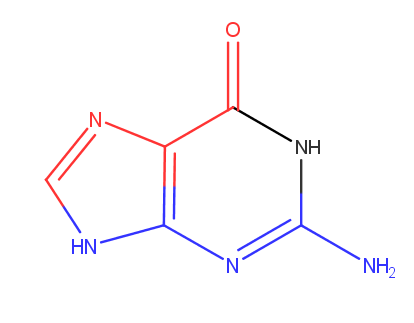
Сравнение обращения атомов гуанина в стороны бороздок у разных форм ДНК |
|||
А-форма ДНК |
В-форма ДНК |
Z-ворма ДНК |
|
Атомы, обращенные в сторону большой бороздки |
C8, N7 |
C6, O6, C5, N7, C8 |
C2, N3 |
Атомы, обращенные в сторону малой бороздки |
N3, N9, C2, N1, C4 |
C4, N2, C2, N3, N9 |
С6, C5, C4, N9, C8, N7 |
Остальные атомы |
C5, C6 |
N1 |
N1 |
Сравнение основных спиральных параметров разных форм ДНК |
|||
| Форма ДНК | А-форма |
В-форма |
Z-форма |
Тип спирали |
правая |
правая |
левая |
Шаг спирали (А) |
28.03 |
33.75 |
48.5 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
1.681 нм |
1.721 нм |
1.830 нм |
Ширина малой бороздки |
0.798 нм |
1.169 нм |
0.720нм |
С помощью команды Torsion в программе Jmol я измерила торсионные углы нуклеотида, содержащего гуанин, в A- и B-формах ДНК. Результаты приведены в таблице ниже.
Сравнение торсионных углов нуклеотида, содержащего аденин, в разных формах ДНК |
|||||||
Значения из презентации |
|||||||
| Форма ДНК | α |
β |
γ |
δ |
ε |
ζ |
Χ |
A |
62 |
173 |
52 |
88 |
178 |
-50 |
-160 |
B |
63 |
171 |
54 |
131 |
155 |
-90 |
-117 |
Значения, полученные с помощью Jmol |
|||||||
A |
64.1 |
174.8 |
41.7 |
79.0 |
-147.8 |
-75.1 |
-157.2 |
B |
85.9 |
136.4 |
31.1 |
143.4 |
140.8 |
-160.5 |
-98.0 |
Далее был проведен анализ структур нуклеиновых кислот (А-, В- и Z-форм ДНК, тРНК с PDB ID 1n78 и ДНК с PDB ID 1i3j) с помощью программ find_pair и analyze пакета 3DNA (т.к. он работает только со старым PDB форматом, скачанные файлы были переведены в нужный формат командой remediator), В результате будет создан ряд файлов с описанием разных параметров структуры. Необходимая информация (значения торсионных углов и параметры водородных связей), находится в файлах формата XXXX.out. Средние значения каждого из торсионных углов (без рассмотрения краевых нуклеотидов) были найдены в программе Excel (файл с вычислениями) и указаны в таблице ниже в градусах.
| Угол | α | β | γ | δ | ε | ζ | Χ |
| A-ДНК | -51,70 | 174,80 | 41,70 | 79,00 | -147,80 | -75,10 | -157,20 |
| B-ДНК | -29,90 | 136,40 | 31,10 | 143,40 | -140,80 | -160,50 | -98,00 |
| Z-ДНК(С) | -139,50 | -136,80 | 50,90 | 137,60 | -96,50 | 82,00 | -154,30 |
| Z-ДНК (G) | 51,90 | 179,00 | -173,80 | 94,90 | -103,60 | -64,80 | 58,70 |
| тРНК (PDB ID 1n78) | -44,34 | 60,23 | 51,76 | 85,76 | -135,01 | -60,75 | -147.45 |
| ДНК (PDB ID 1i3j) | -22,98 | -29,51 | 8,03 | 148,66 | -80,84 | -115,98 | -98,19 |
Наиболее схожие значения торсионных углов наблюдаются у А- и В-форм ДНК (наибольшая разница у углов зета - 85.4 градуса и дельта - 64.2 градуса), что дает возможность судить об их сходстве. Z-форма ДНК (как для гуанина, так и для цитозина) существенно отличается от А- и В-форм и друг от друга. Т-РНК (PDB ID 1n78) по средним значениям торсионных углов больше всего похожа на А-ДНК. Самый "деформированный" нуклеотид в тРНК - 42 аденин второй цепи. Он больше всех остальных отклоняется по торсионному углу гамма и эпсилон , а также в значительной степени по углам альфа, бета и зета.
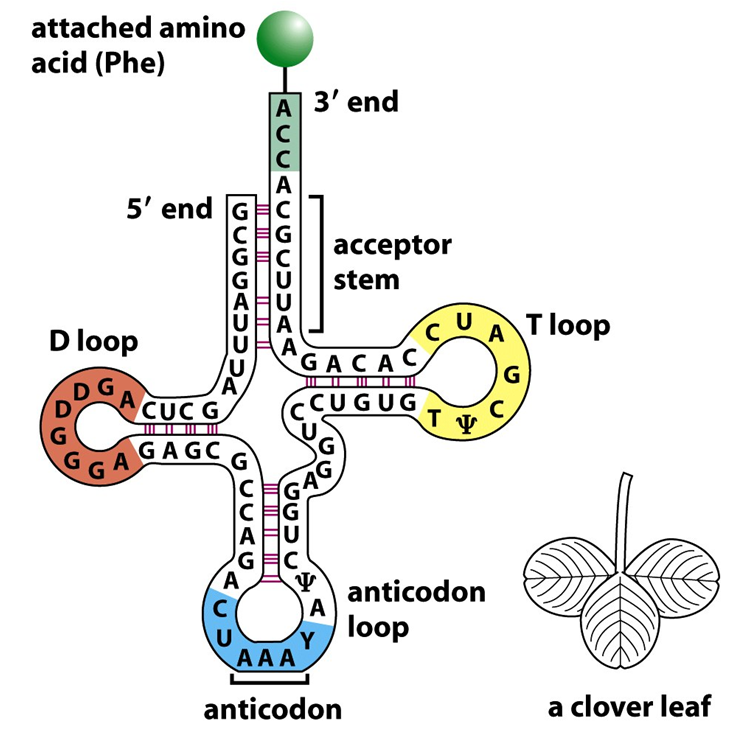
Изучим структуру тРНК (PDB ID 1n78):
Данные для определения положения "стеблей" в структуре были взяты из файла 1n78_old.out. "Стеблевые" нуклеотиды идут по порядку. Далее на базе информации, полученной из файла, в программе JMol были окрашены соответствующие участки (Зеленый - акцепторный "стебель", желтый - Т-стебель, оранжевым - D-стебель, синим - антикодоновый стебель, остальное - красный(связи не образуют стебли)).
 | 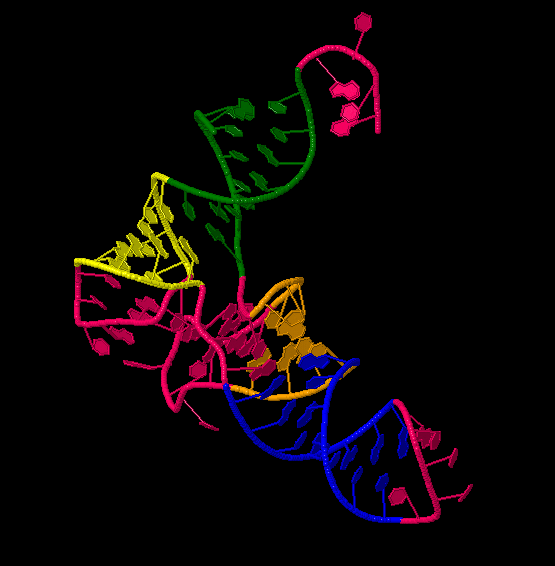 |  |  |
Структура также содержит 9 неканонических пар нуклеотидов:
Рассмотрим стекинг-взаимодействия. В файле 1n78_old.out содержатся данные по структуре тРНК, в том числе данные о величине площади "перекрывании" 2-х последовательных пар азотистых оснований.
Стэкинг-взаимодействие с большой вероятностью будет у пар с большой площадью перекрытия. Наибольшее и наименьшие значения у следующих пар:
2 GC/GU 7.23( 4.42) 0.00( 0.00) 0.00( 0.00) 6.56( 3.95) 13.79( 8.36).
21 AG/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
28 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
29 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
43 UA/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00
В файле stacking.pdb хранится информация о структуре всех комплементарных пар РНК, записанных как отдельные модели. С помощью команды stack2img (с параметром cdolt) на основе модели структуры пары с наибольши перекрытием было построено изображение.
Для большей наглядности и проверки взаимной ориентации пары были также рассмотрены в программе JMol.
Для сравнения также были получены изображения пар с наименьшим перекрытием (21 AG/CG).
| stack2img | JMol |
| Пары азотистых оснований с наибольшим перекрытием | |
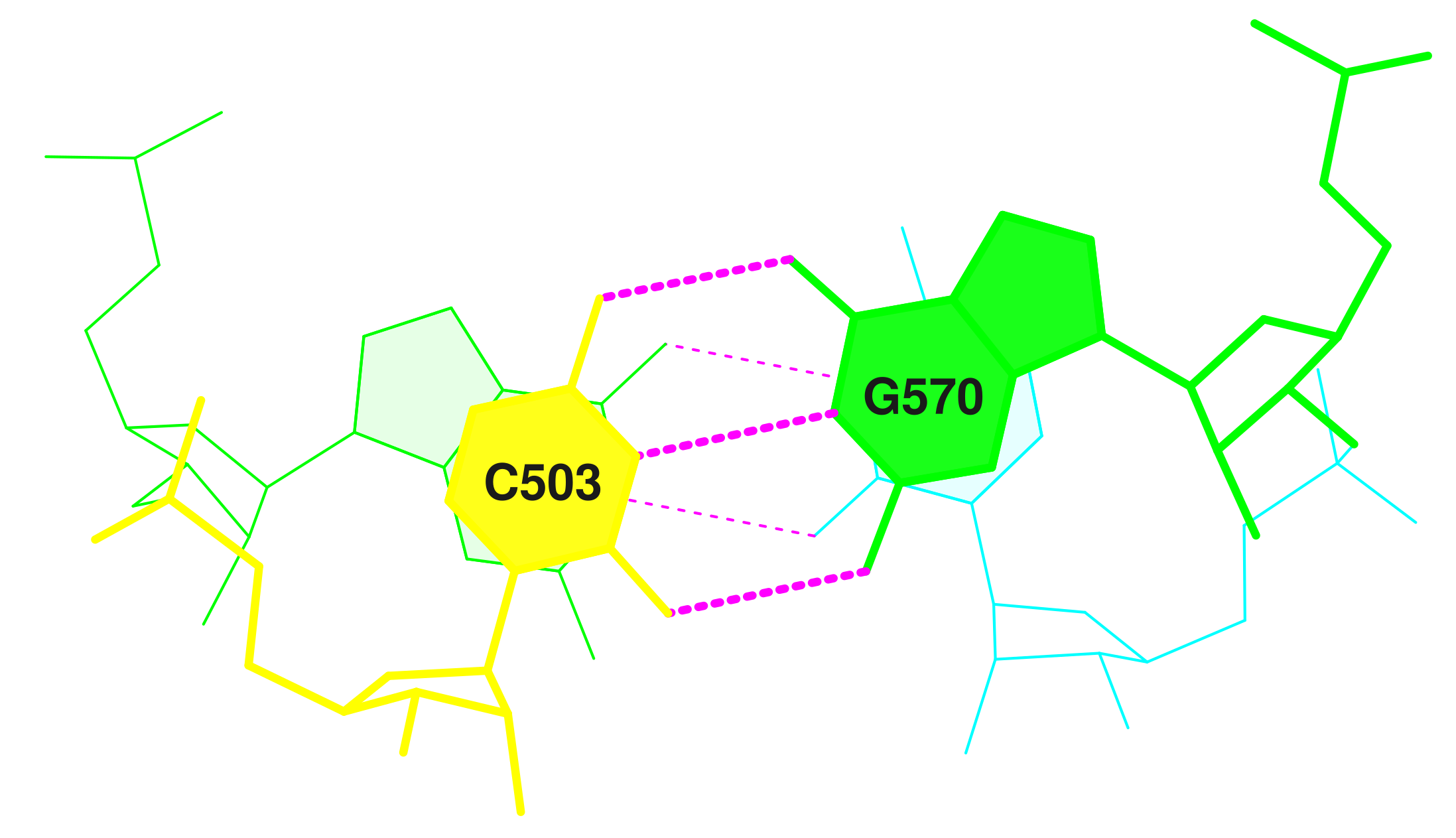 | 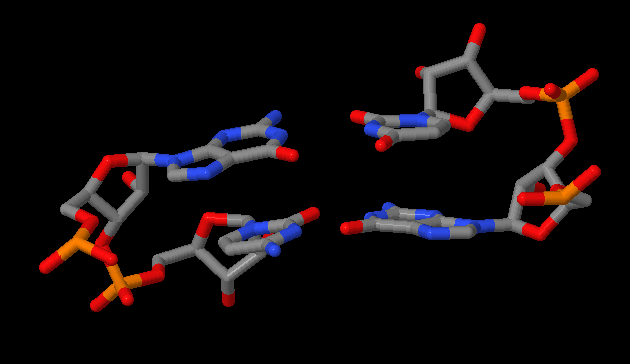 |
| Пары азотистых оснований с наименьшим перекрытием | |
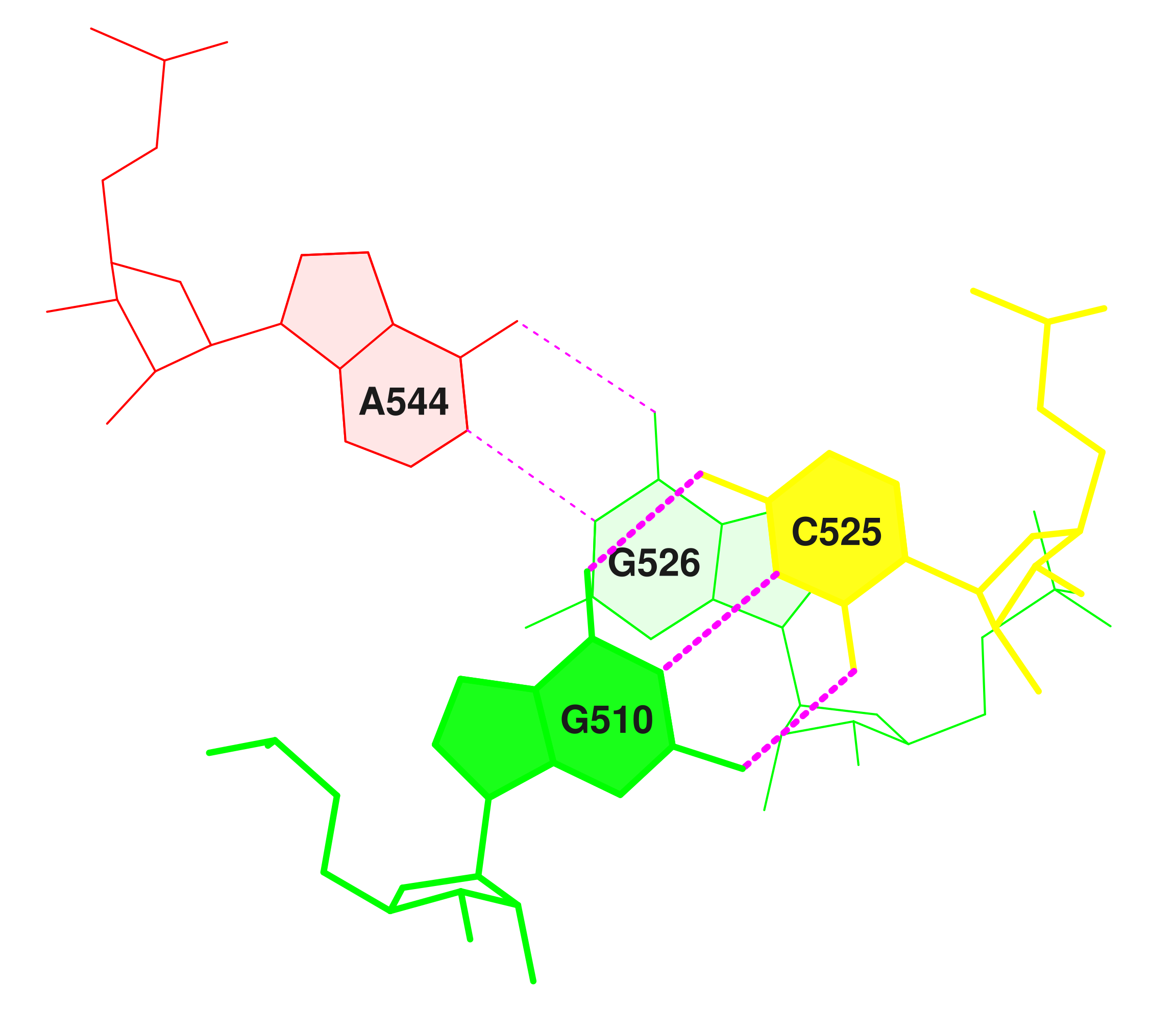 | 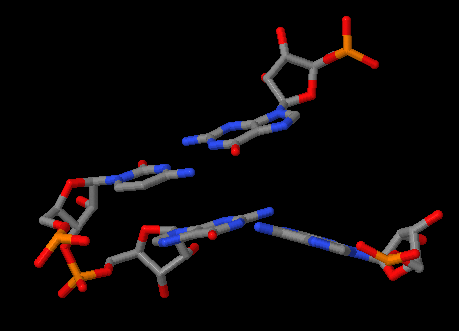 |
© Кучеренко Варвара 2015