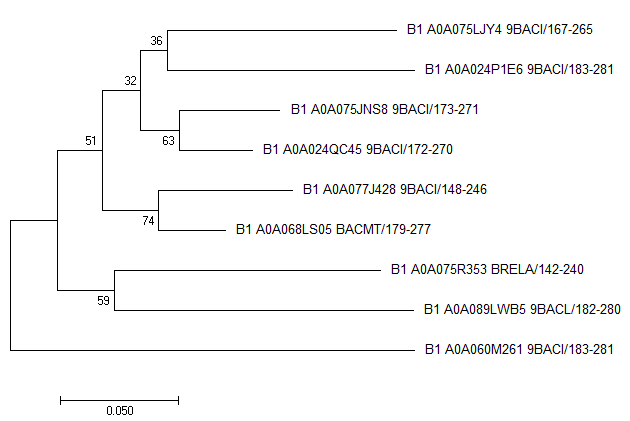
Дерево представителей "хорошего" подсемейства (NJ + Bootstrap, MEGA)
Для построения профиля из выравнивания, полученного в практикуме 11, в отдельный файл были извлечены представители "хорошего" подсемейства (таксон Bacilli; доменна архитектура 1).

Дерево представителей "хорошего" подсемейства (NJ + Bootstrap, MEGA)
Для построения и калибровки профиля применялся пакет HMMER, установленный на kodomo. Использовались следущие команды:
hmm2build profile vibor.fasta
hmm2calibrate profile
Далее в БД UniProt был осуществлен по белкам, включающим домен Hydrolase_2:
hmm2search profile PF07486_full.fasta >> pr12_outputФайл: pr12_output
Далее находки были проанализированы в MS Excel (ссылка на файл). Результаты поиска были перенесены в файл Excel и отсортированы по убыванию нормализованного веса. Всего было получено 2321 находок, среди которых были отмечены представители нашего подсемейства (1 в колонке Profile, если находка принадлежит подсемейству, и 0 в противном случае). Затем были вычислены показатели чувствительности и специфичности.
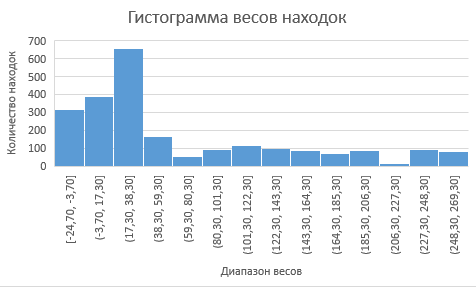 | 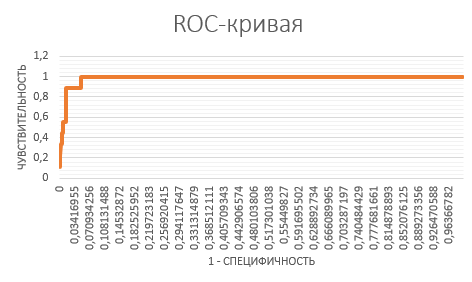 |
| Гистограмма весов находок | ROC-кривая |
На основании построенной ROC-кривой было выбрано пороговое значение E-value. Критерий выбора - максимум разности [Чувствительность - (1-Специфичность)]. Полученный порог E-value - 8,80E-71. При таком пороге наблюдаются достаточно хорошие чувствительноть (1) и специфичность (0.950259516) профиля, то есть профиль вполне пригоден к использованию для выделения подсемейства.
| На самом деле | Принадлежит подсемейству | Не принадлежит подсемейству | Сумма |
| Выше порога по профилю | 9 | 115 | 124 |
| Ниже порога | 0 | 2197 | 2197 |
| Cумма | 9 | 2312 | 2321 |
© Кучеренко Варвара 2015