Гены эукариот
Файл, содержаший контиги сборки X5 был проанализирован с помощью программы infoseq пакека EMBOSS. Из полученного списка контигов был случайным образом выбран unplaced-307 (длина - 84991bp).
Далее потребовалось установить хотя бы примерную таксономию организма, сборку генома которого мы исследуем. Поскольку алгоритм AUGUSTUS будет работать с генами, большинство из которых кодирует белки, имело смысл искать родственные организмы по схожим последовательностям белков, а именно - провести поиск по blastx (ищет трансляции входной нуклеотидной последовательности в 6 рамках по базе белков.) Поиск по нуклеотидным последовательностям мог оказаться неэффективным, учитывая сложную экзонно-интронную структуру генов эукариот.
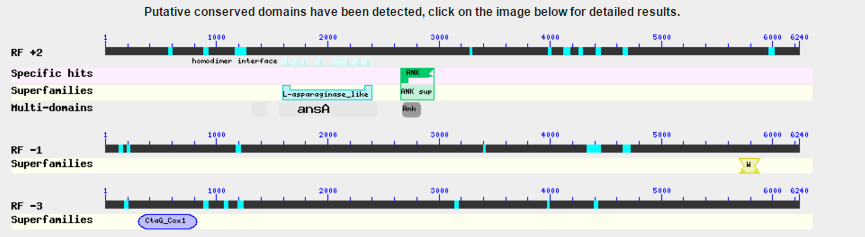
Поскольку длина контига превышает разумные пределы, он был разбит на несколько фрагментов длиной порядка 5-15 kb, которые и были поданы на вход алгоритму blastx. Ниже приведены результаты для одного из них:
 |
 |
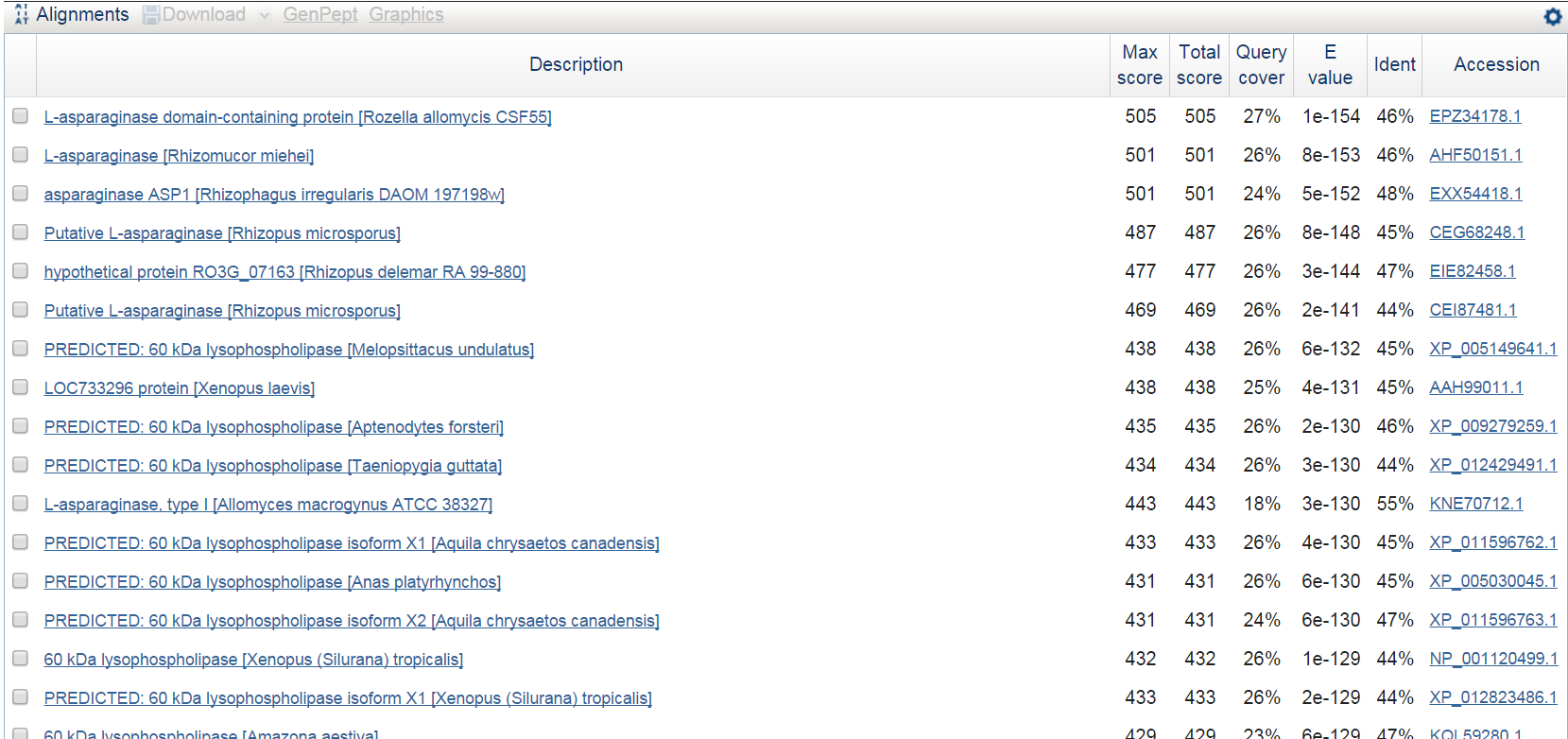 |
Таксономическое положение лучших находок следующее:
- Rozella allomycis: Eukaryota; Fungi; Cryptomycota; Rozella
- Rhizomucor miehei: Eukaryota; Fungi; Fungi incertae sedis; Mucoromycotina; Mucorales; Lichtheimiaceae; Rhizomucor
- Rhizophagus irregularis: Eukaryota; Fungi; Glomeromycota; Glomeromycetes; Glomerales; Glomeraceae; Rhizophagus
- Rhizopus microsporus: Eukaryota; Fungi; Fungi incertae sedis; Mucoromycotina; Mucorales; Mucorineae; Rhizopodaceae; Rhizopus
- Rhizopus delemar: Eukaryota; Fungi; Fungi incertae sedis; Mucoromycotina; Mucorales; Mucorineae; Rhizopodaceae; Rhizopus
Итак, сразу после царства (Fungi) начались расхождения. Анализ еще нескольких фрагментов указал самым перспективным объектом поиска представителей рода Rhizopus, как наиболее часто встречающихся. В списке AUGUSTUS нашелся организм Phizopus oryzae; указав его в поле выбора организма и подав на ввод весь контиг unplaced-307, я получил на выдаче -tar.gz архив с результатами.
Распаковав его командой tar -xzvf predictions.tar.gz, я получил следующий набор файлов:
- augustus.aa - аминокислотные последовательности белков - продуктов предсказанных генов в формате .fasta скачать
- augustus.cdsexons - последовательности предсказанных экзонов скачать
- augustus.codingseq - предсказанные CDS генов в формате .fasta скачать
- augustus.gbrowse - информация о расположении структурных элементов пре-мРНК для каждого предсказанного гена скачать
- augustus.gtf - предсказание генов скачать
- augustus.gff - всё вышеперечисленное в одном файле скачать
Далее требовалось проверить полученное предсказание с помощью Blast. Для проверки были выбраны первые 5 генов: g1.t1, g2.t1, g3.t1, g4.t1, g5.t1. Аминокислотные последовательности были получены из файла augustus.aa. Использовался алгоритм blastp по базе данных SwissProt, описание экзон-интронной структуры на основе вышеперечисленных файлов. Результаты описаны ниже:
- Предсказанный ген g1.t1.
Содержит один экзон и два интрона:
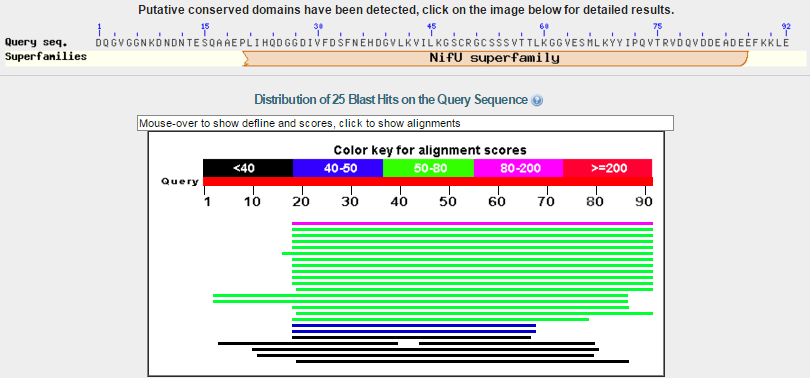
Результаты blastp по продукту предсказанного гену g1.t1.
Обнаруженный консервативный домен позволяет предположить, что экзонно-интронная структура предсказана правильно, и отнести белок к транспортерам железа. Гомологи белка принимают участие в железо-серном кластере, помогающем организмам перенести недостаток железа или окислительный стресс. Таксономия находок разнится: Например, есть белки человека, мыши, дрожжей.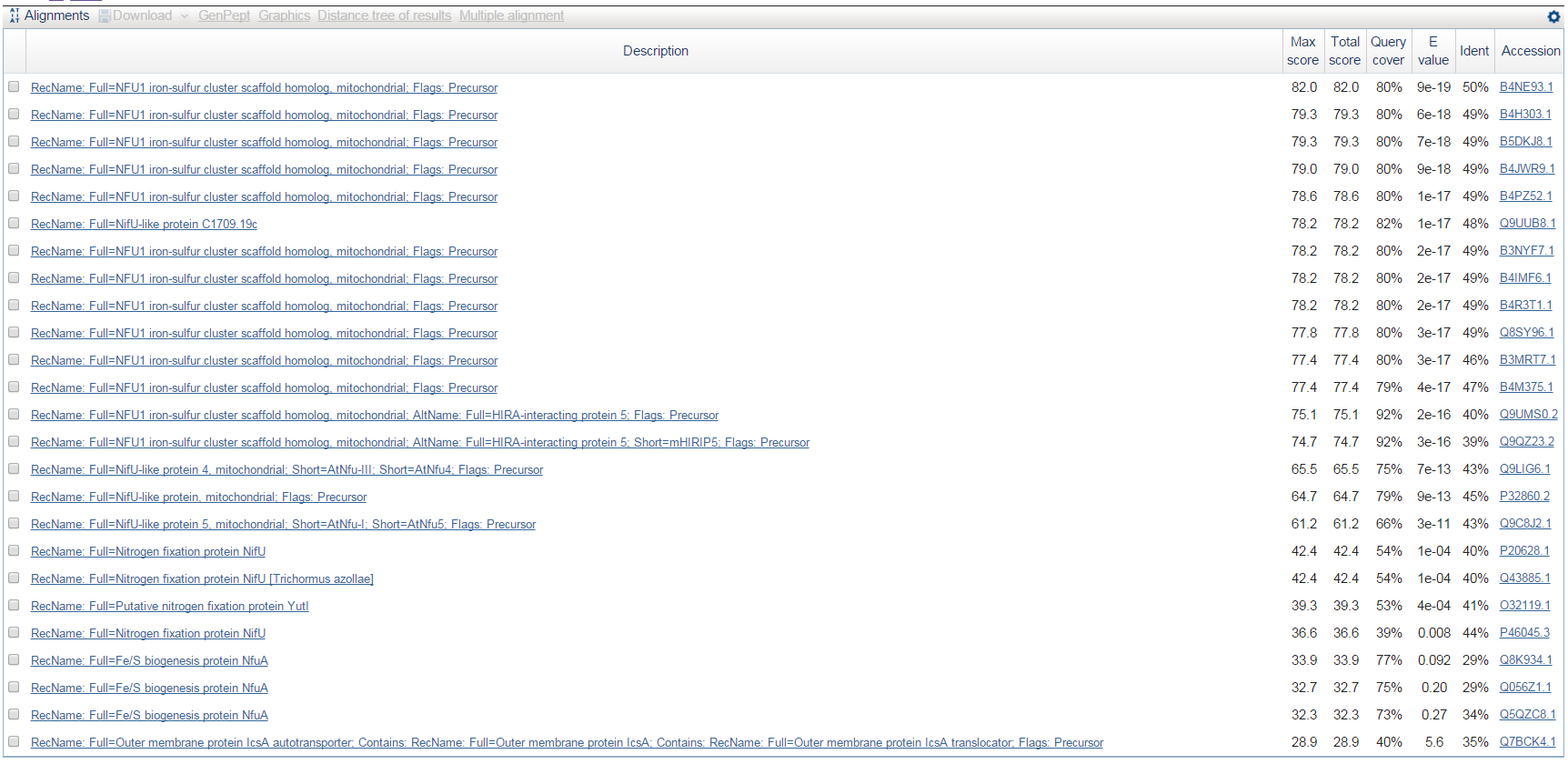
Список находок blastp для продукта g1.t1. - Предсказанный ген g2.t1.
Ген содержит два интрона; поиск по базе SwissProt результатов не дал; такой же результат наблюдался при поиске по его кодирующей последовательности; Это позволяет сделать вывод, что ген был предсказан неправильно. - Предсказанный ген g3.t1.
В отличие от первого случая, нет интронов; обнаружен консервативный домен L-аспарагиназ (ЕС 3.5.1.1) - Ферментов класса гидролаз, катализирующих гидролиз преимущественно L-аспарагина. Применяются в медицине как противоопухолевое цитостатическое средство в терапии некоторых лейкозов, внесены в список наиболее важных лекарств, необходимых в базовой системе здравоохранения: ссылка на drugportal.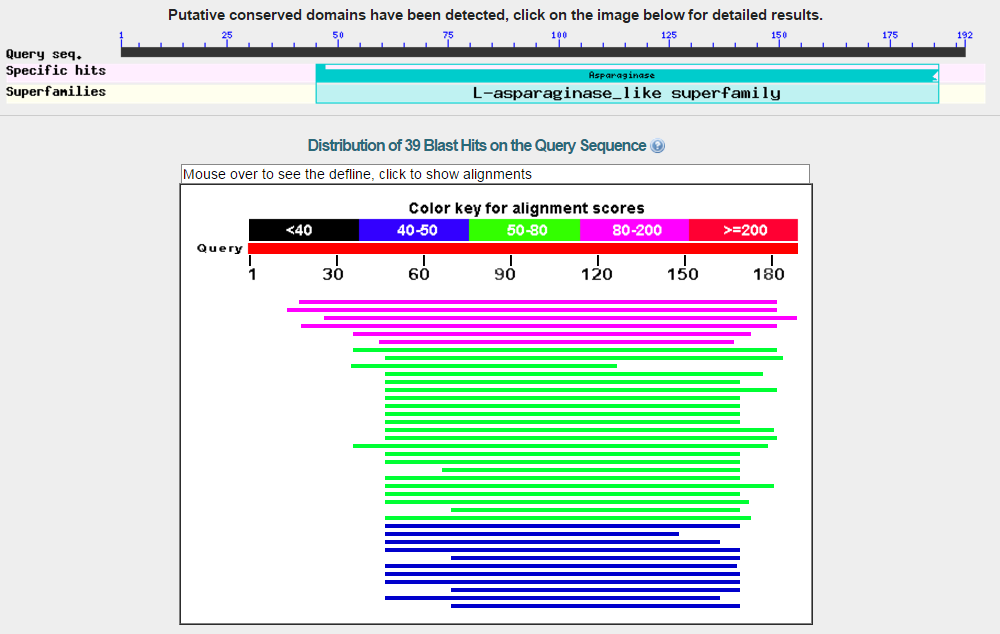
Результаты blastp по продукту предсказанного гена g3.t1. Список находок опять не даёт никаких отсылок к грибам, много находок из млекопитающих: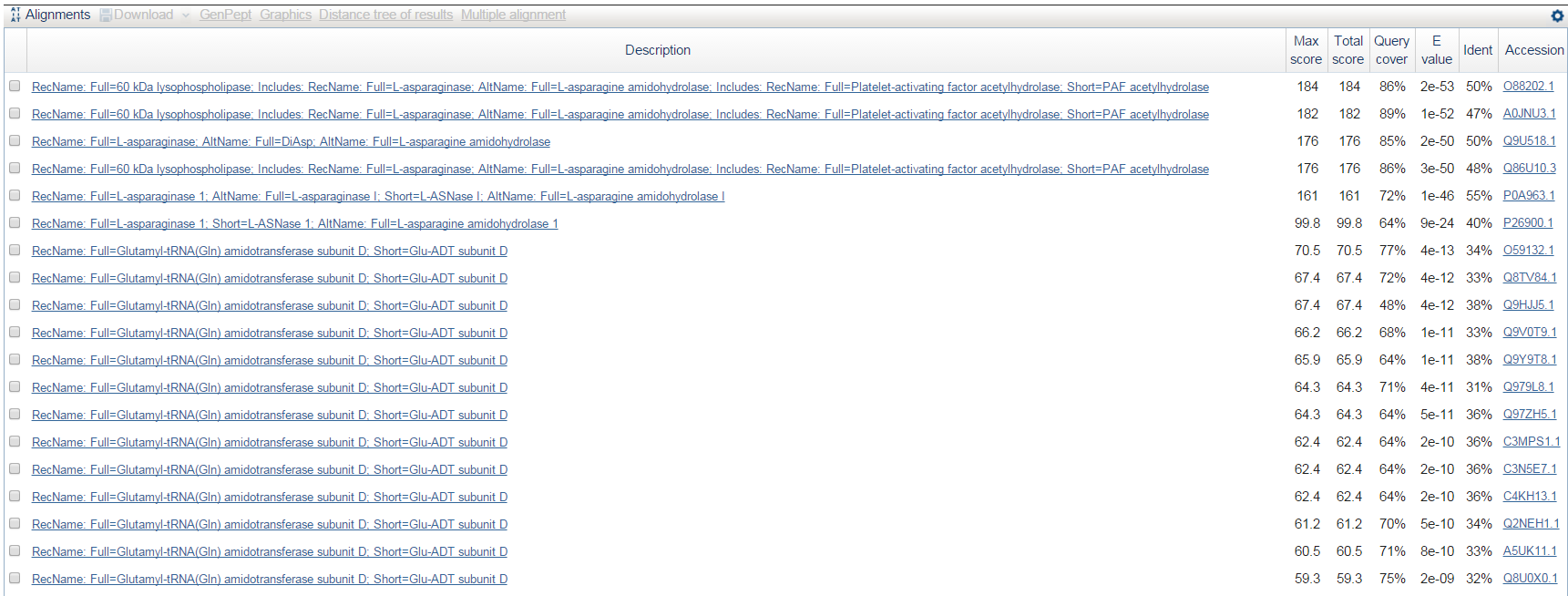
Список находок blastp для продукта g3.t1. - Предсказанный ген g4.t1.
Ген не содержит интронов. В этом случае структура обнаруженных blast'ом доменов была сложнее: помимо однозначно определенного анкиринового повтора было найдено предположительное пристуствие фосфорилирующего агента (6-фосфофрукто-2-киназа, она же фруктозо-2,6-бифосфатаза - фермент, ответственный за регулирование скорости гликолиза и глюконеогенеза, ЕС 2.7.1.105) и, опять же предположительно, белка калиевого канала. Анкирины — семейство внутриклеточных адаптерных белков, участвующих в присоединении других белков к разным участкам клеточной мембраны. Анкириновый повтор - мотив длинной 33 ако, состоящий из двух альфа-спиралей, разделенных петлями. Являются посредниками белок-белковых взаимодействий, одни из наиболее распространенных структурных мотивов в известных белках. Они характерны для белков бактерий, архей и эукариот, но чаще всего встречаются именно у эукариот.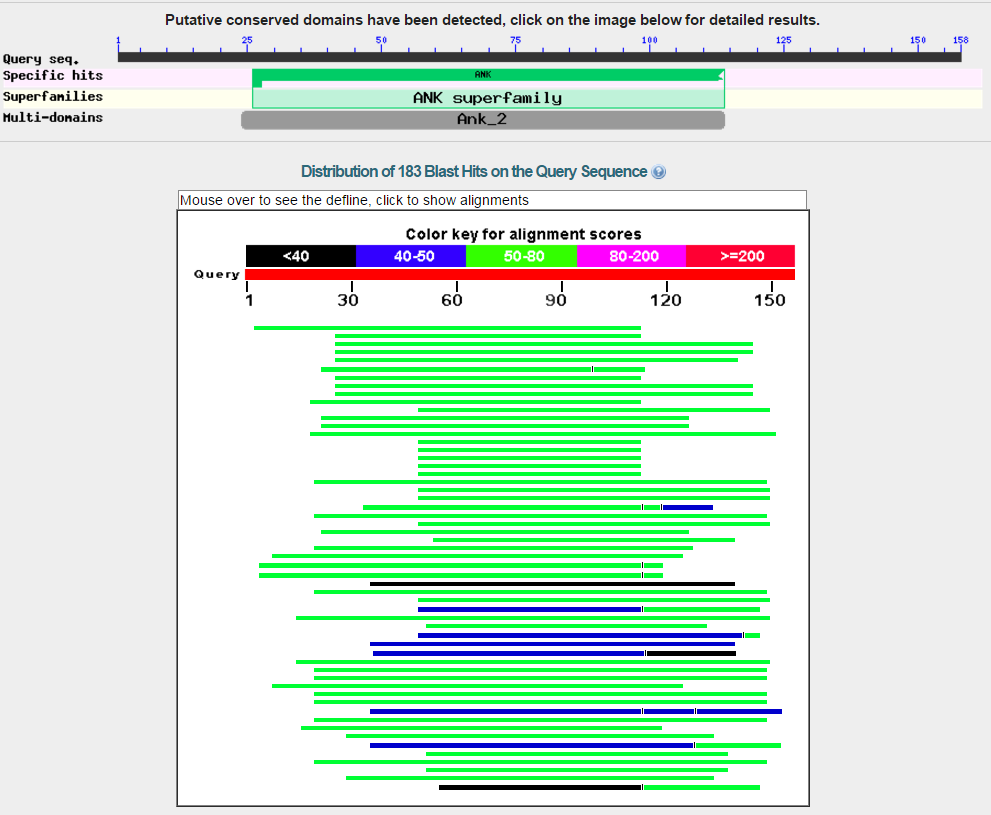
Результаты blastp по продукту предсказанного гена g4.t1. 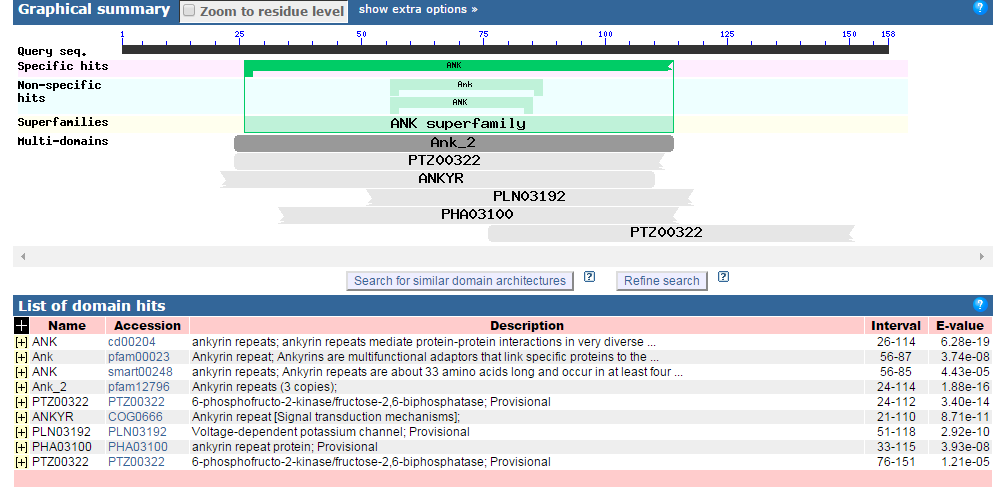
Предложенная Blastp доменная структура Список находок сильно разнился: в списке лучших находок присутствуют уже знакомая нам L-аспарагиназа, калиево-транспортные белки и просто анкириновые кофакторы - и никакого намёка на грибы.
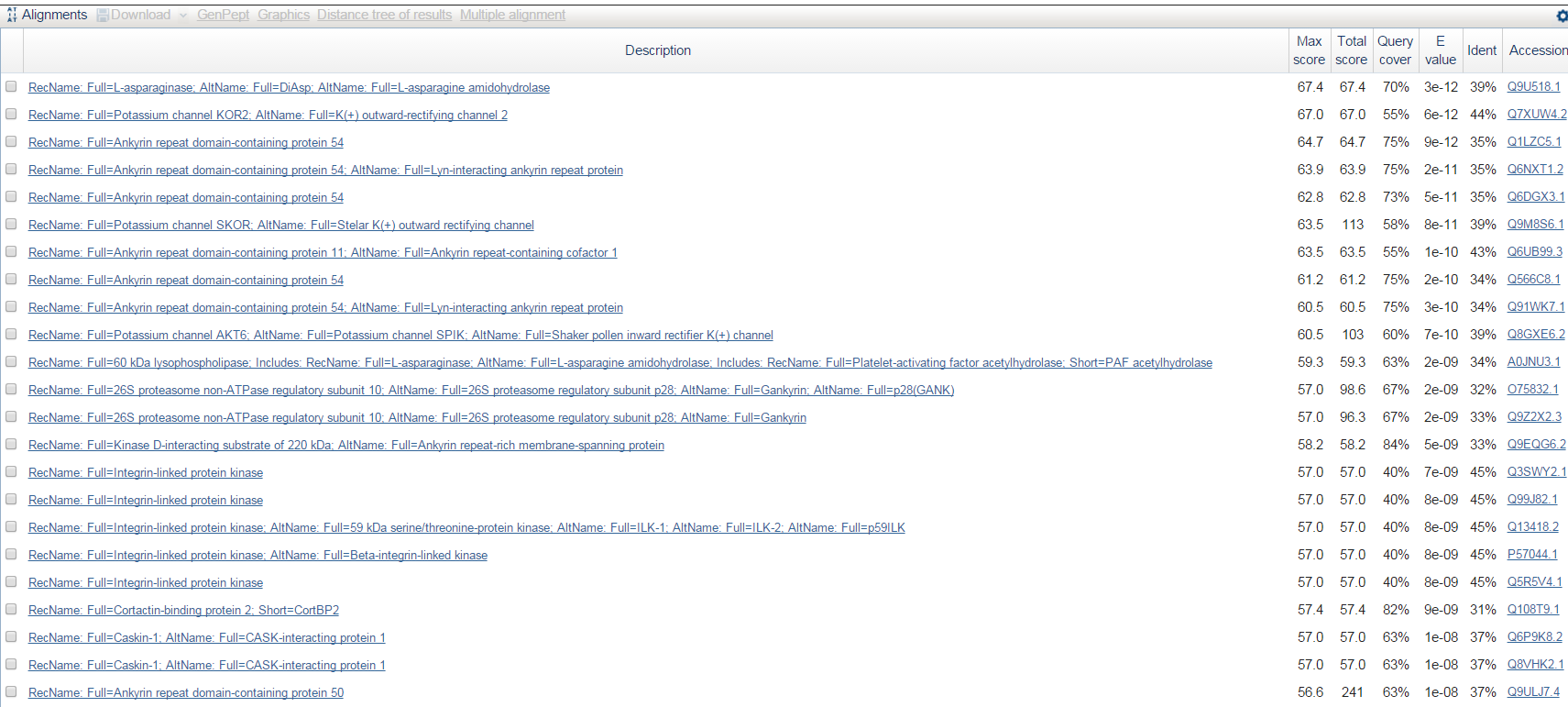
Список находок для продукта g4.t1 Ситуация неоднозначная, поэтому я дополнительно провёл поиск по CDS предсказанного гена. Результат был довольно печальным:
Результат blastn по CDS предсказанного гена g4.t1 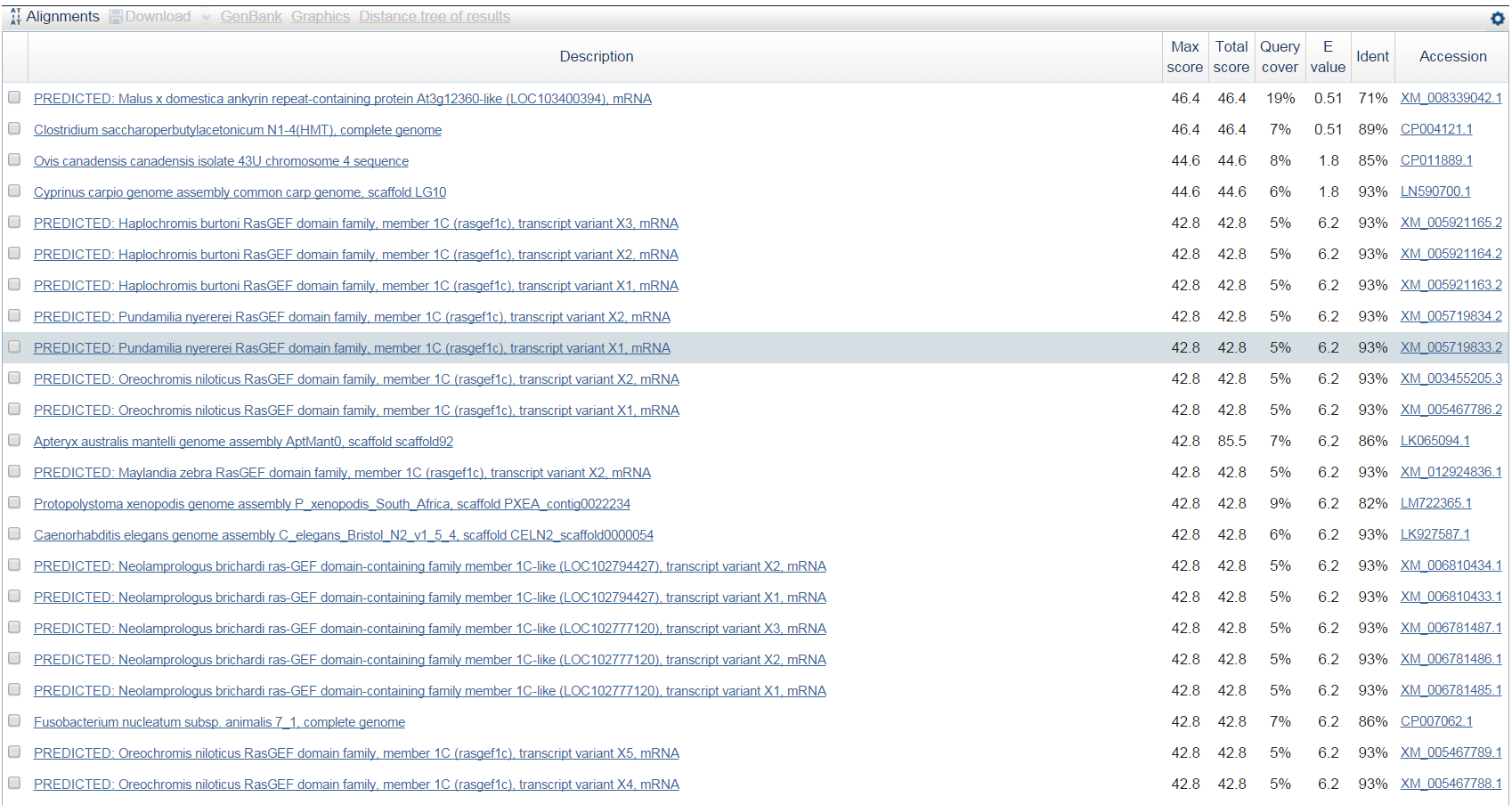
Список находок blastn Обрывки контигов из различных геномов, лучшая из которых - предсказанная последовательность мРНК анкирин-содержащего белка домашней яблони с Query cover 19% - явно не то, что нам нужно. Ген предсказан неправильно.
- Предсказанный ген g5.t1.
Ген содержит 3 интрона. Никаких консервативых доменов обнаружено не было, но среди бедного списка находок большая часть - белок рибосомального биогенезиса NSA1, выделенный у дрожжей Saccharomycetales (АС A5DKC4).
Результаты blastp по продукту предсказанного гена g5.t1. 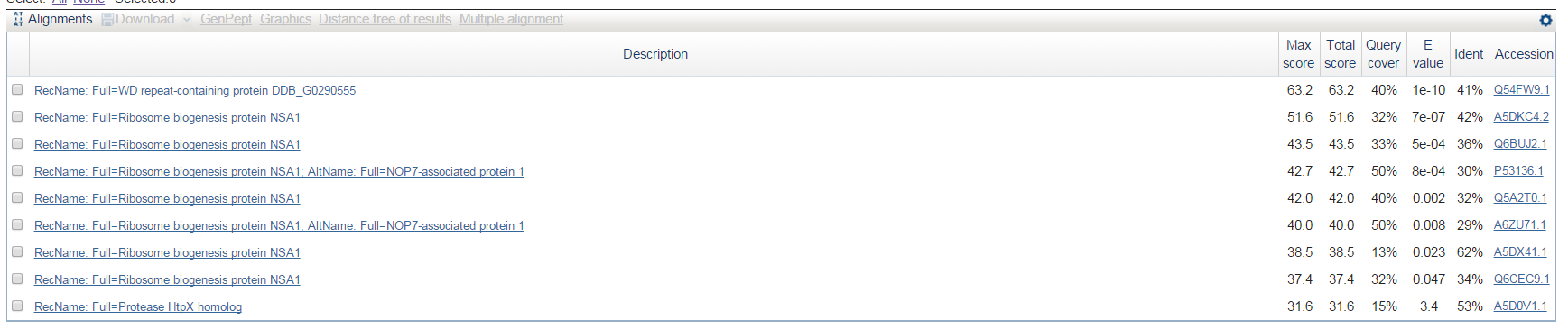
Список находок для продукта g5.t1 Однако, малая степень соответствия находок поданной на вход последовательности заставляет предположить, что ген всё-таки предсказан неправильно.
Таблица с кратким описанием проанализированных генов.
Задание 2. Сравнение аннотации Refseq и AUGUSTUS одного гена человека.
Вкачестве исследуемого гена был выбран ALDH1A1, кодирующий альдегиддегидрогеназу H1A1 человека - фермент, участвующий в
как детоксикации после приёма алкоголя и поддержании прозрачности роговицы, также известный, как Retinal dehydrogenase 1, АС P00352.
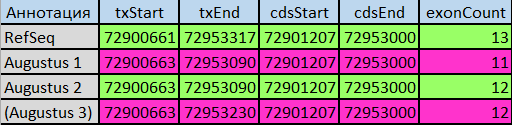
Ген расположен на 9 хромосоме, от 72900663 до 72953230 п.н.,на комплементарной цепи.
Использовался ресурс UCSC Genome Browser. Там была выбрана последняя доступная сборка генома человека (hg38) и, помощью поиска в Genome Browser, был найден интересующий ген ALDH1A1. Далее я оставил только три трека: base position, RefSeq и AUGUSTUS. Ниже приведен скриншот окна браузера с двумя аннотациями гена ALDH1A1.
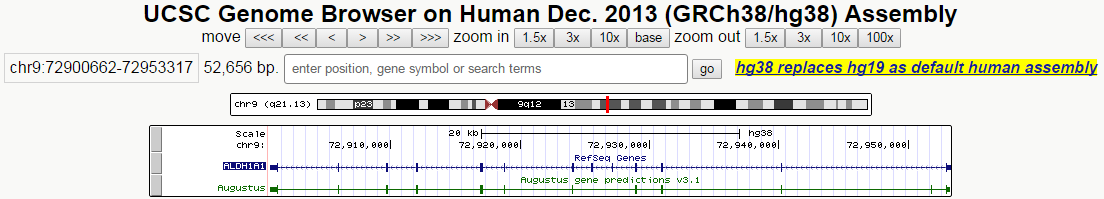
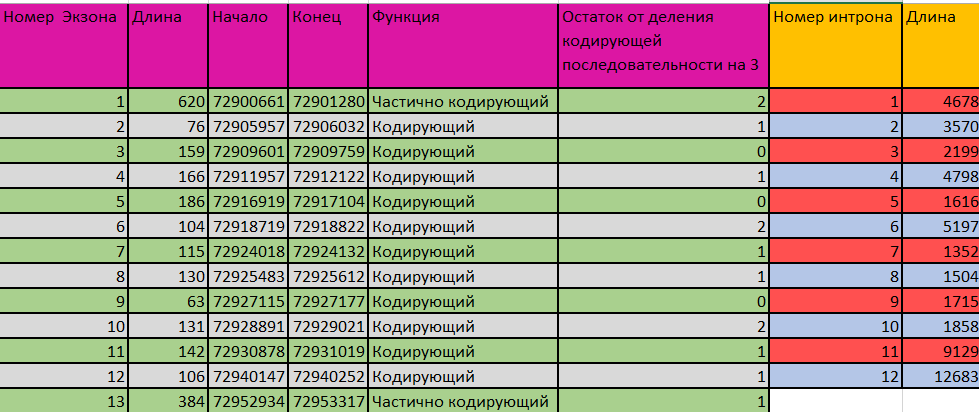
Затем были по отдельности получены предсказания экзон-интронной структуры, выполненные RefSeq и AUGUSTUS, в табличном формате. На листе RefSeq Таблицы представлена информация об экзон-интронной структуре гена, в соответствии с аннотацией RefSeq: всего предсказано 13 экзонов и 12 интронов, координаты кодирующей части (CDS) - от 72901207 до 72953000 п.н. Необходимо иметь в виду, что первый и последний экзон являются частично кодирующими, так как в них присутствуют 5' и 3'-нетранслируемые области, соответственно. Поэтому для вычисления остатка от деления длины кодирующего участка на 3 нужно было использовать полученные координаты CDS, а для остальных - просто использовать форумулу Excel для соответствующих значений длины.
AUGUSTUS делает 3 разных предсказания. В первом (лист Augustus 1 в таблице) предсказано 11 экзонов и 10 интронов соответственно, координаты кодирующей части (CDS) - от 72901207 до 72953000 п.н.
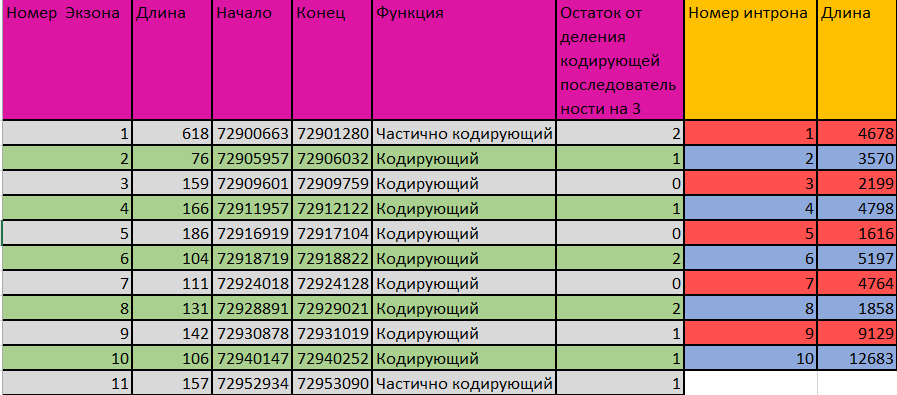
Во втором предсказании AUGUSTUS (лист Augustus 2 в таблице) выделяет 12 экзонов и 11 интронов, координаты кодирующей части (CDS) - от 72901207 до 72953000 п.н.
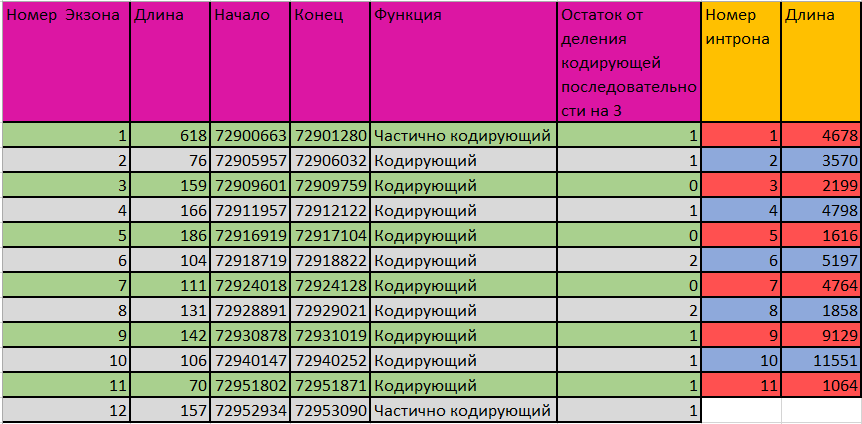
В третьем предсказании AUGUSTUS (лист Augustus 3 в таблице) выделяет снова 12 экзонов и 11 интронов, отличие от второго предсказания - в разных длинах 11 и 12 экзона (и интрона между ними соответственно), координаты кодирующей части (CDS) - от 72901207 до 72953000 п.н.
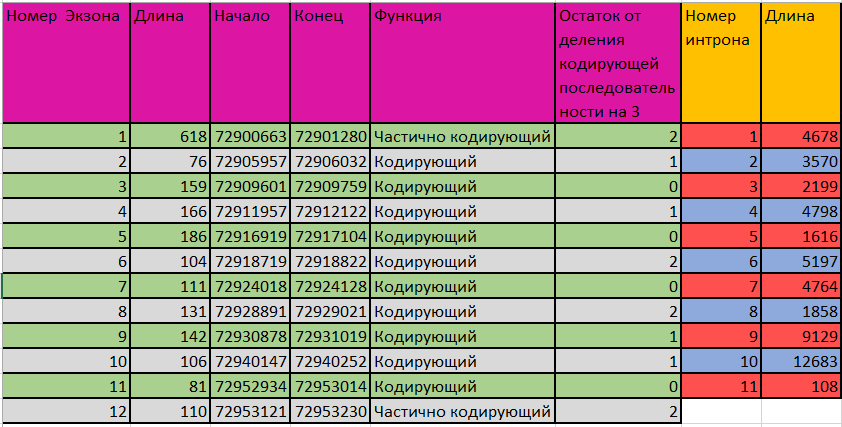
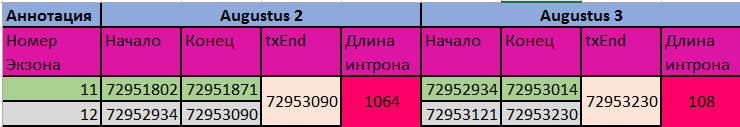
Пришло время сравнить аннотации. Из общего для всех четырех можно отметить то, что и длины кодирующих областей (CDS), и суммы длин всех экзонов кратны трём (иначе к аннотации могли бы возникнуть вопросы) , и что суммы длин интронов превосходят длины экзонов как минимум в 10 раз, что характерно для эукариот.
Все аннотации дали разное количество экзонов и разные координаты гена и кодирующей последовательности. При этом 2 и 3 аннотация AUGUSTUS толичаются только по координатам txEnd и (как было замечено ранее) координатами и длиной двух последних экзонов и последнего интрона между ними. При этом, интрон с более правдоподобной длиной находится во второй аннотации AUGUSTUS (1064, почти в 10 раз длинее интрона из аннотации 3). Поэтому в дальнейшем будем считать более правдоподобной именно аннотацию AUGUSTUS 2. Сравнение аннотаций приведено в таблице на листе 2v3:
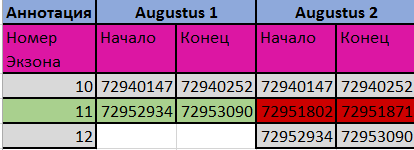
Далее приведем сравнение первой и второй аннотации AUGUSTUS. Их кодирующие последовательности и CDS имеют одинаковые координаты. При этом можно заметить, что в первой аннотации небольшой экзон длины 70 просто отсутствует. Сравнение приведено в таблице на листе 1v2:
Теперь сравним эти две аннотации с Refseq. Для начала посмотрим на различия в длинах областей txStart, txEnd, кодирующих областей и числа экзонов (см. таблицу, лист 1v2vR):
В аннотации RefSeq довольно сильно отличаются координаты начала и конца транскрипции от соответствующих координат во всех аннотациях Augustus, причем различие начинается еще с певого экзона. Тем не менее, если сравнить координаты последних экзонов трёх исследуемых аннотаций, можно заметить, что экзое №11 из аннотации Augustus не имеет аналогов в двух других, на основании чего наиболее правдоподобной можно считать аннотацию Augustus 1, которая отличается от аннотации RefSeq незначительными отличиями в координатах экзонов и пропущенным экзоном № 9. Сравнение представлено в таблице на том же листе:
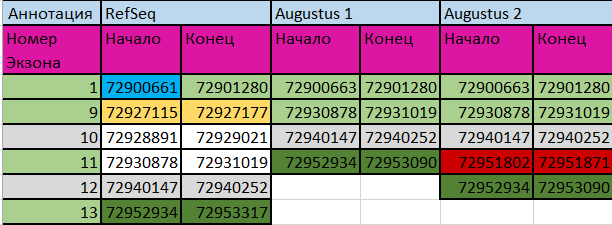
Голубым выделено отличие начала первого экзона аннотации RefSeq от аннотаций Augustus, бордовым - несовпадающий с прочими аннотациями экзон №11, тёмно-зеленым - почти одинаковые последние экзоны, песочным - пропущенный экзон №9 из аннотации RefSeq.
В целом, Augustus неплохо справляется с предсказанием генов эукариотических организмов, но в процессе аннотации генов могут возникать неоднозначные моменты, каждый из которых приходится досконально проверять (как мы только что убедились).