Ресеквенирование. Поиск полиморфизмов у человека.
Для начала я создал свою директорию crescent8547 по адресу /nfs/srv/databases/ngs/ на сервере kodomo и скопировал в нее файл с ридами chr16.fastq и файл с последовательностью хромосомы chr16.fasta. Затем с помощью команды fastqc chr16.fastq я произвёл контроль качества чтений и получил zip-архив. В архиве находится файл fastqc_report.html, в котором содержится отчёт о проделанной программой работе. В том числе на это странице находится и представленная на Рис.1 таблица с оценкой качества чтений:
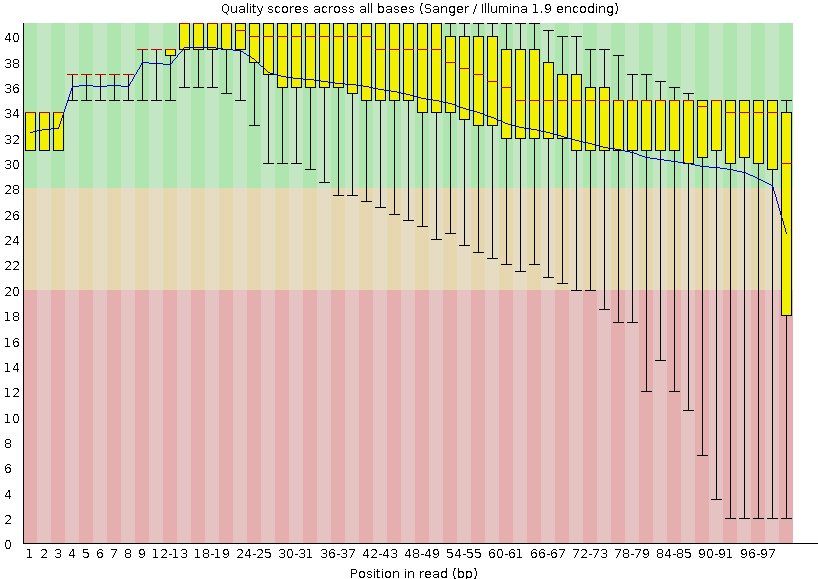
Как видно из рисунка, качество чтения высокое (score > 20) почти на всём протяжении последовательности. Ридов в последовательности 3965.
Далее требовалось провести очистку чтений от участков плохого качества с помощью программы Trimmomatic, установленной на Kodomo. Для выполнения этого задания я применил следующие три команды:
- java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr16.fastq trim1.fastq LEADING:20 - для удаления участков со скором меньше 20 из начала чтений;
- java -jar /usr/share/java/trimmomatic.jar SE -phred33 trim1.fastq trim2.fastq TRAILING:20 - для удаления участков со скором меньше 20 из конца чтений;
- java -jar /usr/share/java/trimmomatic.jar SE -phred33 trim2.fastq trim3.fastq MINLEN:50 - для удаления чтений длиной менее 50.
Далее к полученному файлу trim3.fastq была применена команда FastQC: результат.
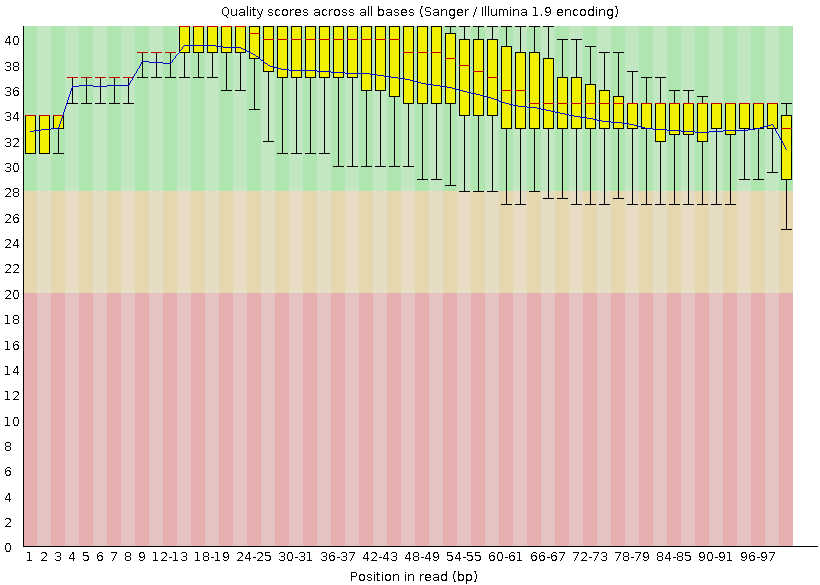
Теперь ридов 3796 - исчезли прочтения низкого качества, "заходящие" в область с качеством меньше 20.
Часть II. Картирование чтений.
Очищенные чтения были картированы с помощью программы BWA. Сначала рeференсная последовательность была проиндексирована с помощью команды bwa index:
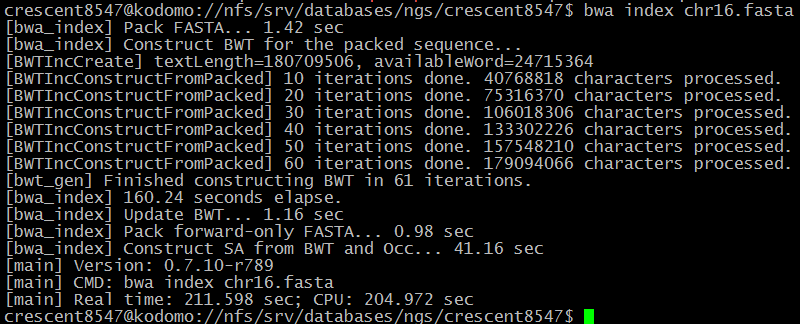
Затем было произведено выравнивание референса с прочтениями:
- bwa mem chr16.fasta trim3.fastq > align.sam. Полученный файл в формате .sam можно скачать здесь.
Проанализируем полученное выравнивание:
- samtools view -b align.sam -o aln.bam - Переведём его в бинарный формат;
- samtools sort aln.bam out.predix - отсортируем по координате в референсе начала чтения;
- Наконец, проиндексируем полученный файл: samtools index out.predix.bam, после чего мы, наконец, сможем узнать, сколько ридов было откартировано:
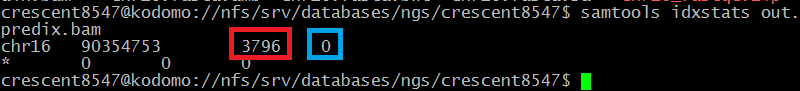
Как и ожидалось, на хромосому картирвались все риды без исключения.
Часть III. анализ SNP.
займёмся поиском SNP и инделей в выданной хромосоме. Для этого применим следующие команды:
- samtools mpileup -uf chr16.fasta out.predix.bam > snp.bcf - создадим файл с полиморфизмами в формате .bcf
- bcftools call -cv snp.bcf > snp.vcf - получаем файл со cписком полиморфизмов в читаемом формате:
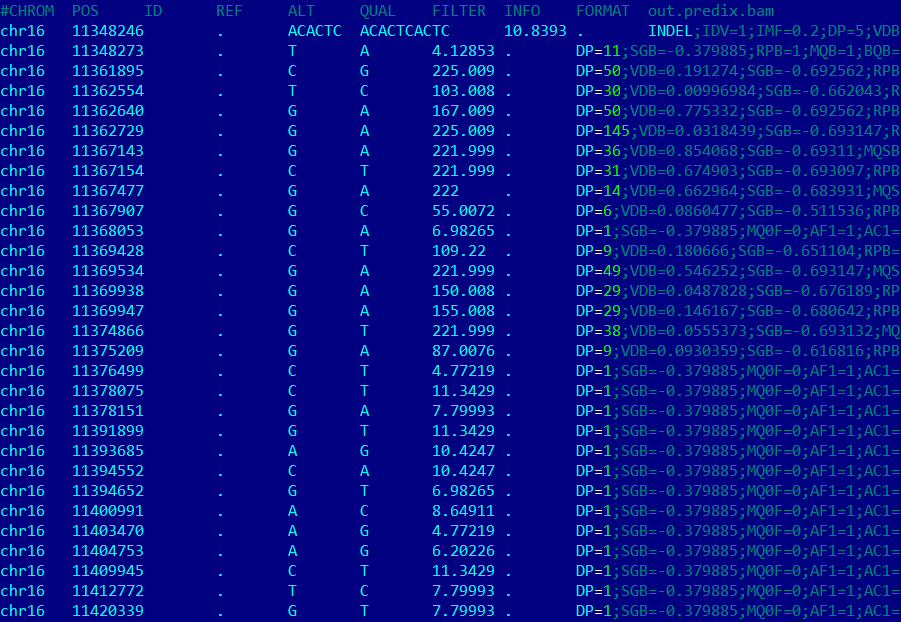
Из списка, содержащего 63 snp и 2 инделя, были выбраны три полиморфизма:
- Полиморфизм с координатами 11348246 - индель (АСАСТСАСТС вместо АСАСТС), качество чтения 10.8393, глубина 11;
- Полиморфизм с координатами 11348273 - замена Т на А, качество чтения 4.12853, глубина 50;
- Полиморфизм с координатами 11361895 - замена C на G, качество чтения 225.009, глубина 30.
Далее требовалось аннотировать все SNP. Для этого я удалил из файла аннотации двух инделей, которые в дальнейшем не будут нас интересовать. После этого я обратился к программе annovar:
- perl /nfs/srv/databases/annovar/convert2annovar.pl -format vcf4 snp2.vcf > converted.avinput - перевод файла snp2.vcf в формат, с которым может работать annovar.
- perl /nfs/srv/databases/annovar/annotate_variation.pl -out RefGeneAnnotation -build hg19 converted.avinput //nfs/srv/databases/annovar/humandb/ - аннотация содержимого файла convert.avinput по базе данных RefGene (директория humandb) с помощью скрипта annotate_variation.pl (в директории annovar). Выдача представлена в двух формах: в файле variant_function собраны все SNP, а в exonic_variant_function - только SNP из экзонов.
- perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out SNP138Annotate -build hg19 -dbtype snp138 converted.avinput /nfs/srv/databases/annovar/humandb/ - аннотирует содержимое файла converted.avinput по базе данных snp138 из директории humandb с помощью скрипта annotate_variation.pl, находящемся в директории annovar. Проанализировав выдачу, можно узнать, что 57 snp имеют rs.
- perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype 1000g2014oct_eur -buildver hg19 -out 1000gAnnotate converted.avinput /nfs/srv/databases/annovar/humandb/ - аннотирует содержимое файла converted.avinput по базе данных 1000 genomes с помощью скрипта annotate_variation.pl.
- perl /nfs/srv/databases/annovar/annotate_variation.pl -regionanno -build hg19 -out gwasAnnotate -dbtype gwasCatalog converted.avinput /nfs/srv/databases/annovar/humandb/ - аннотирует содержимое файла converted.avinput по базе данных Gwas с помощью скрипта annotate_variation.pl.
- perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 converted.avinput -outfile snp_clinvar /nfs/srv/databases/annovar/humandb/ - аннотирует содержимое файла converted.avinput по базе данных Clinvar с помощью скрипта annotate_variation.pl.
- 1000 genomes - Общая информация по полиморфизмам (например, частота встречаемости);
- RefGene - Ген, в котором находится конкретный полиморфизм;
- snp138 - Собственно, база snp;
- Gwas - Связь между полиморфизмом и заболеванием;
- Clinvar - Возможно, связь между полиморфизмом и фенотипом (в данном случае совпадений не было найдено).
После аннотации в этих базах можно получить следующую информацию:
Данная информация была сведена в таблицу, которая содержит общую информацию о snp и информацию, которую можно получить о каждом из них из упомянутых выше баз данных (лист "Полиморфизмы"). После анализа данных можно прийти к выводу, что пациент страдает ожирением (частота 0.68), имеет предрасположенность к метаболическому синдрому (частота 0.14) и, вероятно, к болезни Паркинсона (частота 0.37).
Не лишним будет подметить, что предрасположенность к ожирению встречается в популяции довольно часто, а к метаболическому синдрому - почти в 5 раз меньше. Совокупность распространенного и редко встречающегося полиморфизма, связанных с одним заболеванием, позволяет с большй долей уверенности говорить о наличии у пациента проблем со здоровьем, связанных с избыточным весом. Скорее всего, в других хромосомах найдутся схожие полиморфизмы, указывающие на проблемы в данной области. Для определения предрасположенности к болезни Паркинсона у нас слишком мало данных.
Также, в таблице на листе "Замена АК" были приведены данные о заменах аминокислот, имевшиеся в базе данных RefGene.
В целом, метод аннотации snp выглядит довольно перспективным в сфере ранней диагностики заболеваний и упреждающих методов лечения, подбора лекарств и их дозировок.