A- и В- формы ДНК. Структура РНК
Задание 1. С помощью fiber были созданы pdb-файлы с А- и В- структурами ДНК, состоящей из 5 повторов последовательности GATC, и Z-структурой, которая представляет собой 10 раз повторённый участок gc, т.к. команда fiber пакета 3DNA не предполагает введения последовательности, а лишь принимает на ввод количество gc-повторов).: gatc-a.pdb,gatc-b.pdb и gatc-z.pdb.
Задание 2. Задание полностью представлено в jmol-апплете ниже (скрипты Task_2_ex[1-3]).
- Упражнение 1. Для выполнения упражнения был использован файл gatc-a.pdb, созданный при выполнении первого задания. Запускаемый кнопкой Task_2_ex_1 скрипт последовательно показывает: сахарофосфатный остов ДНК; все нуклеотиды; все нуклеотиды содержащие основание аденин; атом N7 во всех гуанинах.
- Упражнение 2. С сайта PDB были скачаны файлы 1I9V и 1MDM. Просмотреть их можно с помощью кнопки Task_2_ex_2.
- Упражнение 3. В полученных структурах разрывов нет (см. скрипт Task_2_ex_3). Координаты атомов ДНК и РНК сохранены в файлах newrna.pdb и newdna.pdb.
| После того, как структура загрузится, надо выбрать сценарий:
1. Запустить скрипт: 2. Продолжить исполнение скрипта:
Text of the script for 1st task |
Задание 3.
- Упражнение 1. Выбранное азотистое основание (Тимин, [T]35:B) показан желтым цветом при выполнении скрипта Task_3_ex_1. Проводилось сравнение
тимидиловых нуклеотидов из А- и В-формы, получеенное с помощью ChemSketch (в Z-форме отсутствуют тимидиловые нуклеотиды). Красным цветом выделены атомы, смотрящие в сторону большой бороздки,
синим - в сторону малой.
- В А-форме в сторону большой бороздки обращены атомы T7.C6, T7.O4, T7.C5M, T7.C5, в сторону малой - T7.C2, T7.O2.
- В В-форме в сторону малой бороздки обращены атомы T7.C6, T7.O4, T7.C5M, , T7.C5, T7.C4, в сторону большой - T7.C2, T7.O2.
На рисунке 1 показаны атомы, направленные в сторону большой и малой бороздок:
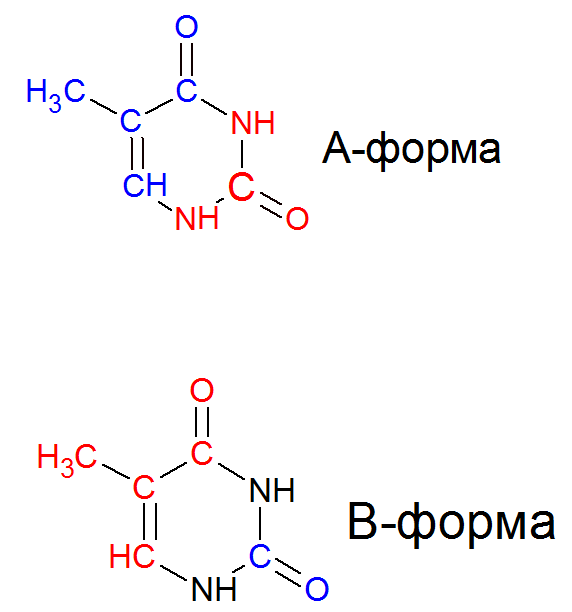
Рисунок 1. - Упражнение 2. Таблица 1: сравнение структур А-, В- и Z-форм ДНК.
Основные спиральные характеристики форм ДНК
A-форма
B-форма
Z-форма
Тип спирали (правая или левая) Правая
Правая
Левая
Шаг спирали (A) 28,03
33,75
43,5
Число оснований на виток 11
10
12
Ширина большой бороздки (A) 16.83 ([G]25:B.P - [A]18:A.P)
17,21 ([C]4:A.P - [A]34:B.P)
16,08 18.03 ([C]14:B.P - [C]4:A.P)
Ширина малой бороздки (A) 7,98 ([T]3:A.P - [C]32:B.P)
11,69 11.69 ([A]26:B.P - [T]19:A.P)
7,2 7.2 ([G]5:A.P - [G]19:B.P)
- Упражнение 3. Требовалось измерить и сравнить торсионные углы тимидилового нуклеотида А- и В-форм ДНК. Полученные при измерении данные представлены в Таблице 2.
Форма/Угол alpha beta gamma delta epsilon zeta chi A -51.7 174.8 41.7 79.0 -147.8 -75 -157 B -29.9 136.3 31.1 143.4 -140.8 -160.5 -98
Задание 4. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
- С помощью пакета 3DNA были измерены значения торсионных углов структурах А-, В- и Z-форм ДНК (файлы gatc-b.pdb, gatc-а.pdb, gatc-z.pdb, созданные при
выполнении задания 1) и в выданной структуре тРНК (в таблице приведены средние значения углов); результаты представлены в Таблице 3.
Форма/Угол alpha beta gamma delta epsilon zeta chi A -51.7 174.8 41.7 79.0 -147.8 -75 -157 B -29.9 136.3 31.1 143.4 -140.8 -160.5 -98.0 Z (цитозин) -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 Z (гуанин) 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 1I9V (тРНК-структура) -38 102,69 65,95 85,30 -108,91 -55,71 -148,66
Как можно заметить, для Z-формы торсионные углы у гуанина и цитозина сильно различаются; Выданная структура тРНК больше всего похожа на А-форму ДНК. - Далее были измеряны торсионные углы структур ДНК и тРНК в полученных ранее файлах. После перевода файлов в старый формат программой remediator к ним были применены команды find_pair и analyze. Результаты - в Таблице 4. Представлена информация по каждому изнуклеотидов кроме крайних, для которых, очевидно, невозможно определить два угла из восьми. Самый деформированный нуклеотид в РНК по углу χ - 12-й цитозин второй цепи, в ДНК - 17-й цитозин первой цепи по углу ζ.
- Определение структуры водородных связей.
Рассмотрим водородные связи в структуре тРНК и на основе этого попробуем определить особенности вторичной структуры. Для этого используем файл
newrna_old.out, полученный при анализе файла с РНК-структурой:
Strand I Strand II 1 (0.010) A:...1_:[..G]G-----C[..C]:..72_:A (0.013) 2 (0.011) A:...2_:[..C]C-----G[..G]:..71_:A (0.010) 3 (0.018) A:...3_:[..G]G-----C[..C]:..70_:A (0.008) 4 (0.014) A:...4_:[..G]G-*---U[..U]:..69_:A (0.006) 5 (0.018) A:...5_:[..A]A-----U[..U]:..68_:A (0.008) 6 (0.009) A:...6_:[..U]U-----A[..A]:..67_:A (0.006) 7 (0.013) A:...7_:[..U]Ux----A[..A]:..66_:A (0.017)Акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 66-72.8 (0.017) A:..49_:[..C]C-----G[..G]:..65_:A (0.021) 9 (0.009) A:..50_:[..U]U-----A[..A]:..64_:A (0.012) 10 (0.013) A:..51_:[..G]G-----C[..C]:..63_:A (0.010) 11 (0.007) A:..52_:[..U]U-----A[..A]:..62_:A (0.023)
T-стебель состоит из участка 49-52 и комплементарного ему 62-65.16 (0.014) A:..40_:[..C]C-----G[..G]:..30_:A (0.015) 17 (0.008) A:..41_:[..U]U-----A[..A]:..29_:A (0.014) 18 (0.008) A:..42_:[..G]G-----C[..C]:..28_:A (0.009) 19 (0.011) A:..43_:[..G]G-----C[..C]:..27_:A (0.014)
Антикодоновый стебель состоит из участка 40-43 и комплементарного ему 27-30.21 (0.005) A:..10_:[..G]G-----C[..C]:..25_:A (0.004) 22 (0.021) A:..11_:[..C]C-----G[..G]:..24_:A (0.014) 23 (0.019) A:..12_:[..U]U-----A[..A]:..23_:A (0.013) 24 (0.011) A:..13_:[..C]C----xG[..G]:..22_:A (0.020)
D-стебель состоит из участка 10-13 и комплементарного ему 22-25. Всего 7 неканонических пар. Пример неканонической пары:A:...4_:[..G]G-*---U[..U]:..69_:A,
т.е. G4-U69.
Найдем дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру. Для этого рассмотрим комплементарные пары, не имеющие отношения к стеблям.19_:[..G]G-----C[..C]:..56
То есть сюда относится пара G19-G56. - Возможные стекинг - взаимодействия.
Стэкинг взаимодействия - взаимодействия между основаниями, уложенными одно над другим. В них участвуют силы Ван-дер-Ваальса и диполь-дипольные взаимодействия.
Используем уже знакомы й нам файл newrna_old.out с характеристикой структуры тРНК. В нем была найдена информация о перекрывании пар нуклеотидов:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GC/GC 3.98( 1.77) 0.00( 0.00) 0.00( 0.00) 6.04( 2.90) 10.02( 4.67) 2 CG/CG 0.76( 0.02) 0.00( 0.00) 2.50( 0.28) 0.54( 0.03) 3.80( 0.33) 3 GG/UC 2.14( 0.70) 0.00( 0.00) 0.33( 0.00) 0.08( 0.00) 2.55( 0.70) 4 GA/UU 4.08( 2.64) 0.00( 0.00) 0.00( 0.00) 1.13( 0.31) 5.22( 2.95) 5 AU/AU 5.44( 3.94) 0.00( 0.00) 0.00( 0.00) 3.86( 2.50) 9.31( 6.43) 6 UU/AA 0.52( 0.00) 0.00( 0.00) 0.00( 0.00) 2.22( 2.14) 2.74( 2.14) 7 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.93( 1.39) 2.93( 1.39) 8 CU/AG 0.00( 0.00) 0.00( 0.00) 0.10( 0.00) 4.44( 3.29) 4.55( 3.29) 9 UG/CA 0.00( 0.00) 0.00( 0.00) 2.58( 1.52) 0.28( 0.00) 2.86( 1.52) 10 GU/AC 7.09( 4.53) 0.00( 0.00) 0.00( 0.00) 3.04( 1.53) 10.13( 6.06) 11 UG/CA 0.01( 0.00) 0.00( 0.00) 4.34( 2.04) 0.00( 0.00) 4.36( 2.04) 12 Gu/AC 8.29( 3.00) 0.00( 0.00) 0.00( 0.00) 3.56( 0.99) 11.85( 3.99)*** 13 uA/UA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 14 AA/CU 0.99( 0.00) 0.00( 0.00) 0.00( 0.00) 4.33( 1.92) 5.32( 1.92) 15 AC/GC 0.16( 0.00) 0.00( 0.00) 0.00( 0.00) 7.55( 3.90) 7.70( 3.90) 16 CU/AG 0.00( 0.00) 0.00( 0.00) 1.07( 0.00) 1.27( 1.27) 2.34( 1.27) 17 UG/CA 0.14( 0.00) 0.00( 0.00) 2.82( 1.70) 0.00( 0.00) 2.96( 1.70) 18 GG/CC 3.49( 2.01) 0.00( 0.00) 0.45( 0.00) 0.00( 0.00) 3.94( 2.01) 19 GA/GC 2.00( 0.58) 0.00( 0.00) 0.00( 0.00) 3.53( 1.54) 5.53( 2.12) 20 AG/CG 0.00( 0.00) 0.00( 0.00) 0.71( 0.00) 0.59( 0.08) 1.30( 0.08) 21 GC/GC 2.48( 0.13) 0.00( 0.00) 0.00( 0.00) 7.37( 3.95) 9.85( 4.09) 22 CU/AG 1.58( 0.11) 0.00( 0.00) 0.00( 0.00) 2.53( 2.08) 4.11( 2.19) 23 UC/GA 2.57( 0.90) 0.00( 0.00) 0.00( 0.00) 1.03( 0.07) 3.61( 0.97) 24 CA/UG 0.00( 0.00) 2.02( 0.00) 2.65( 0.33) 0.00( 0.00) 4.67( 0.33) 25 AG/CU 3.04( 1.09) 0.00( 0.00) 0.09( 0.00) 0.00( 0.00) 3.14( 1.09) 26 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Стэкинг-взаимодействие наиболее вероятно между самыми перекрывающимися парами. Самое большое перекрывание, у пары, помеченной звездочками (12-я).В файле stacking.pdb была найдена соответствующая двойка под номером 12. С помощью команды
ex_str -15 stacking.pdb step15.pdb данная структура была вырезана в отдельный файл, после чего, используя команду stack2img -cdolt step15.pdb step15.ps мы получили изображение структуры в формате .ps, для просмотра которого был использован on-line сервис http://view.samurajdata.se/. Получившееся изображение представлено на рисунке ниже.
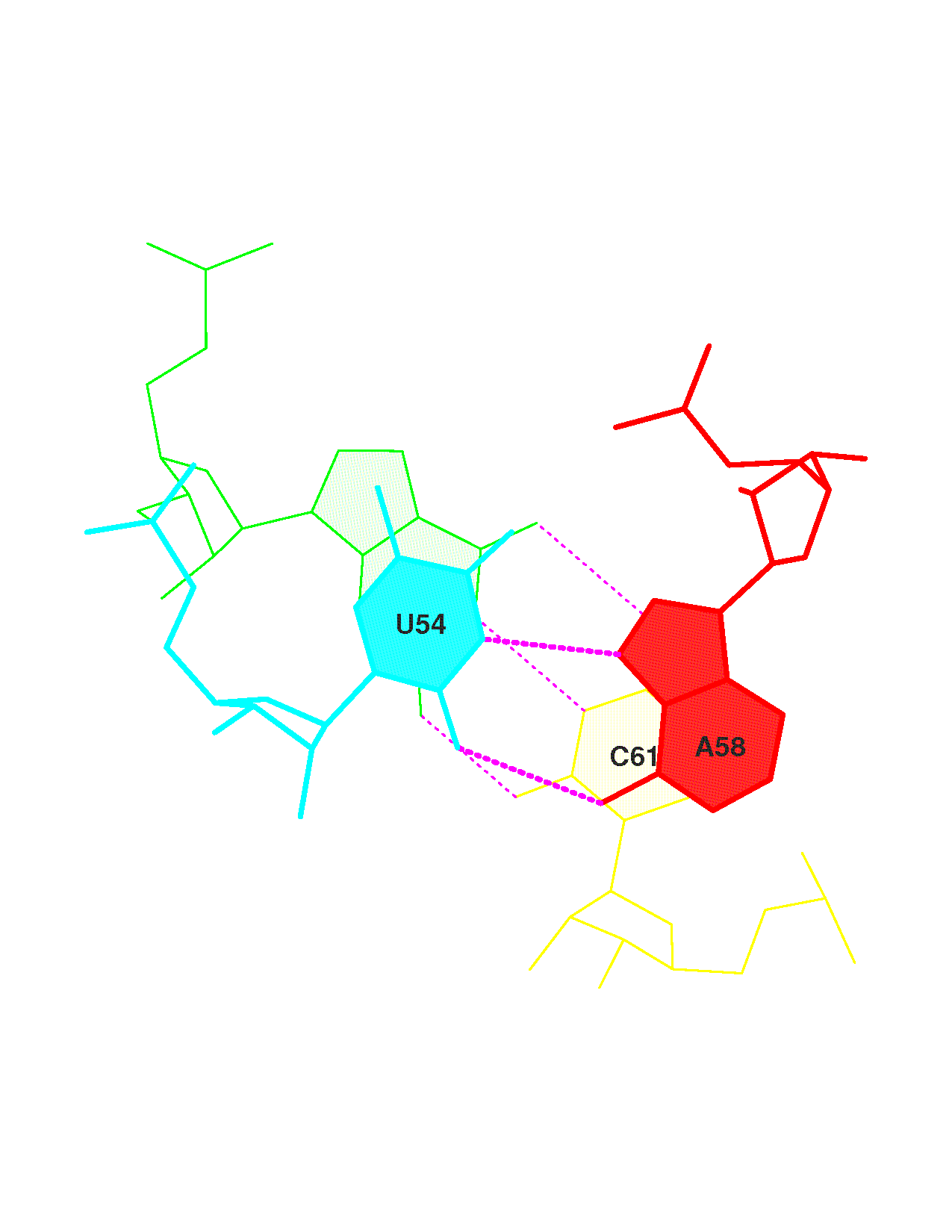
Назад к странице семестров