Сигналы
Мне досталась бактерия Peptoclostridium difficile, GenBank AC FN668375.1. С помощью веб-сервиса (используя метод Карлина) была получена таблица, содержащая информацию об ожидаемом количестве и контрасте всех сайтов из списка в геноме бактерии. Значение контраста (отношение наблюдаемое/ожидаемое число сайтов) находится в столбце Karlin's ratio. Сайты, ожидаемое число которых значительно отличается от числа наблюдаемых были найдены по принципу контраст меньше 0.78. Их оказалось 17, и они приведены в таблице результатов на листе "task1".
Далее требовалось проделать аналогичную работу с набором контигов той же бактерии из метагенома кишечника человека. С выходным файлом можно ознакомиться по ссылке Был найден уже 51 сайт рестрикции, против которых велся отбор, с их списком можно ознакомиться в таблице результатов на листе "task2".
Далее общие для двух штаммов сайты (всего 13) были найдены и сведены в сравнительную таблицу, приведенную на листе "task3". Ожидаемое количество сайтов в наборе контигов из метагенома для разных сайтов может быть как больше, так и меньше. Это может говорить о том, что оба штамма недавно потеряли разные рестриктазы.
Последовательность Шайна-Дальгарно — сайт связывания рибосом на молекуле мРНК прокариот, обычно на расстоянии около 10 нуклеотидов до стартового кодона AUG. Консенсусом является последовательность из шести нуклеотидов AGGAGG, ей комплементарна т.н. последовательность анти-Шайна-Дальгарно, расположенная на 3'-конце молекулы 16S рибосомной РНК — CCUCCU. Комплементарное взаимодействие между последовательностями Шайна-Дальгарно и анти-Шайна-Дальгарно служит для помещения старт-кодона мРНК в P-сайт рибосомы для начала биосинтеза белка.
При выполнении этого задания мне нужно было найти такие последовательности в геноме выданной мне в первом семестре бактерии Bacteroides thetaiotaomicron VPI-5482. Для этого были проделаны следующие шаги:
- Была найдена страница бактерии в БД Assembly на NCBI и последовательность хромосомы.
- Были сохранены фаста-файл с последовательностью хромосомы и таблица особенностей (Feature Table); последняя была обработана скриптом features2CDSs.py. Результат - таблица с координатами кодирующих последовательностей.
- Для дальнейшего анализа было выбрано 300 самых длинных кодирующих последовательностей. Мотив последовательности Шайна-Дальгарно не очень длинный, следовательно, не очень редкий. Поэтому разумно будет расширить область поиска до 16 нуклеотидов от старт-кодона. Области поиска последовательности Шайна-Дальгарно были вычислены с помошью логической функции ЕСЛИ в Excel. Требуемая для запуска скрипта таблица находится на второй вкладке таблицы результатов. Скрипт выдал последовательности фрагментов, по которым будет вестись поиск.
- Полученные последовательности были использованы для построения PWM на сайте MEME suit. Поиск проводился со следующими параметрами:

Был найден мотив, лого которого представлено на рис.2:
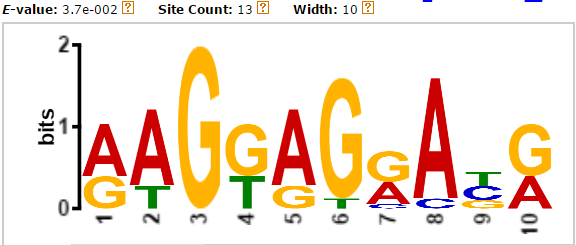
В полученном лого явно видна искомая последовательность Шайна-Дальгарно, так что найденную PWM будем использовать для поиска последовательности по всему геному. Для этого были получены координаты предполагаемого нахождения последовательности Шайна-Дальгарно для всех генов (расстояние - 16 нуклеотидов до старт-кодона). Искомые данные можно найти на третьей вкладке таблицы результатов. Найденная PWM была подана на вход алгоритму FIMO, который искал мотив последовательности Шайна-Дальгарно уже по всем генам. Порог E-value был поднят до 0.01. Параметры запуска алгоритма - на рис.3:

В результате было найдено 722 гена, удовлетворяющих заданным условиям. Выдача программы приведена в таблице результатов на четвертой вкладке.