Домены и профили

Наименования пары: DinB_2, FGE-sulfatase x 2
PF12867 (10664 seq) PF03781 (14664 seq)
Число последовательностей с подобной архитектурой: 688
Запрос в uniprot: taxonomy: "Bacteria [2]" database:(type:pfam pf03781) database:(type:pfam pf12867)
скачать табличку
Распределение длин последовательностей:
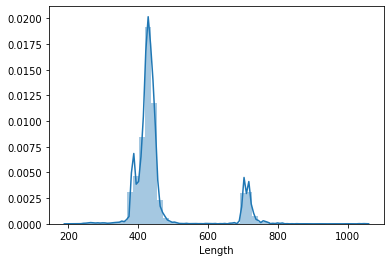
Так как семейств оказалось больше, чем 60, для того, чтобы выбрать последовательности характерной длины, было взято 30 самых представленных семейст и из них по 2 самых представленных рода.
скачать табличку с 60 последовательностями
С помощью Jalview были построены выравнивания (последовательности получены через fetch sequences из uniprot) при помощи WebService->Muscle. ссылка на необрезанное выравнивание. Далее я обрезала выравнивание после 604 позиции и перед 56-й позицией. (ссылка на выравнивание).
Затем по выравниванию был построен и откалиброван профиль командами
hmm2build -g pr9.hmm alignment_hmm.fa
hmm2calibrate pr9.hmm
Будем рассматривать домен 03781. Последовательности были скачаны из uniprot при помощи команды:
taxonomy:"Bacteria [2]" database:(type:pfam pf03781)
При помощи команды hmm2search был произведен поиск, то есть последовательности с доменом pf03781 выравнивались с hmm профилем. Команда:
hmm2search -E 0.1 pr9.hmm domain.fasta > findings.txt
ссылка на файл findings.txt
Затем через какое-то время я поняла, что в этом файле лежат не только нужные мне колонки, но и выравнивания. Поэтому я вырезала нужную часть и обработала в питоне при помощи библиотеки sklearn (функция roc_curve).
ссылка табличку с нужными колонками
В таблице по столбцам находки из юнипрота, по строкам выборка HMM.
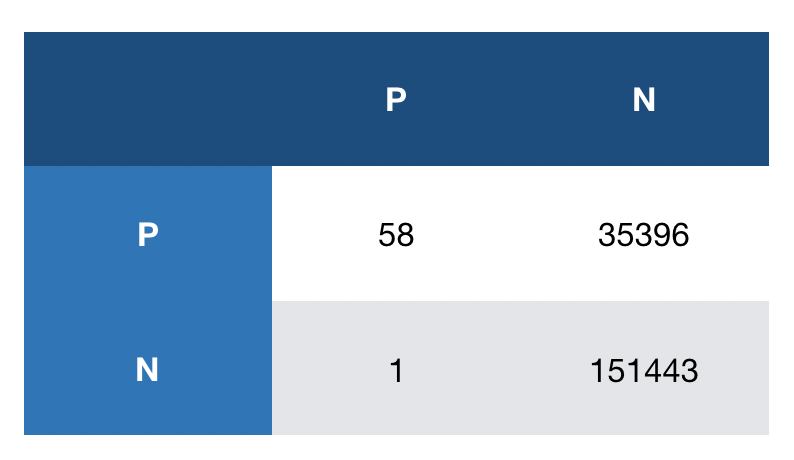
Получились такие картинки:
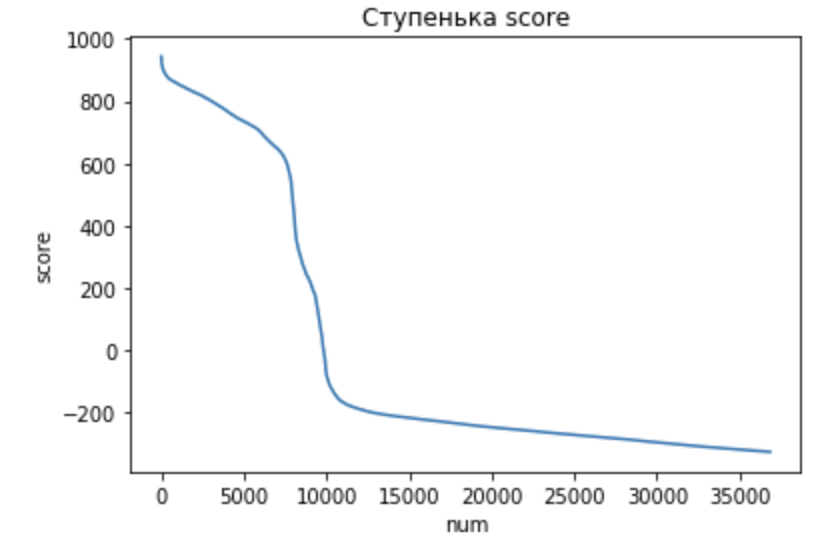
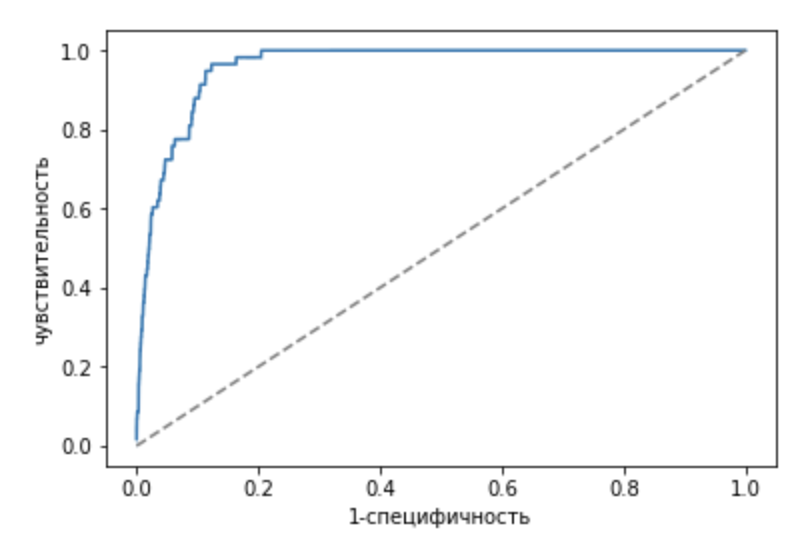
Порог: 928.6.
Кривая лежит выше диагонали, значит, данный порог позволяет предсказывать лучше, чем случайное предсказание