Описание олигосахарид-4-альфа-D-глюкозилтрансферазы из генома бактерии Cellvibrio japonicus Ueda107
Основная информация о белке
Здесь Вы можете найти описание фермента из генома грамотрицательной почвенной бактерии Cellvibrio japonicus. Информация была получена из базы данных NCBI, а именно из опубликованного в 2008 году полного генома бактерии. Название белка - из базы данных Uniprot. Все данные приведены в таблице 1.
| Идентификатор белка | ACE84782.1 |
|---|---|
| Идентификатор генома | CP000934.1 |
| Название белка | Oligosaccharide 4-alpha-D-glucosyltransferase, EC 2.4.1.161 |
| Координаты гена в геноме | 3881837..3884287 |
| Длина гена (п.н.) | 2451 |
| Цепь | Прямая |
| Длина белка (а.о.) | 816 |
Вы можете скачать аминокислотную последовательность белка в FASTA формате.
ACE84782.1.fasta
Она была получена из базы данных NCBI. Кодировка файла - ASCII, кодировка концов строк - Unix. Длина одной строки последовательности - 60 символов.
Данный фермент переносит невосстанавливающий концевой остаток альфа-D-глюкозы от альфа-1,4-D-глюкана (к ним относятся, например, гликоген
и амилоза) на C4-атом свободной глюкозы или остатка глюкозила на невосстанавливающем конце альфа-1,4-D-глюкана. Это обеспечивает
реорганизацию олигосахаридов в клетке. Изучение различных ферментов, отвечающих за превращения углеводов в клетке Cellvibrio japonicus,
представляет особенный интерес, так как эта бактерия может разлагать клеточную стенку растений, состоящую из сложной сети соединенных друг с другом
полисахаридов. Например, в 2012 году у этой бактерии был обнаружен новый тип трансглюкозилаз, имеющий наиболее высокую активность с мальтотриозой в качестве
донора остатка (аналогичный фермент из амебы не работает с мальтозой). Принципы работы многих ферментов еще не описаны, около многих из них, в том
числе около приведенного здесь белка, в описании генома Cellvibrio стоит надпись "putative" - "предполагаемый". Впрочем, с 2008 года,
когда последовательность была опубликована, что-то могло проясниться.
Источники:
1. https://www.ncbi.nlm.nih.gov/protein/ACE84782.1>
2. DeBoy R. T., Mongodin E. F., Fouts D. E., Tailford L. E., Khouri H., Emerson J. B., Mohamoud Y., Watkins K., Henrissat B.,
Gilbert H. J., Nelson K. E. (2008) Insights into plant cell wall degradation from the genome sequence of the soil bacterium
Cellvibrio japonicus. J. Bacteriol. 190, 5455–5463
[PubMed]
3. Johan Larsbrink, Atsushi Izumi, Glyn R. Hemsworth, Gideon J. Davies, Harry Brumer (2012) Structural Enzymology
of Cellvibrio japonicus Agd31B Protein Reveals α-Transglucosylase Activity in Glycoside Hydrolase Family. J Biol Chem. 287(52): 43288–43299
[PMC free article]
Геномное окружение гена белка
Информация о геномной окружении была получена с помощью геномного браузера NCBI в режиме просмотра рамок считывания. Полученные данные приведены на рисунке 1.
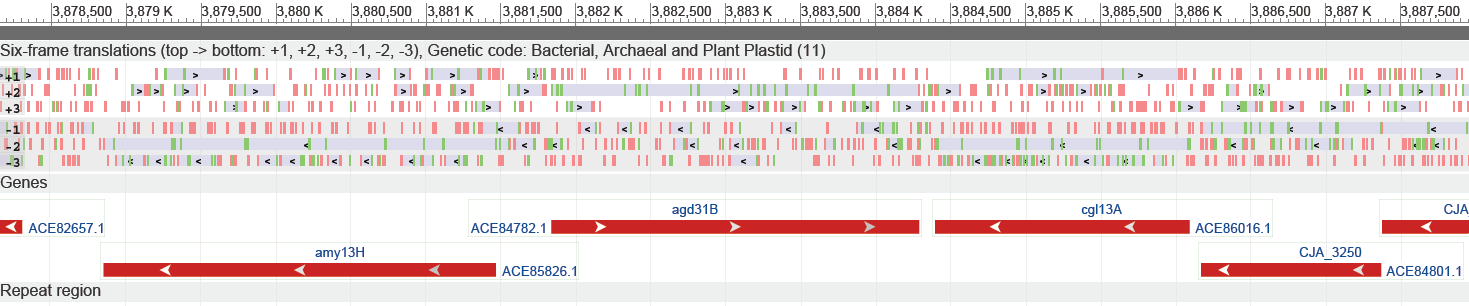
Белыми стрелками показана цепь ДНК; у нашего белка они направлены вправо, значит цепь прямая. 6 строчек наверху - 6 рамок считывания (по 3 на каждой цепочке). Зеленые области - старт-кодоны, красные области - стоп-кодоны, серые - открытые рамки считывания. Очевидно, что гену исследуемого белка соответствует рамка считывания +2. Серых областей больше, чем старт-кодонов, так как выделяются только открытые рамки считывания длины больше 19 кодонов.
Исследуемый ген закодирован на прямой цепи, в отличие от подавляющего большинства генов из его окружения. Рамка считывания: +2.
Итог практикума:
Разбираться в статьях и в геномном браузере интересно, а делать дизайн долго.
Главный вывод после того, как я 3 часа выбирал цвет фона и не выбрал: мне не стоит идти в веб-дизайнеры.