Поиск гомологов белка Dihydroxyacetone kinase из бактерии Citrobacter freundii
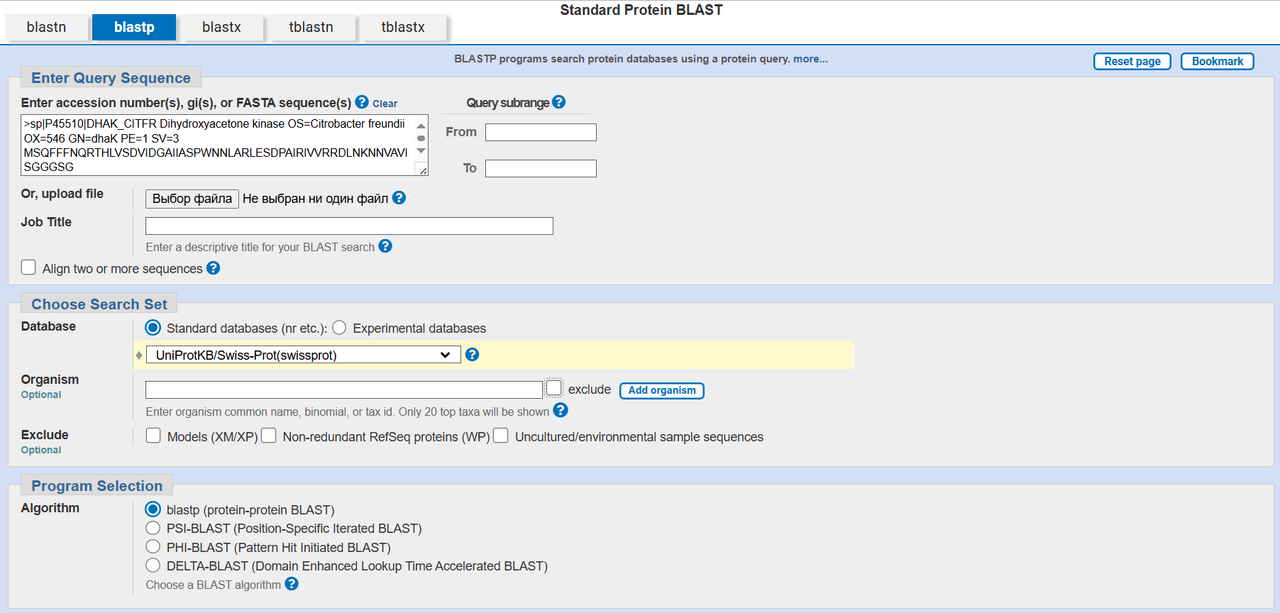
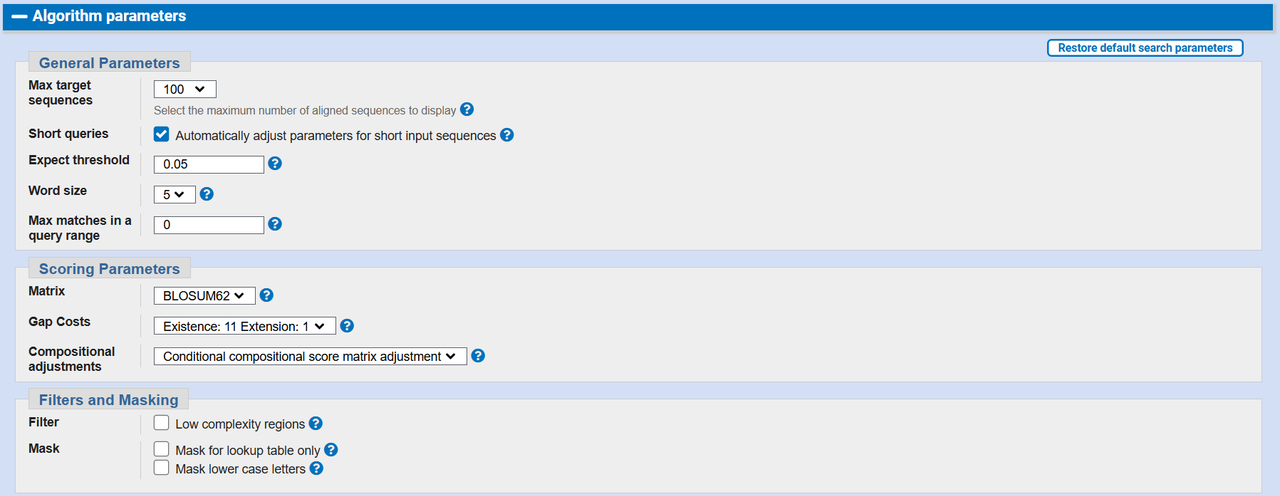
При запуске BLAST были введены следующие параметры:

Вообще у белка довольно базовая функция (катализирует фосфорилирование дигидроксиацетона) и ожидалось большое количество результатов, но получили 24 последовательности. По всей видимости белок специфичен и в основном в организмах присутствуют его аналоги.
Текстовый файл с результатами поискаНаиболее идентичным оказался белок из Solanum lycopersicum (томат). Затем идёт ряд млекопитающих, грибов и только потом с меньшим процентом идентичности белки из других бактерий. Были выбраны:
O04059 RecName: Full=Putative 3,4-dihydroxy-2-butanone kinase [Solanum lycopersicum]
Q3LXA3 RecName: Full=Triokinase/FMN cyclase [Homo sapiens]
Q4KLZ6 RecName: Full=Triokinase/FMN cyclase [Rattus norvegicus]
Q55EE0 RecName: Full=Probable dihydroxyacetone kinase [Dictyostelium discoideum]
A0R758 RecName: Full=L-erythrulose 1-kinase [Mycolicibacterium smegmatis MC2 155]
По выравниванию можно сказать, что белки являются гомологичными. Наибольшее количество точечных несовпадений наблюдается у Mycolicibacterium smegmatis MC2 155. В целом у белка из этого организма такой же принцип работы, просто он катализирует фосфорилирование не дигидроксиацетона, а L-эритрулозы. В его пространственной конформации тоже можно заметить отличие. 2 домена более приближены друг к другу.
Ещё примечателен белок Probable dihydroxyacetone kinase из Dictyostelium discoideum. Его конфигурация схожа с нашим белком, но на выравнивании у него можно заметить вставки, которые в структуре его выступают петлями в одном из доменов.
У остальных белков ничего интересного по сравнению со структурой исходного белка нет. Если говорить про функции, то можно выделить ещё белок из томата.
Гомологи Polyprotein P1234 из Aura virus (AURAV)
ID: POLN_AURAV
AC: Q86924
В полипротеин входит 8 разных белков. Выберем RNA-directed RNA polymerase nsP4 (1890-2499). Вырежем его последовательность в отдельный файл с помощью команды:
seqret 'sw:poln_aurav[1890:2499]' segment.fasta
Текстовый файл выдачи BLASTВ выдаче 35 результатов. Для 27 E-value очень малы (0.0), у них высокий процент идентичности и процент покрытия.
Для выравнивания были выбраны:
Q86924 RecName: Full=Polyprotein P1234 [Aura virus]
P03317 RecName: Full=Polyprotein P1234 [Sindbis virus]
P27283 RecName: Full=Polyprotein P1234 [Ockelbo virus]
P08411 RecName: Full=Polyprotein P1234 [Semliki Forest virus]
P87515 RecName: Full=Polyprotein P1234 [Barmah Forest virus]
P13896 RecName: Full=Polyprotein P1234 [Western equine encephalitis virus]
В целом белки гомологичны, но у них можно выделить более вариабельный участок в начале последовательности и консервативный, начиная где-то с 269 и до конца.
Исследование зависимости E-value от объёма банка
RNA-directed RNA polymerase 2a из Cowpea chlorotic mottle virus (P20179) при первом поиске выдало E-value = 7e-07. При указании организмов значение составило 3е-08 (файл выдачи BLAST).
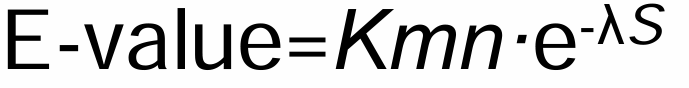
Из всех этих переменных мы изменили размер базы данных. Найдём долю вирусных организмов: 3е-08/7e-07 = 0,0429 или 4,3%.