Практикум 10. Мотивы в белках, паттерны и PSSM для их поиска.
Задание 1
Для данного практикума я выбрала домен PF13346. Согласно описанию в Pfam, это домен ABC2-мембранных транспортеров.
В выравнивании seed 42 последовательности. Удаление похожих посл-тей с порогом сходства 90% не дало результата, все 42 последовательности остались.
Я произвела покраску по порогу идентичности, при пороге идентичности 65% выделился консервативный участок с 94 по 106 остаток. Данную последовательность можно описать паттерном LP.{2}R.{3}V.{2}KY. При поиске в выравнивании JalView по составленному паттерну был найден только фрагмент, по которому паттерн был составлен - значит, паттерн действительно является уникальным и хорошо характеризует выбранный участок выравнивания.
В формате Prosite паттерн можно записать как L-P-x(2)-R-x(3)-V-x(2)-K-Y. При поиске в ScanProsite по базе SwissProt было найдено всего две находки - белок вителлогенин из Bombyx mori (VIT_BOMMO) и неохарактеризованный белок aq_272 из Aquifex aeolicus (strain VF5) (). Следовательно, паттерн был задан слишком узко, попробуем немного укоротить его до R-x(3)-V-x(2)-K-Y (поскольку наиболее консервативны R и Y, а L слева от R не является настолько консервативным). По данному запросу в ScanProsite нашлось 113 белков, в основном бактериальных. Есть среди них, конечно, случайные находки (например, белок капсида одного из вирусов, который, конечно, не имеет отношения к ABC2-транспортерам, однако большое количество найденных белков - это GTPase Der из различных бактерий, белок обладающий ГТФазной активностью. Эта находка меня порадовала, поскольку этот белок обладает ГТФазной активностью, а наш ABC2-транспортер обладает АТФазной (хоть какое-то сходство и какой-то успех!). Также из находок, имеющих отношение к транспортерам - Putative oligopeptide transporter YGL114W дрожжей Saccharomyces cerevisiae (YGL4_YEAST).
Задание 2
Далее в JalView было построено по выравниванию филогенетическое дерево методом NJ. Была отделена клада, содержащая последовательности E6TUT4_EVAC2, Q5WLF0_ALKCK, Q7AJY1_HALH5, U5LJF0_9BACI, для этой клады построено выравнивание. В пределах данной клады последовательности оказались очень консервативными, я выбрала мотив NILLNSLPYTR (с 58 по 68 остатки в выравнивании). При поиске этого мотива во всем выравнивании оказалось, что он встречается только в 4 последовательностях клады и больше нигде в выравнивании, что говорит о том, что данный мотив специфичен для выбранной клады.
Задание 3
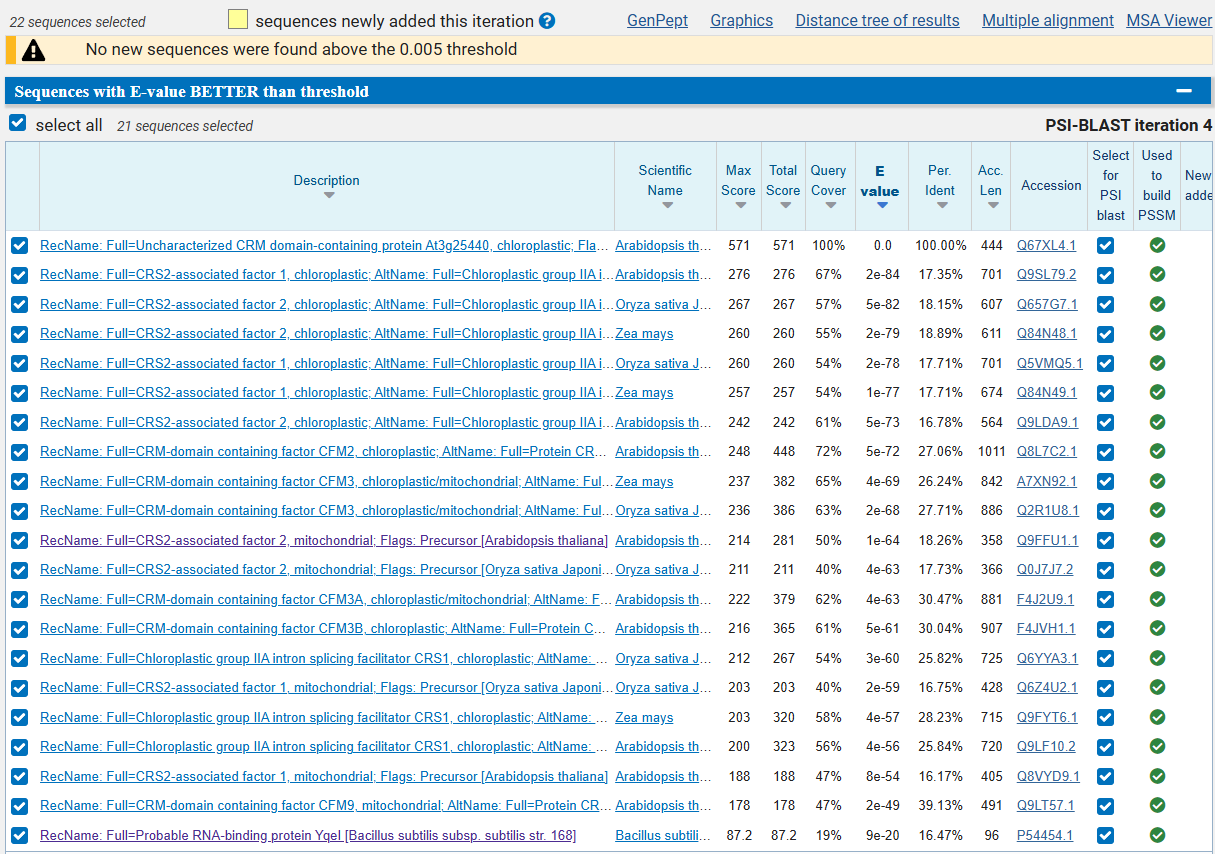
Целью задания был поиск гомологов выбранного белка с помощью PSI-BLAST. Я выбрала из списка белок Q67XL4 - это хлоропластный белок At3g25440 с доменом CRM (в UniProt он описывается как 'Uncharacterized CRM domain-containing protein At3g25440, chloroplastic') из Arabidopsis thaliana. Для данного белка был проведен поиск PSI-BLAST по базе SwissProt. После 4 итерации список находок перестал увеличиваться.
Таблица 1. Результаты поиска гомологов белка Q67XL4 программой PSI-BLAST.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 18 | Q9FFU1.1 | 0.004 | Q67XL4.1 | 0.0 |
| 2 | 20 | Q9LDA9.1 | 2,00E-29 | Q67XL4.1 | 0.0 |
| 3 | 21 | P54454.1 | 2,00E-05 | Q67XL4.1 | 0.0 |
| 4 | 21 | P54454.1 | 8,00E-20 | Q67XL4.1 | 0.0 |
В целом, результаты поиска вполне предсказуемы - найдены хлоропластные и митохондриальные белки из растительных организмов. Меня удивила только находка с самым высоким e-value, добавившаяся на последней итерации - РНК-связывающий белок Bacillus subtilis, но после того как я уточнила, что CRM - это РНК-связывающий домен, эта находка вызывает у меня уже меньше удивления.
Задание 4
Домену CRM соответствует идентификатор IPR001890 в базе InterPro, где содержится 28 белков с данным доменом и статусом 'reviewed'. Последовательности этих 28 белков были скачаны, выравнены в JalView с помощью muscle. После удаления сходных последовательностей ('remove redundancy' с порогом 90%) осталось 19 последовательностей. Эти последовательности были сохранены в файл, из них были удалены все гэпы, в результате чего получился файл prac10_4_nogaps.fasta.
Далее на Kodomo была запущена программа MEME: meme prac10_4_nogaps.fasta -o results -mod anr -minw 4 -maxw 8 -nmotifs 4. Все 4 найденных мотива имели низкие e-value. Для мотива с самым низким e-value (мотив ILYRGKBY; e-value 1.2e-114) было далее проверено число вхождений с помощью FIMO.
Запуск FIMO на Kodomo: fimo --norc -motif ILYRGKBY results/meme.txt prac10_4_nogaps.fasta. FIMO показала, что мотив встречается 48 раз; поскольку последовательностей белков 19, то такой результат может говорить о том, что в некоторых белках мотив встречается несколько раз.
Задание 5
Целью задания было оценить представленность сайтов GATC в геноме бактерии Xanthomonas cucurbitae. Был использован файл permutations.txt, содержащий все возможные перестановки из букв G, A, T, C.
На Kodomo была запущена программа cbcalc для вычисления контрастов: cbcalc -s permutations.txt -M -o res_calc.tsv ../prac9/genome.fna. Результат работы программы - файл res_calc.tsv, представляющий собой таблицу, в которой для каждой перестановки в столбце O/E ratio (MM) записано отношение наблюдаемого числа таких тетрануклеотидов (Observed) к ожидаемому их числу (Expected).
Была построена гистограмма, отражающая O/E ratio для перестановок каждого вида (гистограмма контрастов obs/exp):
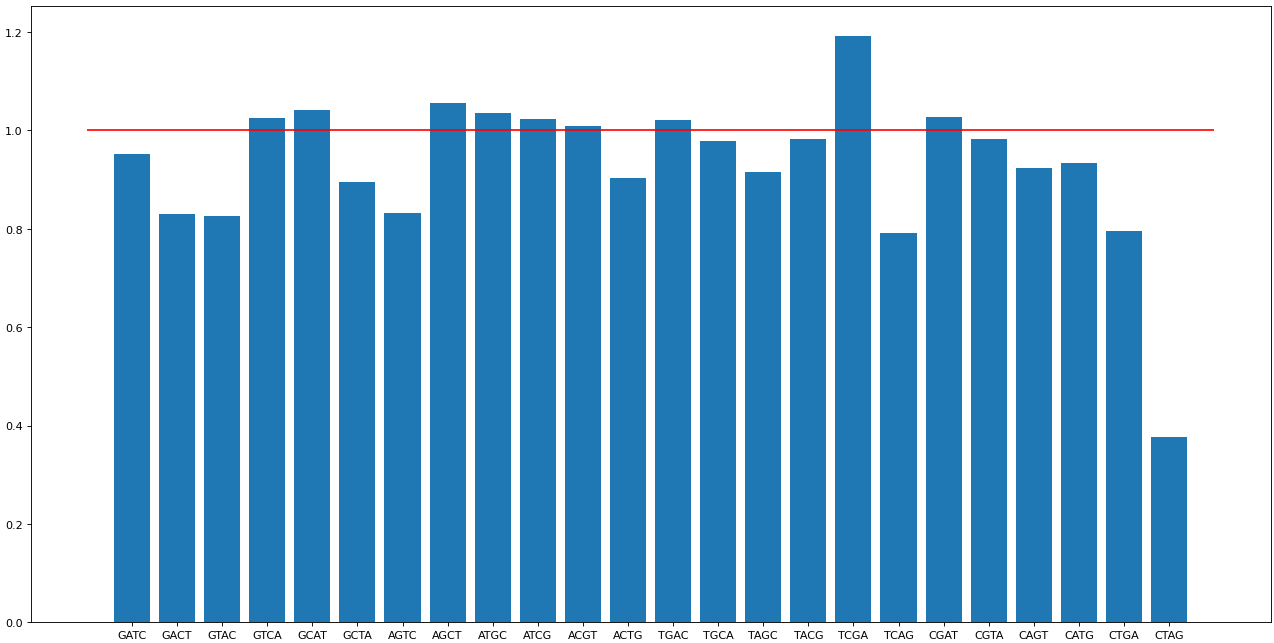
Из полученной гистограммы видно, что сайт GATC в геноме бактерии немного "недопредставлен" - отношение O/E меньше единицы (0.953) говорит о том, что он встречается реже, чем ожидалось бы. А самый перепредставленный сайт из исследованных тетрануклеотидов - это TCGA, у него самое высокое отношение O/E (1.193).