| Главная страница | Семестры | О себе | Ссылки |
BLAST - семейство компьютерных программ, предназначенных для поиска белковых или нуклеотидных последовательностей, сходных с данной по первичной структуре, что может быть признаком гомологии.
Поиск гомологов ДНК-лигазы Streptococcus pneumoniae штамм P1031
Для поиска гомологов ДНК-лигазы (последовательность аминокислот) я выбрала BLAST на сайте NCBI. Поиск осуществлялся по базе данных RefSeq. Из огромного количества находок я выбрала одну, информация о которой представлена в таблице 1 и на рисунке 1.
Таблица 1. Параметры выравнивания выбранного гомолога
Параметр |
Расшифровка |
Значение |
Description |
Название и описание находки |
DNA ligase [Bifidobacterium pseudolongum] |
Max score |
Максимальный вес выравнивания |
323 |
Total score |
Общий вес выравнивания |
417 |
Query cover |
Процент покрытия запроса находкой |
90% |
E value |
Количество последовательностей с тем же или большим весом выравнивания |
1е-99 |
Ident |
Процент идентичности запроса и находки |
42% |
Accession |
Идентификатор находки |
WP_022857699.1 |
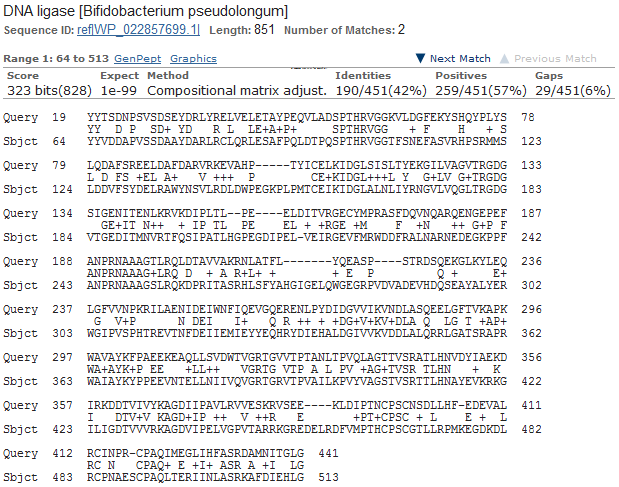
Рис. 1. Выравнивание находки и запроса
Карта локального сходства между ДНК-лигазами Streptococcus pneumoniae и Bifidobacterium pseudolongum
Карта локального сходства - наглядное отображение наличия или отсутствия сходства двух последовательностей. По осям отложены координаты аминокислотных остатков, учитывавшихся в выравнивании. Линия не прерывается, если остатки, соответствующие координате точек линии по оси абсцисс, совпадают с такими же остатками по оси ординат. Свою карту локального сходства я строила, опять же, с помощью BLAST NCBI, введя в окошки последовательность своего белка и последовательность находки.
При построении карты к выравниванию добавляется еще 2 фрагмента, в итоге на карте (рисунок 3) отображено 3 участка совпадения. Характеристики выравнивания меняются (см. рисунок 2).

Рис. 2. Выравнивание ДНК-лигаз, построенное по последовательностям аминокислотных остатков
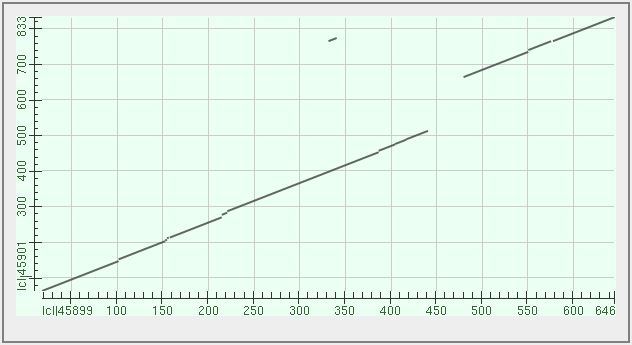
Рис. 3. Карта локального сходства между последовательностями
Далее в тексте последовательность ДНК-лигазы пневмококка я буду называть последовательностью 1, последовательность такого же белка бифидобактерии - последовательностью 2.
Как видно из рисунков 1 и 2, первая линия на карте (посл. 1: с 19 по 441 а. к.; посл. 2: с 64 по 513) покрывает большую часть обеих последовательностей, препываясь в местах, соответствующим усчасткам с большим числом гэпов в посл. 1: от 104 до 108, от 158 до 161, от 216 до 222, от 229 до 232, от 386 до 389 а. к. По этой линии точно наблюдается гомология последовательностей, так что разрывы на ней соответствуют делециям остатков из последовательности 1 или инсерции остатков в последовательность 2.
Вторая линия на карте (посл 1: с 481 по 646 а. к.; посл. 2: с 665 по 833 а. к.). Этот участок сходства тоже довольно большой, так что там явно есть гомология. В линии есть 2 разрыва: первый соответствует делеции остатков на позициях 552-556 в посл. 1 или инсерции с 736 по 740 остатков в посл. 2, второй - инсерции 577 и 578 остатков в посл. 1 или делеции двух остатков, соотвкетствующим 764 и 765 позициям посл. 2.
Третья линия на ккарте (посл 1: с 333 по 341 а. к.; посл. 2: с 766 по 774 а. к.) очень короткая, находится в стороне от других двух линий и вообще выбивается из картины. Значит, сходство между последовательности там случайно и гомологии на самом деле нет.
Эукариотические гомологи ДНК-лигазы пневмококка
Для поиска эукариотических гомологов своего белка я использовала ту же программу и базу данных, что и для предыдущего задания. В поле "Organism" я ввела "Eucaryotae". В итоге мне было выдано достаточно много последовательностей (452), из которых 297 имели Е value < 0,001, то есть, скорее всего, гомологичные данной. Что интересно, у значительной части находок нашлось совпадение с одним и тем же участком запроса - концевым. Возможно, он выполняет важную для многих клеток функцию, например, связывание с ДНК. Из 297 находок я выбрала 10 с большими значениями Query cover, сохранила их (последовательности в формате fasta) и составила выравнивание в JalView с помощью muscle. Более подробно о выравнивании можно узнать из таблицы 2.
Таблица 1. Статистические характеристики выравнивания эукариотических гомологов ДНК-лигазы
Число позиций |
Процент |
|
Все позиции |
2419 | 100% |
Абсолютно консервативные |
16 | 0,661% |
Абсолютно функционально консервативные |
12 | 0,496% |
Из таблицы следует, что нельзя говорить о гомологии между последовательностями. Но и нельзя сказать, что совпадения случайны: они не равномерно распределены по выравниванию, а собраны в неплохие блоки. Так что возможна гомология между частями последовательностей.
© Дарья Горбачева | изменено 8.08.2014 |

