| Главная страница | Семестры | О себе | Ссылки |
JalView - программа для наглядного отображения множественных выравниваний. Ее удобство и польза заключается в возможности наглядного отображения степени консервативноести позиций в выравнивании (процента совпадения аминокислотных остатков в "столбике"). Чем больше консервативных позиций находится рядом, тем вероятнее гомология на этом участке выравнивания.
Поиск гомологичных участков
Для выполнения данного задания мне нужно было построить с помощью Jalview выравнивание нескольких последовательностей (я выбрала эти 15). Полное выравнивание имеет такой вид (здесь и далее использована раскраска Clustalx при консервативности более 70%).
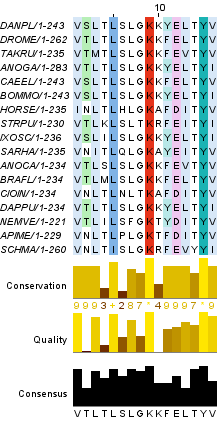
В данном выравнивании есть несколько блоков, то есть участков, на которых можно ожидать гомологию всех аминокислот. 2 из них приведены на рисунке 1.
|
|
Рис. 1. Блоки в выравнивании
Таблица 1. Статистические характеристики левого блока (позиции 96-111)
Число позиций |
Процент |
|
Все позиции |
16 | 100% |
Абсолютно консервативные |
4 | 25% |
Абсолютно функционально консервативные |
6 | 37,5% |
Консервативные на 70% |
3 | 18,75% |
Функционально консервативные на 70% |
3 | 18,75% |
На рисунке 2 представлен участок последовательности, на котором скорее всего наблюдается гомология первых двух последовательностей (DANPL и BOMMO, совпадают на 75%), но не гомология остальных.

Рис. 2. Участок выравнивания, на котором, скорее всего, есть гомология между первыми двумя последовательностями
На рисунке 3 представлен участок выравнивания, содержащий 2 близко расположенных вертикальных блока и позиции межлу ними. Из 10 позиций участка между блоками 2 содержат гэпы, значит, процент позиций с гэпами 20%.
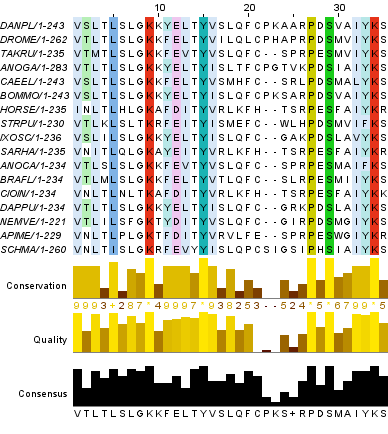
Рис. 3. Участок выравнивания с двумя блоками
Выравнивание, с которым я работала, и его части содержатся в этом проекте.
Добавление новой последовательности к уже имеющемуся выравниванию
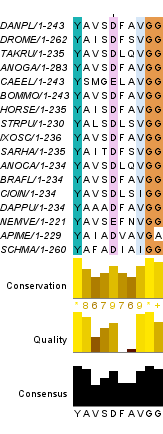
К своему выравниванию я добавила последовательность и вручную выровняла с блоком, изображенным на рисунке 1 справа. Я использовала функцию поиска участков последовательности с регулярным выражением ".", обозначающим любую аминокислоту. Один из вариантов представлен на рисунке 4.

Рис. 4. Блок, выровненный вручную с добавленной последовательностью (последняя)
Согласно информации, представленной в окне JalView, консенсусная последовательность для выравнивания с рисунка 4:
>Consensus/1-10 Percentage Identity Consensus YAVSDFAVGG

Рис. 5. Консенсус, полученный с помощью сервиса Weblogo
Само выравнивание можно скачать здесь.
Множественное выравнивание заведомо негомологичных белков
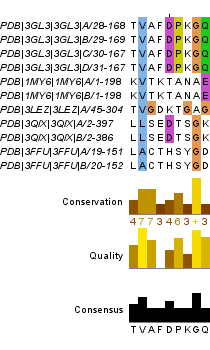
Для выполнения этого задания я случайным образом выбрала белки из тех, с которыми работают мои однокурсники. Идентификаторы PDB: 3GL3; 1MY6; 3LEZ; 3QIX; 3FFU. Для выравнивания был использован web-сервис Muscle.
На получившееся вырвнивание можно посмотреть здесь. На все выравнивание, содержащее 396 позиций, приходится всего 3, консервативных более чем на 70%, и, вероятнее всего, эти совпадения случайны. Во многих местах наблюдается гомология между последовательностями субъединиц одного белка, но не всех белков выравнивания.
|
|
Рис. 6. "Блоки" в выравнивании негомологичных последовательностей. Для изображения этих блоков выбрана окраска Crustalx, не учитывающая консервативность, из-за того что консервативность позиций очень низкая.
Проект в формате jar с выравниванием можно скачать здесь.
© Дарья Горбачева | изменено 8.08.2014 |