| Главная страница | Семестры | О себе | Ссылки |
Данный практикум выполнялся с помощью сервиса PDBeFold, который позволяет находить в базе PDB белки, аминокислотные последовательности, схожие с последоватедьностью заданного, и отображать наложение пространственных структур белков. С помощью этого сервиса я искала белки, похожие на ДНК-лигазу А бактерии Streptococcus pneumoniae с идентификатором PDB 4GLW.
Поиск со стандартными настройками выдал мне 24 последовательности, 2 из которых представляли собой сами цепи ДНК-лигазы. Всего было обработано 95948 структур, 247648 цепей.
Из 22 вариантов для анализа соответствия я выбрала цепь С ДНК-лигазы Enterococcus faecalis штамм V583 (идентификатор PDB: 1TAE). Сравниваемые ферменты выполняют одну и ту же функцию - являются НАД-зависимыми ДНК-лигазами, то есть сравниваю я два "одинаковых" белка бактерий, принадлежащих одному порядку (Lactobacillales), но разным семействам (Streptococcaceae и Enterococcaceae). В таблице 1 представлена информация о совмещении полипептидных цепей белков.
Таблица 1. Параметры, описывающие схоство 4GLW:A и 1TAE:C
Название на сервисе PDBeFold |
Расшифровка |
Значение |
RMSD |
Особым образом расчитываемая мера сходства структур |
1.21 |
N_align |
Число сопоставляемых CA-атомов |
237 (86% от числа аминокислот 4GLW:A) |
N_g |
Число гэпов |
2 |
%seq |
Процент совпадающих остатков |
59 |
%sse |
Процент совпадающих элементов вторичной структуры |
86 |
Совмещение полипептидных цепей белков представлено на рисунках 1 и 2. Видно, что на достатьчно большом участке цепи белков ведут себя схожим образом, но они далеки от того, чтобы быть названными идентичными.

Рис. 1. Общий вид совещения цепей 4GLW:A (желтый) и 1TAE:C (лиловый), шариками показано "геометрическое ядро" - СА-атомы, расстояние между которыми не превышает 1,5 ангстрем
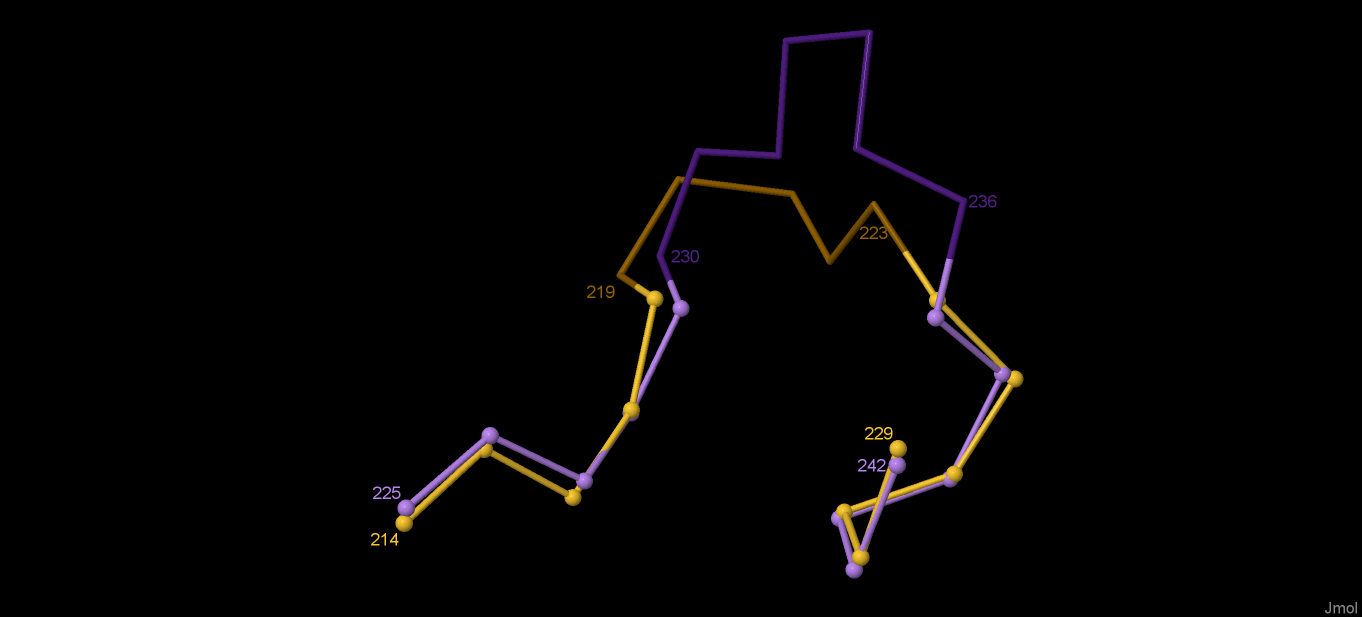
Рис. 2. Участок совмещения цепей. Темным показан несовпадающий участок пространственных структур. Цифры - номера остатков в цепях
© Дарья Горбачева | изменено 8.08.2014 |

