| Главная страница | Семестры | О себе | Ссылки |
Поиск мотива
ProSite - сервис для поиска мотивов, то есть консервативных участков белковых последовательностей. Для поиска мотивов были выбраны последовательности гомологов ДНК-лигазы из предыдущего практикума, так как белки, последовательности которых включены в выравнивание, были найдены через поиск вероятных гомологов, значит, и мотивы в этом выравнивании будут свидетельствовать о гомологии.
В восьми последовательностях было найдено 17 участков, подходящих под один из трех мотивов: BRCT, DNA_LIGASE_N1, DNA_LIGASE_N2. Для построения паттернов мной был выбран мотив DNA_LIGASE_N2 (NAD-dependent DNA ligase signature 2, PS01056), так как встречается в НАД-зависимых (прокариотических) ДНК-лигазах, в отличие от BRCT domain, который содержится в широком спектре белков, скорее всего, негомологичных.
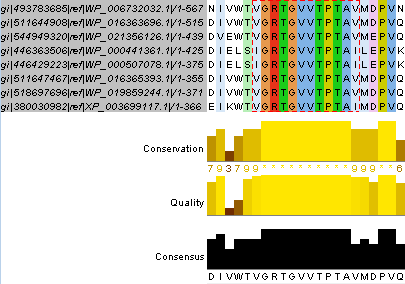
Рис. 1. Участок в выравнивании (полученное с помощью mafft), соответствующий выбранному мотиву - в красной рамочке
Составление паттернов
Сильный паттерн
Все соответствующие мотиву колонки очень консервативны, так что тут проблем с составлением паттерна не возникло:
[VI]-G-R-T-G-V(2)-T-P-T-A-[VI]-[ML]-[ED]-P-V |
С помощью мотива было найдено 38 последовательностей в SwissProt и 793 - в TrEMBL. Все найденные последовательности принадлежат ДНК-лигазам, прокариотическим (возможны единичные исключения, которые я проглядела), значит, паттерн, скорее всего, специфичен для НАД-зависимых ДНК лигаз.
Слабый паттерн
Для того, чтобы ослабить требования на колонки, мне пришлось построить выравнивание с большим числом гомологов (20).

Рис. 2. Гомологи ДНК-лигазы, выбранные для построения более полного выравнивания
Полученное выравнивание, как и выравнивание с рисунка 1, было получено с помощью mafft.
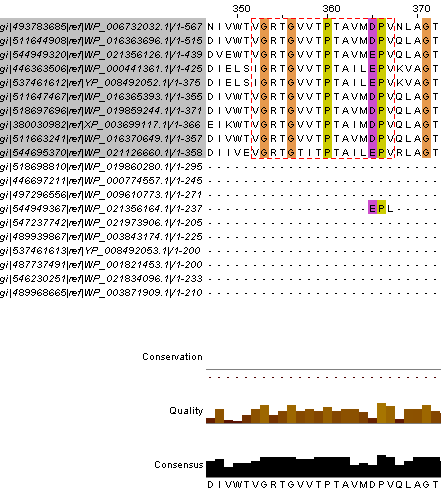
Рис. 3. Участок в "расширенном" выравнивании, соответствующий выбранному мотиву - в красной рамочке
К сожалению, большого результата все это не дало - половина последовательностей из нового выравнивания совсем не содержит выбранного мотива, так что сильно ослабить паттерн не удается. Однако я попробую без явных предпосылок расширить некоторые позиции близкими по свойствам остатками:
[VIL]-G-[RK]-T-G-[VT]-[VLI]-T-P-T-A-[VLI]-[MLI]-[DE]-P-V |
Было получено 49 находок в SwissProt и 956 в TrEMBL (все ДНК-лигазы), из чего можно заключить, что "слабый" паттерн на деле ненамного слабее сильного, представленного выше.
Исходный паттерн
Также я решила провести поиск по паттерну, указанному в описании выбранного мотива:
[IV]-G-[KR]-[ST]-G-x-[LIVM]-[STNK]-x-[VTLYF]-x(2)-[LVMF]-x-[PS]-[IV] |
Очевидно, этот паттерн гораздо слабее предыдущих двух, что согласуется с полученными результатами: 703 находоки в SwissProt, количество находок в TrEMBL превысило лимит в 10000, и при этом все находки остаются ДНК-лигазами.
© Дарья Горбачева | изменено 8.08.2014 |

