| Главная страница | Семестры | О себе | Ссылки |
Построение моделей структур A-, B- Z-форм ДНК
Был использован пакет 3DNA - набор программ для простейшего моделирования и анализа структур нуклеиновых кислот. С его помощью были получены модели трех форм ДНК:
Структуры были получены с помощью команд:
|
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin export X3DNA=/home/preps/golovin/progs/X3DNA fiber -a gatc-a.pdb fiber -b gatc-b.pdb fiber -z gatc-z.pdb |
Обработка структур нуклеиновых кислот
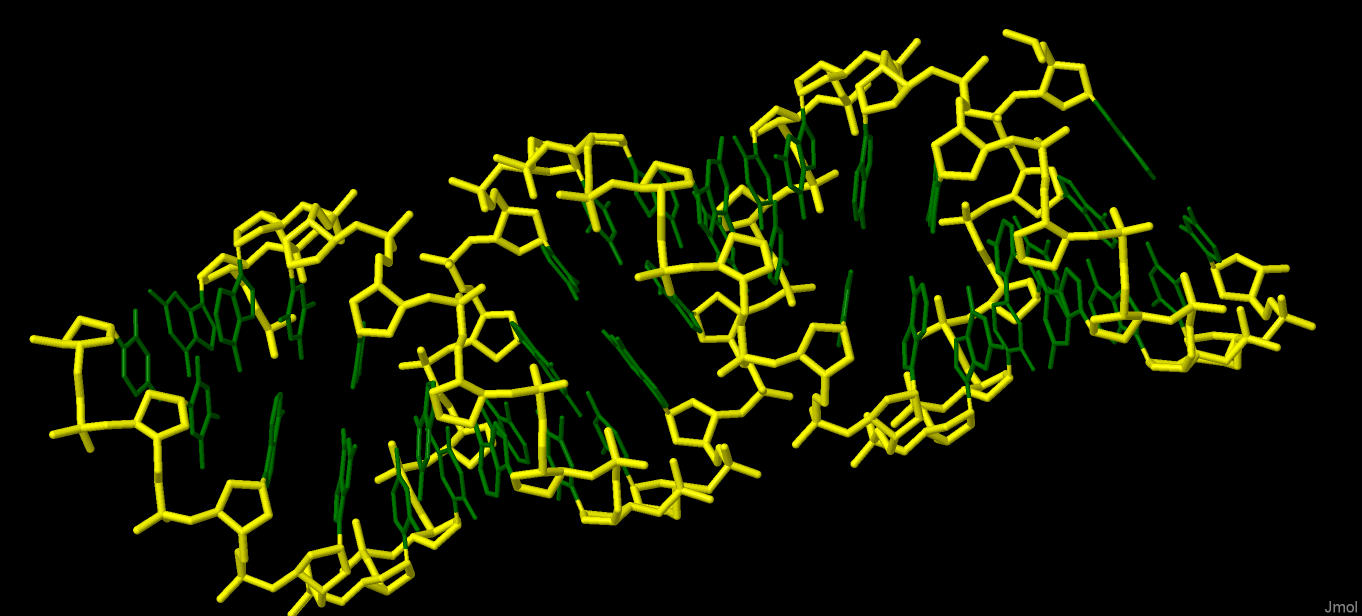
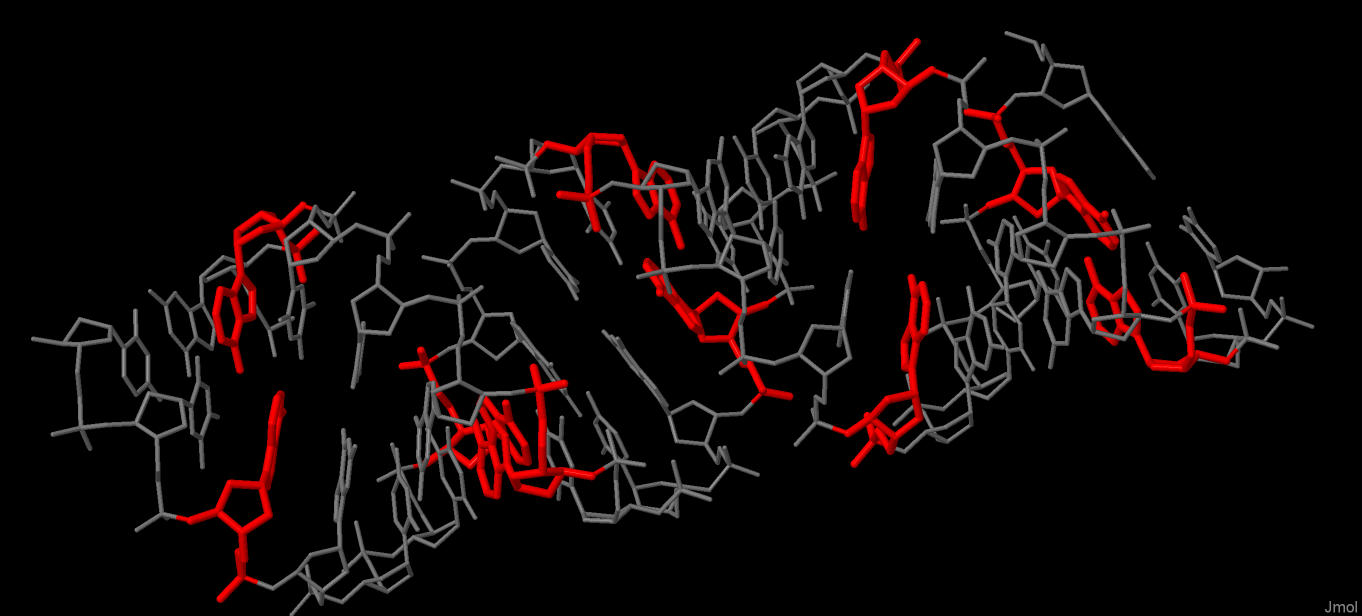
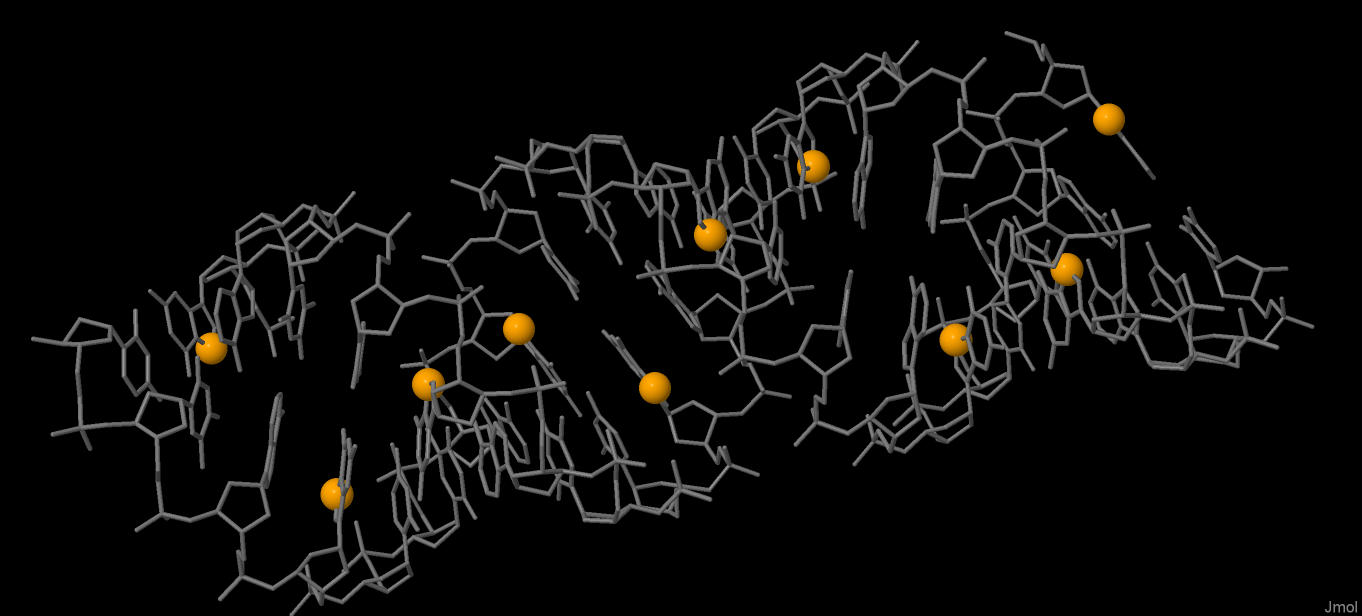
На четырех рисунках ниже - нужные для выполнения задания изображения, полученные в JMol из структуры A-формы. Под каждым - скрипт, воспроизводящий в JMol изображенную структуру.
|
Рис. 1. Желтым выделен сахарофосфатный остов |
Рис. 2. Синим выделены нуклеотиды (вся молекула состоит из нуклеотидов) |
|
Рис. 3. Красным выделены аденины |
Рис. 4. Выделен атом N7 в глутаминах - оранжевый шарик |
Затем средствами JMol предлагалось проверить, есть ли разрывы в нуклеиновых кислотах молекул, структуры которых лежат в файлах 1N77.pdb и 1A02.pdb. На рисунках 5 и 6 видно, что разрывов в структурах нет.
|
Рис. 5. ДНК из 1A02.pdb |
Рис. 6. РНК из 1N77.pdb |
Сравнение структур различных форм ДНК
Требовалось посмотреть на различия структуры больших и малых бороздок в зазных формах ДНК на примере заданного азотистого основания - гуанина. На рисунке 7 - структурная формула гуанина, рассмотренная с точки зрения его положения в бороздчатой структуре B-формы ДНК. В таблице 1 - подобные данные и относительно гуанина в других формах ДНК.
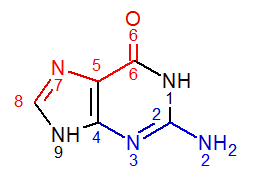
Рис. 7. Гуанин в B-форме ДНК. Красным - атомы, обращенные в сторону большой бороздки, синим - малой. Проект ChemSketch
Таблица 1. Атомы гуанина относительно бороздок ДНК
Форма |
В сторону большой бороздки |
В сторону малой бороздки |
Остальные |
A |
[G]29:B.C5; [G]29:B.C6; [G]29:B.C8; [G]29:B.N7; [G]29:B.O6 |
[G]29:B.C2; [G]29:B.N3; [G]29:B.C4; [G]29:B.N2 |
[G]29:B.N1; [G]29:B.N9 |
B |
[G]29:B.C5; [G]29:B.C6; [G]29:B.C8; [G]29:B.N7; [G]29:B.O6 |
[G]29:B.C2; [G]29:B.C4; [G]29:B.N3; [G]29:B.N2 |
[G]29:B.N1; [G]29:B.N9 |
Z |
[G]29:B.C5; [G]29:B.C6; [G]29:B.C8; [G]29:B.N7; [G]29:B.O6 |
[G]29:B.C2; [G]29:B.C4; [G]29:B.N3; [G]29:B.N2 |
[G]29:B.N1; [G]29:B.N9 |
Далее предлагалось сравнить основные спиральные параметры разных форм ДНК. Результат в таблице 2.
Таблица 2. Сравнение спиралей ДНК
A-форма |
В-форма |
Z-форма |
|
Тип |
правая |
правая |
левая |
Шаг |
28.03 |
33.75 |
43.50 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
16.81 ([T]7:A.P-[C]36:B.P) |
17.21 ([A]6:A.P-[C]32:B.P) |
18.30 ([C]6:A.P-[C]32:B.P) |
Ширина малой бороздки |
7.98 ([C]8:A.P-[T]27:B.P) |
13.10 ([A]14:A.P-[A]30:B.P) |
7.20 ([G]15:A.P-[G]29:B.P) |
Кроме того, нужно было измерить для гуанина торсионные углы. Результаты в таблице 3.
Таблица 3. Торсионные углы гуанина
α (P-O5') |
β (O5'-C5') |
γ (C5'-C4') |
δ (C4'-C3') |
ε (C3'-O3') |
ζ (O3'-P) |
χ (C1'-N) |
|
A-форма |
-51.7 |
174.8 |
41.7 |
79.1 |
-147.8 |
-75.1 |
-157.2 |
В-форма |
-29.9 |
136.3 |
31.2 |
143.3 |
-140.8 |
-160.5 |
-98 |
Определение параметров структур нуклеиновых кислот c помощью 3DNA
Для работы с пакетом 3DNA файлы .pdb были переведены в старый формат:
|
remediator --old ''1N77.pdb > ''1N77_old.pdb remediator --old ''1A02.pdb > ''1A02_old.pdb |
Далее были получены файлы с данными об анализе структур, которые будут приведены далее:
|
export PATH=${PATH}:/Z/x3dna-v2.3/bin/ export X3DNA=/Z/x3dna-v2.3 find_pair -t 1N77_old.pdb stdout | analyze find_pair -t 1A02_old.pdb stdout | analyze ex_str -2 stacking.pdb step2.pdb stack2img -cdolt step2.pdb step2.ps |
Как требовалось, с помощью Excel были найдены средние значения торсионных углов для ДНК и РНК, значения на рисунке 8.
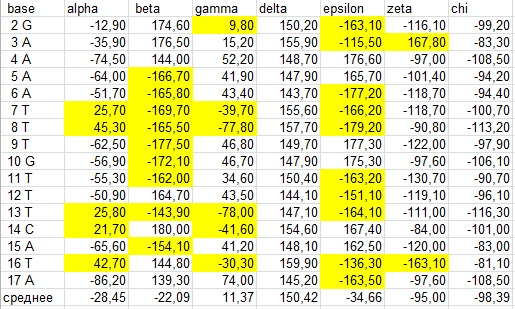
Рис. 8. Торсионные углы в ДНК. Снизу среднее, желтым отмеены сильно отклоняющиеся от среднего значения.
Затем была рассмотрена информация о водородных связях в структуре РНК.
Акцепторный стебель: 501-507 и 572-566
Т-стебель: 549-553 и 565-561
Антикодоновый стебель: 538-544 и 532-526
D-стебель: 510-513 и 525-522
Пары, дополнительно стабилизирующие структуру: 554/558, 555/518, 514/508, 515/548, 519/556
Неканонические пары: G528-A537, U533-C534, G526-A544, G522-U513, G502-U571
На последнем шаге (две последние команды) была составлена картинка стекинг-взаимодействия двух пар с наибольшей площадью перекрывания - GC/GU, площадь 13,40 (рисунок 9).
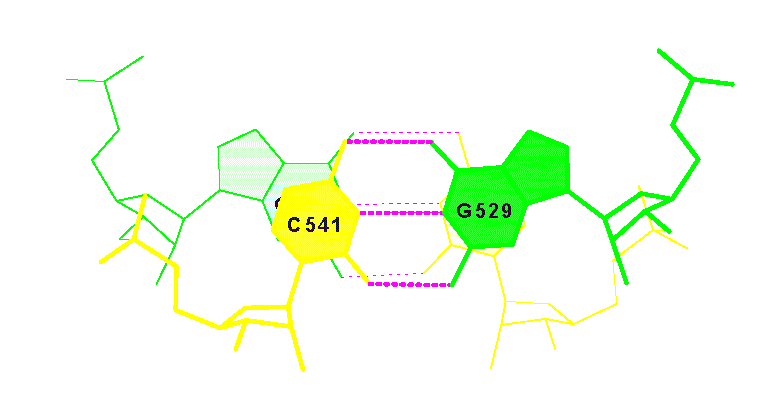
Рис. 9. Стекинг-взаимодействие
© Дарья Горбачева | изменено 14.03.2016 |