| Главная страница | Семестры | О себе | Ссылки |
Определение таксономии и функций нуклеотидной последовательности
Это задание уже было выложено ранее в практикуме "Чтение последовательностей по Сэнгеру".
Сравнение списков находок нуклеотидной последовательности разными алгоритмами blast
Для работы использовалась полученная ранее последовательность, полученная на ББС. Цель: проанализировать ее с помощью трех алгоритмов нуклеотидного BLAST'a NCBI: blastn, megablast, discontiguous megablast.
Область поиска была ограничена типом Echinozoa. Максимальное число находок было установлено на 1000. Остальные параметры оставила дефолтными. Cравнение трех алгоритмов в таблице 1.
Таблица 1. Сравнение выдачи трех алгоритмов нуклеотидного BLAST'a
Алгоритм |
Blastn |
Megablast |
Discontiguous megablast |
Число находок |
48 |
15 |
39 |
E-value худшей находки |
9,4 |
1e-72 |
8e-39 |
Сходство худшей находки |
95% |
81% |
76% |
Также результаты работы BLAST'a представлены на рисунках 1-3.
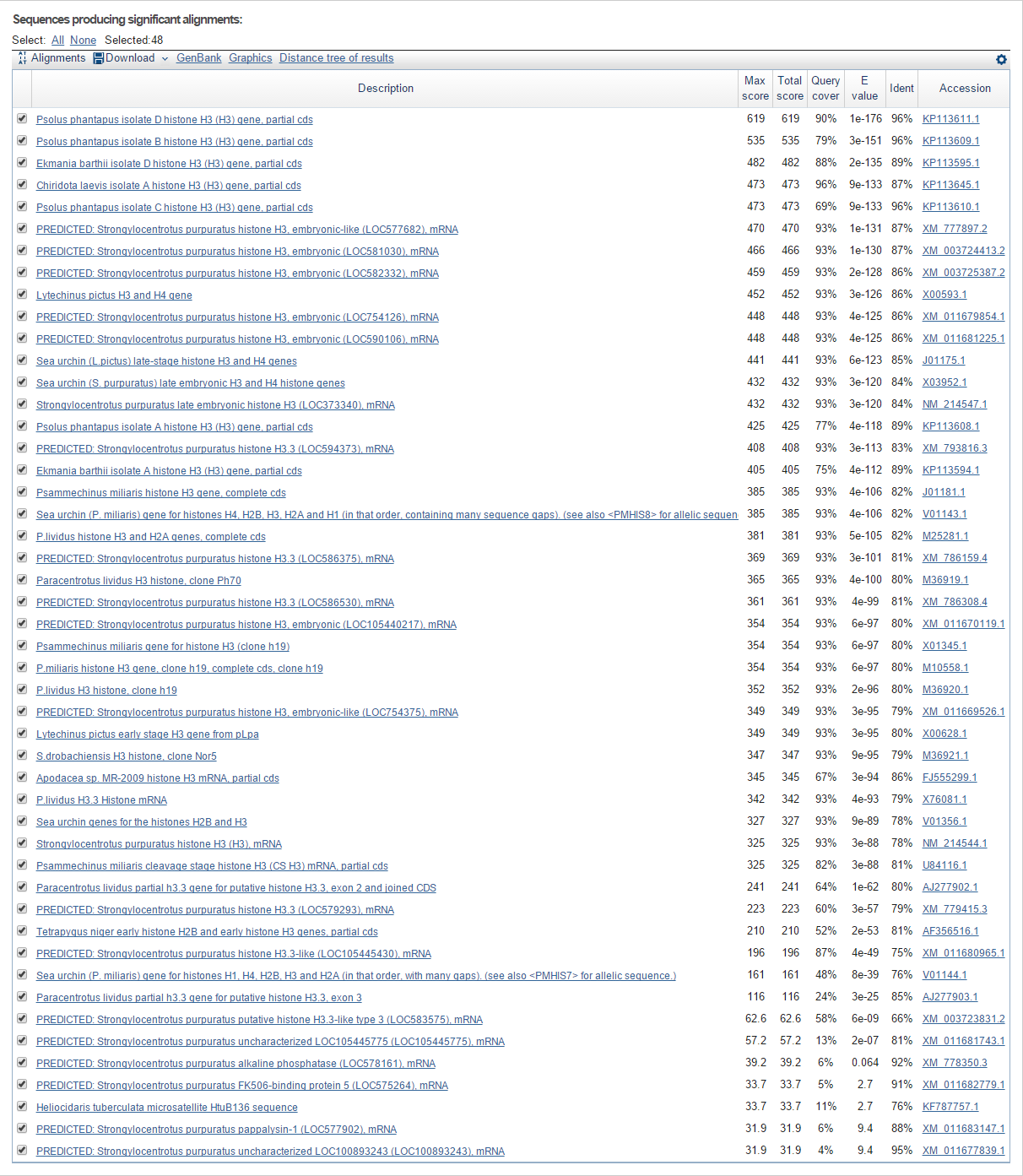
Рис. 1. Список находок blastn
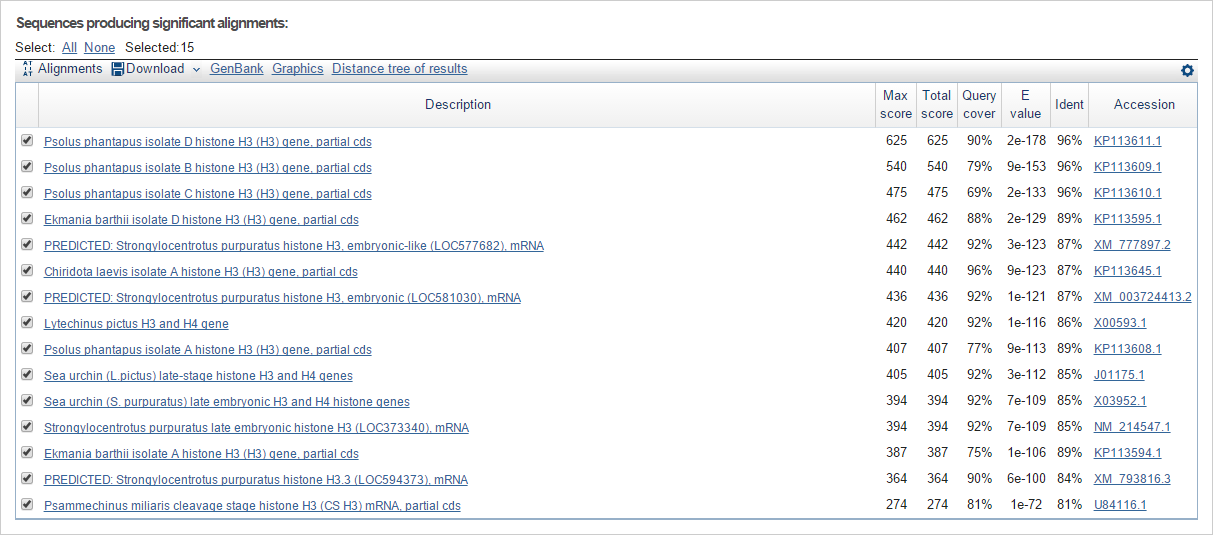
Рис. 2. Список находок megablast
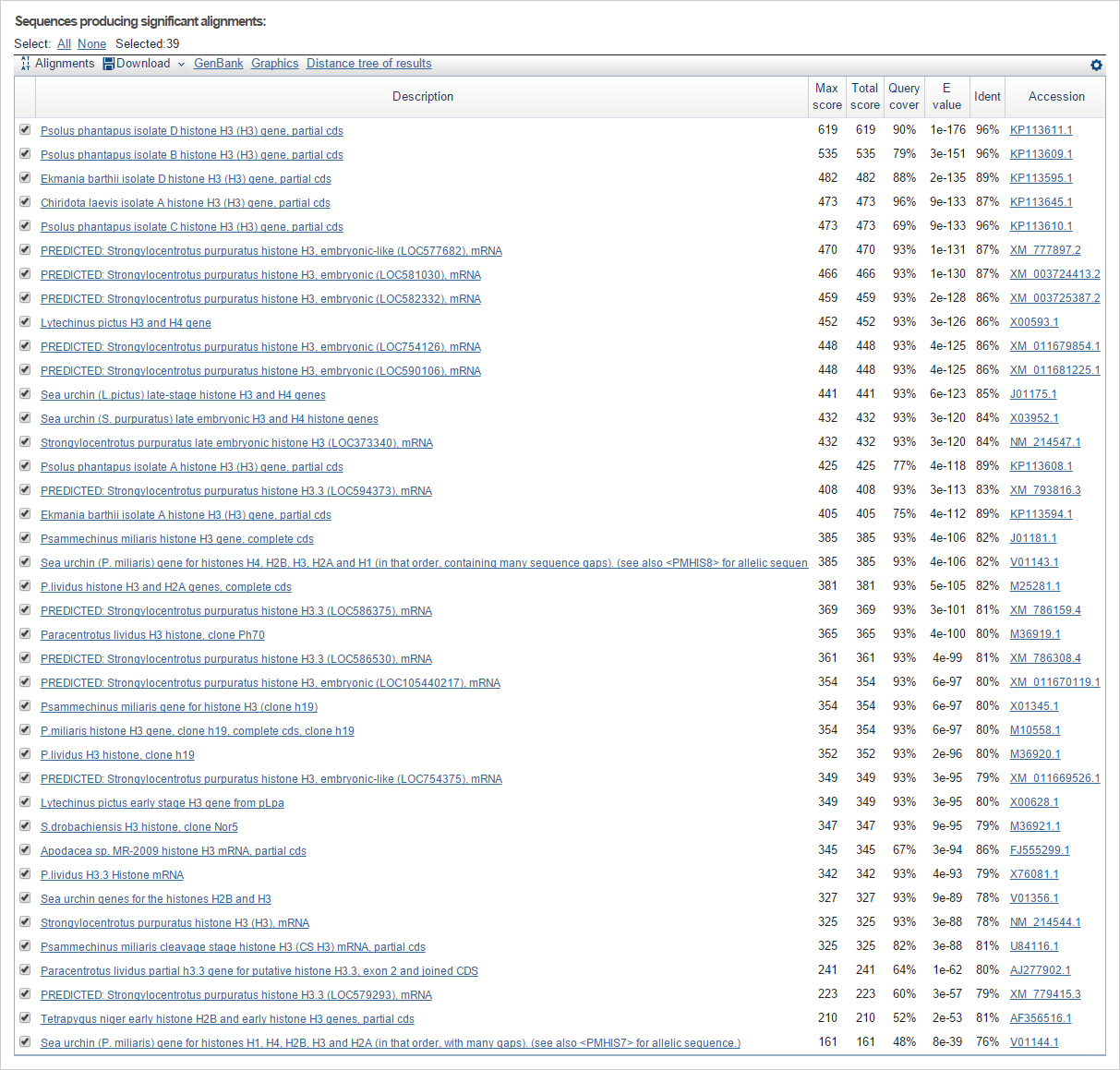
Рис. 3. Список находок discontiguous megablast
Из результатов работы алгоритмов видно, что в выдаче blastn в сравнении с двумя другими алгоритмами есть находки с низкими query cover и очень высокими высокими E-value. Это логично, так как blastn был запрограммирован на поиск любого сходства, даже и небольших участков последовательностей. Что касается других алгоритмов, то megablast расчитан на поиск возможных близких гомологов последовательности-запроса, discontiguous megablast менее требователен и выявляет возможных гомологов, которые достаточно сильно дивергировали. Поэтому megablast и выдал наименьшее количество находок.
Проверка наличия гомологов белков в геноме Amoboaphelidium
Цель: проверить наличие гомологов пяти белков в организме Amoboaphelidium (сборка генома) с помощью алгоритма tblastn. Белки, выбранные мной для работы, перечислены в таблице 2.
Таблица 2. Белки для поиска гомологов в заданном геноме
Название белка |
Запись в UniProt |
Функция |
Белок контроля деления клеток 6 мыши |
Участвует в инициации репликации |
|
β-цепь тубулина человека |
Основной структурный компонент микротрубочек |
|
Белок-цистеин метилтрансфераза человека |
Участвует в репарации метилированного гуанина |
|
Нибрин человека |
Компонент MRN-комплекса, участвующего в рекомбинации, репарации двухцепочечных разрывов ДНК, контроле клеточного цикла |
|
Фактор репликации MCM7 человека |
Компонент основной репликативной хеликазы |
Для удобства работы я объединила все последовательности белков в файл.
Создание банка данных из последовательности генома Amoboaphelidium:
makeblastdb -in X5.fasta -dbtype nucl |
Запуск tblastn:
tblastn -query proteins.fasta -db X5.fasta -out proteins.out -outfmt 7 |
Результат: таблица с разнообразными находками. Как и ожидалось, сходства нашлись для каждого из пяти выбранных белков, притом хорошие (E-value < 0,1). Более подробно результаты работы алгоритма описаны в таблице 3.
Таблица 3. Результаты поиска гомологов в геноме Amoboaphelidium
Название белка |
Число находок (все/хорошие) |
Параметры лучшей находки |
|||
Запись |
Идентичность |
Query cover |
E-value |
||
CDC6_MOUSE |
6/5 |
scaffold-693 |
31,94% |
57,30% |
3e-41 |
TBB5_HUMAN |
13/12 |
unplaced-665 |
84,86% |
96,17% |
0,0 |
MGMT_HUMAN |
3/2 |
scaffold-444 |
45,95% |
17,39% |
0,023 |
NBN_HUMAN |
6/2 |
scaffold-105 |
25,51% |
30,37% |
4e-10 |
MCM7_HUMAN |
15/14 |
scaffold-17 |
46,88% |
91,38% |
2e-179 |
Как видно из таблиы 3, наиболее вероятные гомологи нашлись для самых "популярных" белков из списка - для тубулина и хеликазы. Эти два результата можно считать достоверными. Худший результат был получен для метилтрансферазы. Возможно, найденное сходство даже является случайным и ДНК-метилтрансфераза Amoboaphelidium не похожа на человеческую.
Классификация геномов родственных вирусов по сходству последовательностей
Из базы данных NCBI были получены геномы пяти вирусов рода Potexvirus:
Lily virus X
Nerine virus X
Alstroemeria virus X
Phaius virus X
Allium virus X
Последовательности геномов в указанном порядке объединены в файл.
Из файла с последовательностями была создана база данных:
makeblastdb -in virus.fasta -dbtype nucl |
Далее с помощью следующей команды было построено выравнивание:
tblastx -query virus.fasta -db virus.fasta -out virus.out -evalue 0.001 -outfmt 7 |
C помощью предоставленного нам питоносвского скрипта выравнивание было очищено от неинформативных результатов:
python revise_blast_7.py -i virus.out -s 25 -l 100 -e 0.001 -o virus.xls |
Результат: таблица.
Для оценки сходства вирусов я сранжировала информацию о выравниваниях (строки) по query_id и смотрела, между какими записями наибольшее сходство по identity и aln_len. Таким образом понятно, например, что большое сходство наблюдатся между Lily virus X и Phaius virus X, Alstroemeria virus X и Nerine virus X, Allium virus X ближе всего к Phaius virus X.
© Дарья Горбачева | изменено 28.02.2016 |

