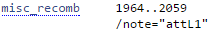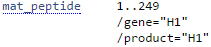| Главная страница | Семестры | О себе | Ссылки |
Качество сборки генома C. elegans
 |
Рис. 1. Микрофотография Caenorhabditis elegans |
В качестве организма, информацию о сборках генома которого я буду представлять, была выбрана нематода Caenorhabditis elegans. Это популярный модельный организм для изучения различных молекулярно-биологических процессов (например, РНК-интерференции), а также биологии развития, генетики и т.д. Поэтому я предположила, что это животное будет очень хорошо изучено, а геном будет отсеквенирован полностью, притом возможно, что не единожды.
Осуществив поиск по названию организма в базе данных Genomes NCBI, я выяснила:
Для организма есть две сборки генома (GCA_000002985.3 и GCA_000975215.1).
Эти сборки соответствуют двум крупным проетам по полногеномному секвенированию и двум образцам. Интересно, что более мелких проектов (по транскриптому, неполногеномному секвенированию) целых 594 на момент написания мной этого отчета.
Представляю информацию о сборке GCA_000975215.1.
Описание образца
Идентификатор: BioSamples - 3334911
Организм: Caenorhabditis elegans Hawaiian Strain CB4856
Опиcание: штамм СВ4856 вышеназванной нематоды был получен в 1972 году на острове Мауи. Этот штамм сложен в изучении значительно отличается от более популярного N2 - например, наличием сфрагиса, тенденциями к социальному поведению (аггрегация), менее сильной корреляцией между температурой среды и размером тела и т.д.
Образец использован в проекте: PRJNA275000
Предоставлен: Университетом Вашингтона 2 сентября 2015.
Описание проекта
Идентификатор: 275000
Тип: секвенирование и сборка генома (получены последовательности 5 аутосом, половой хромосомы, митохондриальной плазмиды).
Публикация на основе проекта: Remarkably Divergent Regions Punctuate the Genome Assembly of the Caenorhabditis elegans Hawaiian Strain CB4856.
Таксономия объекта изучения: Eukaryota; Metazoa; Ecdysozoa; Nematoda; Chromadorea; Rhabditida; Rhabditoidea; Rhabditidae; Peloderinae; Caenorhabditis; Caenorhabditis elegans
Технология секвенирования: Illumina.
Предоставлен: Университетом Вашингтона 21 апреля 2015.
Число контигов: 16 (на странице проекта указано число 17, но в таблице с информацией о контигах 16 позиций).
Число скэффолдов: 15 (по одному на каждую хромосому и 9 неустановленного положения).
Таблица контигов/скэффолдов (формат .txt): *ссылка*
N50: 14890789 п.о.
L50: 3
Самый длинный контиг: 20182852 п.о.
Самый короткий контиг: 607 п.о.
Пример последовательности контига: *ссылка*
Митохондриальные гены мха
 |
Рис. 2. Мох Anomodon rugelii |
Для исследования мне достался мох Anomodon rugelii из семейства Anomodontaceae. С помощью запроса Anomodon rugelii[Organism] AND Mitochondrion[Filter] в базе данных Nucleotide NCBI я нашла два варианта последовательности. Не думаю, что они сильно различаются: выложены с разницей в три дня одними и теми же авторами по одной и той же публикации. Я выбрала первую, поскольку она из RefSeq (базы данных проверенных геномов). Далее я перешла по ссылке Genome в колонке Reference Information, это перевело меня на страницу с таблицей о митохондриальных генах (рис. 3).
 |
Рис. 3. Данные о митохондриальном геноме Anomodon rugelii |
Данные по таблице:
Общее число генов: 76
Число генов РНК: 27
Число генов белков: 46
Число псевдогенов: 3
Дальше я прошла по ссылке Gene в Related Information и попала на страницу с информацией о генах. С помощью опции Sort by Chromosome я сохранила ее так, что гены располагаются в порядке следования по цепи.
Ключи, используемые в таблицах особенностей
Ключи были найены на сайте INCDC по самой нижней ссылке на странице (ссылка ведет сюда, нужная информация в разделе Appendix II). Примеры кликабельны.
№ |
Ключ |
Описание |
Пример использования |
1 |
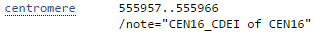
centromere |
Область, о которой экспериментально была доказана принаждежность к центромере - участку ДНК, где связаны сестринские хроматиды и находится кинетохор |
|
2 |
intron |
Участок ДНК, который хоть и транскрибируется, но впоследствии вырезается из транскрипта |
|
3 |
sig_peptide |
Последовательность, кодирующая N-концевой сигнальный (то есть реагирующий с компонентами мембраны) домен секретируемого белка |
|
4 |
regulatory |
Любой участок последовательности, который участвует в регуляции транскрипции или трансляции |
|
5 |
polyA_site |
Участок, кодирующий те места РНК, куда добавятся остатки аденина вследствие посттранскрипционного полиаденилирования |
|
6 |
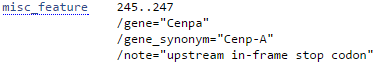
misc_feature |
Необычный участок, не попадающий под описание никаким иным ключом |
|
7 |
misc_recomb |
Участок, на котором часто происходит рекомбинация - разрыв и сшивка ДНК-дуплекса |
|
8 |
stem_loop |
Шпилька - элемент вторичной структуры РНК, образующийся засчет комплиментарных взаимодействий между основаниями в одноцепочечной молекуле |
|
9 |
mat_peptide |
Последовательность, кодирующая финальный пептид или белок (после пост-транскрипционных модификаций); не включает в себя стоп-кодон |
|
10 |
old_sequence |
Представленная последовательность повторяет предыдущий вариант последовательности в этом месте |
BLASTN
Выложено на странице предыдущего практикума "Чтение последовательностей по Сэнгеру".
© Дарья Горбачева | изменено 16.09.2015 |