| Главная страница | Семестры | О себе | Ссылки |
Сравнение составов систем рестрикции-модификации
В задании требовалось сравнить состав систем рестрикции-модификации, закодированных в двух штаммах одного вида.
Сначала нужно быо найти избегаемые сайты рестрикции в геноме выданной бактерии Bacteroides salanitronis (CP002530.1) и в наборе контигов из метагенома кишечника человека. C помощью веб-сервиса и файла со списком всех известных сайтов систем рестрикции-модификации типа II sites.list было посчитано ожидаемое количество и контраст сайтов из файла в геноме бактерии и в метагеноме кишечника (по методу Карлина).
Файлы был профильтрован по пороговому значению контраста 0.78:
Отфильтрованный результат с веб-сервиса для бактерии (16 избегаемых сайтов)
Отфильтрованный результат с веб-сервиса для метагенома человека (21 избегаемый сайт)
Я не предполагала различия в избегаемых сайтах из-за сходных сред обитания бактерий из полного генома и из контигов (соответственно, слпая кишка курицы и кишечник человека). Однако различия оказались достаточно большими (ниже). Возможно, вирусы, с коровыми борятся бактерии при помощи ситрем рестрикции-модификации, достаточно сильно отличаются в курице и в человеке.
Общие сайты избегания: CATATG, CTAG, CTCGAG, CTTAAG, GAGCTC, GATC, GGATCC, GGCC, GTCGAC, TCGCGA
Только для полного генома: CCTAGG, CCWGG, GGNCC, GRGCYC, GTATAC, GTGCAC
Только для контигов: AGTACT, ATGCAT, CCGCGG, CGATCG, CTGCAG, GACGTC, GATATC, GCATGC, GCGCGC, GGCGCC, GGTACC
Поиск последовательностей Шайн-Дальгарно в геноме Streptococcus pneumoniae R6
Последовательности Шайн-Дальгарно - сайты инициации трансляции на мРНК прокариот. Поиск по базе данных PubMed ("Shine-Dalgarno Streptococcus pneumoniae") выдал 10 статей. В них не упоминались отклонения от обычных характеристик последовательностей Шайн-Дальгарно. Упоминалось, что они в подавляющем большинстве случаев необходимы для инициации трансляции [1], была указана такая последовательность для гена recP - AGAAAGGA [2]. Такой мотив, наряду с обычным Шайн-Дальгарно, также будет найден (рисунок 2).
Для своей бактерии я скачала файл с хромосомой и особенности. Особенности были модиицированы с помощью скрипта - результат. Далее с помощью простейшего скрипта из результата были отобраны CDS длиннее 300 пар нуклеотидов, не гипотетические, удалены повторы - таких подходящих последовательностей оказалось 876.
Для создания файла с областями поиска с помощью скрипта файл с выбранными CDS был отформатирован по указанному образцу. Затем по нему был запущен скрипт - результат файл с областями поиска (вручную удалена первая последовательность и одного н.к.). Как видно из моего скрипта, бралось по 17 нуклеотидов в апстримной области.
По тому же механизму были извлечены области поиска для всех генов. Но длина апстримной области была увеличена до 25 нуклеотидов.
На рисунке 1 - параметры работы, заданной MEME.

Рис. 1. Параметры поиска мотивов в апстримных областях генов

Рис. 2. Найденные мотивы
На рисунке 2 мы видим основной результат работы программы. Первый найденный мотив отличается от двух других значительно меньшим e value и значительно большей встречаемостью. Это и есть универсальная прокариотическая последовательность Шайн-Дальгарно - AAGGAG.
Для получения PWM я запустила MEME с прицелом на только один мотив (поскольку результат был уже предсказуем). PWM (позиционная матрица весов) была найдена в текстовой выдаче MEME:
--------------------------------------------------------------------------------
Motif 1 position-specific probability matrix
--------------------------------------------------------------------------------
letter-probability matrix: alength= 4 w= 8 nsites= 541 E= 3.1e-440
0.652495 0.059150 0.090573 0.197782
0.800370 0.020333 0.083179 0.096118
0.194085 0.000000 0.805915 0.000000
0.000000 0.000000 1.000000 0.000000
0.955638 0.000000 0.044362 0.000000
0.231054 0.000000 0.768946 0.000000
0.434381 0.029575 0.388170 0.147874
0.573013 0.070240 0.090573 0.266174
Для работы с апстримными областями всех генов из генома мотив Шайн-Дальгарно был передан сервису FIMO (правая стрелка около мотива в выдаче MEME).
Количество нахождений мотива варировалось в зависимости от задания порога p value. Выдачи были сохранены для разных p value.
Финальный результат сохранен в таблице.

Рис. 3. Лого мотива Шайн-Дальгарно
Определение сайтов связывания транскрипционного фактора в данном участке хромосомы человека
Есть файл chipseq_y14/chipseq_chunk40.fastq с ридами Illumina, полученными в результате Chip-Seq эксперимента. С помощью программы FastQC на kodomo был осуществлен контроль качества прочтений:
fastqc chipseq_chunk40.fastq |
На рисунке 5 отображение качества чтений из html-выдачи FastQC. Видно, что качество чтений очень хорошее (всё в зеленой области), поэтому чистка программой Trimmomatic на требуется. Изначально чтений 6005.
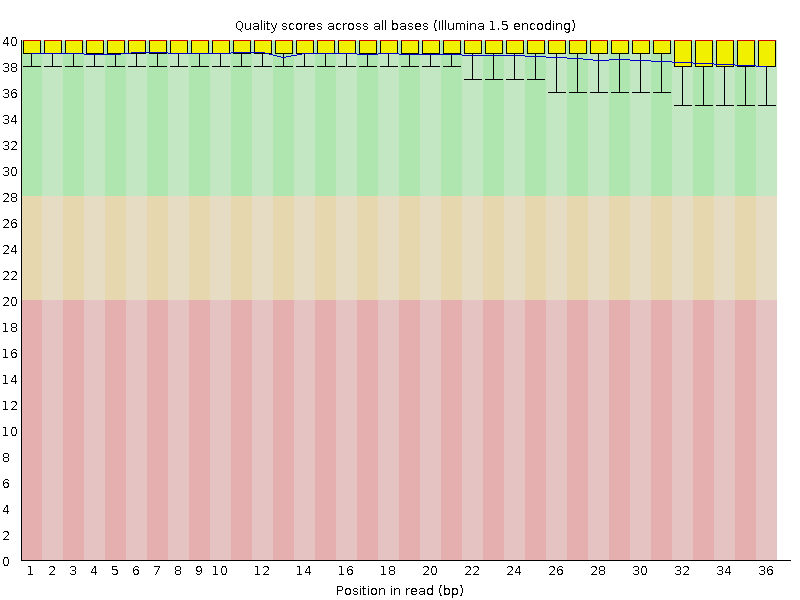
Рис. 5. График качества чтений
Следующий шаг - картирование на геном человека hg19:
bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk40.fastq > chipseq_chunk40.sam |
Перевод выравнивания чтений с референсным геномом в бинарный формат для дальнейшей обработки:
samtools view -bSo chipseq_chunk40.bam chipseq_chunk40.sam |
Сортировка выравнивания по координате начала чтения в референсе:
samtools sort chipseq_chunk40.bam -T chip_temp -o chipseq_chunk40.sorted.bam |
Индексирование отсортированного файла:
samtools index chipseq_chunk40.sorted.bam |
Запись в файл chipseq_chunk40.idxstats информации о количестве чтений, откартированных на каждый элемент генома:
samtools idxstats chipseq_chunk40.sorted.bam > chipseq_chunk40.idxstats |
Выведение информации о числе чтений, откартировавшихся на все элементы генома:
samtools view -c chipseq_chunk40.sorted.bam |
Комманда выведа число 6005. То есть откартировались все риды. При этом распределение чтений по хромосомам (рисунок 6) позволяет предположить, что я работаю с участками 9 хромосомы человека.
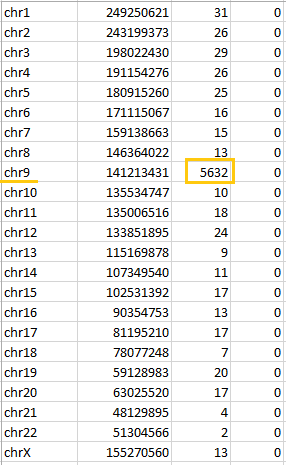
Рис. 6. Распределение числа откартированных чтений по хромосомам. Больше всего чтений откартировано на 9 хромосому (5632 из 6005, что составляет 94%)
Для поиска пиков (peak calling) была попытка использовать программу MACS таким образом:
macs2 callpeak -t chipseq_chunk40.sorted.bam |
Но это не сработало из-за слишком небольшого числа пиков (0). Поэтому была использована команда:
macs2 callpeak -t chipseq_chunk40.sorted.bam -n chipseq_chunk40 --nomodel |
Программа выдала три файла:
Всего найдено 6 пиков, информация о них в таблице на рисунке 7. В принципе, все они примерно равны по достоверности. Пики 2 и 4 чуть лучше, поскольку имеют наибольшие отрицательные логарифмы p и q value.
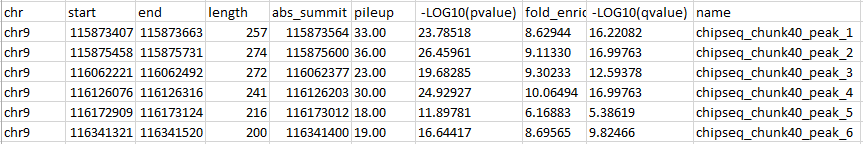
Рис. 7. Таблица с характеристиками пиков
Для последующей визуализации пиков в начало файла _peaks.narrowPeak была помещена дополнительная информация:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" browser position chr9:115873407-116341520 |
Далее пики были визуализированы при помощи инструмента UCSC Genome Browser - результат на рисунке 8.
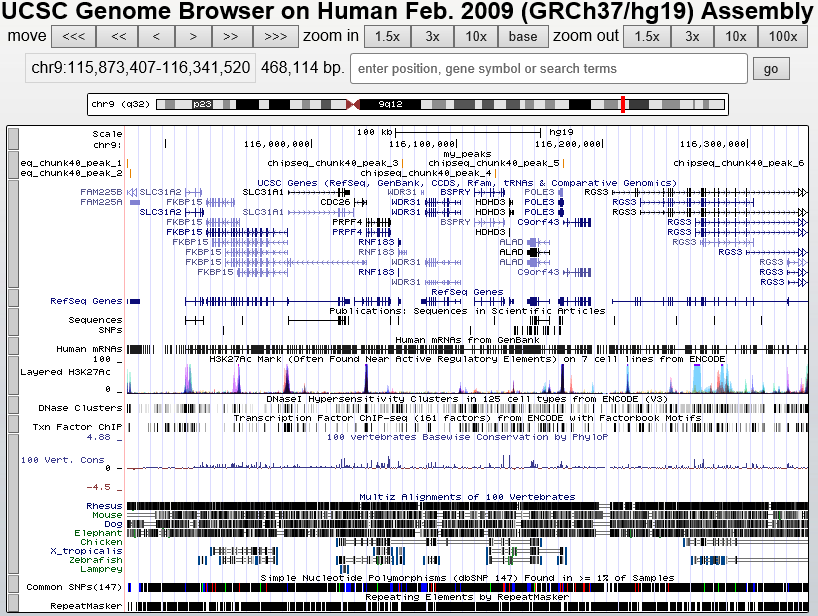
Рис. 8. Расположение найденных пиков в геноме человека (сборка hg19)
Рассмотрим пик 2 (на рисунке 9). Он попал на интрон гена FAM225A (не белок-кодирующий). Также в этом месте нашли сайт связывания фактора в проведенном ранее ChIP-seq эксперименте. Есть метка H3K27Ac, обычно находящаяся близ активных регуляторных элементов. Похоже на настоящий сайт транскрипционного фактора.
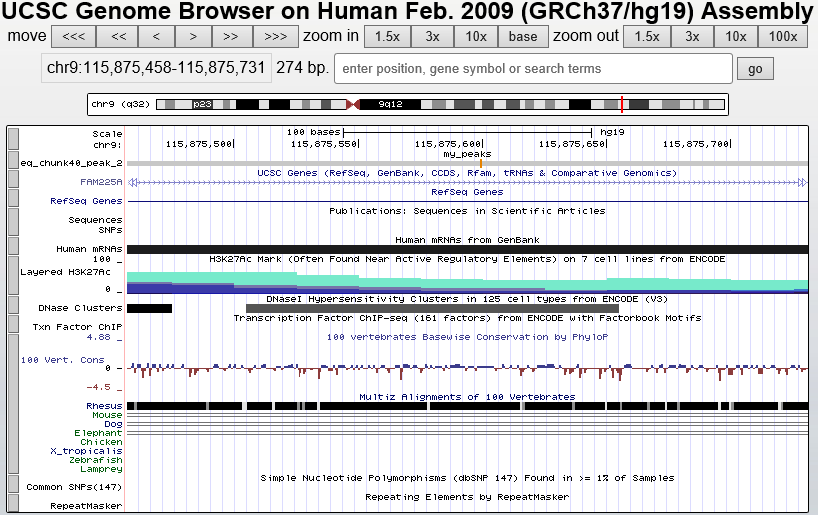
Рис. 9. Расположение пика 2
Рассмотрим пик 4 (на рисунке 10). Тут тоже есть информация о метке и о ранее найденных сайтах связывания. Пик попал в интрон BSPRY - белок-кодирующий ген, возможно контролирующий эпителиальный транспорт кальция.
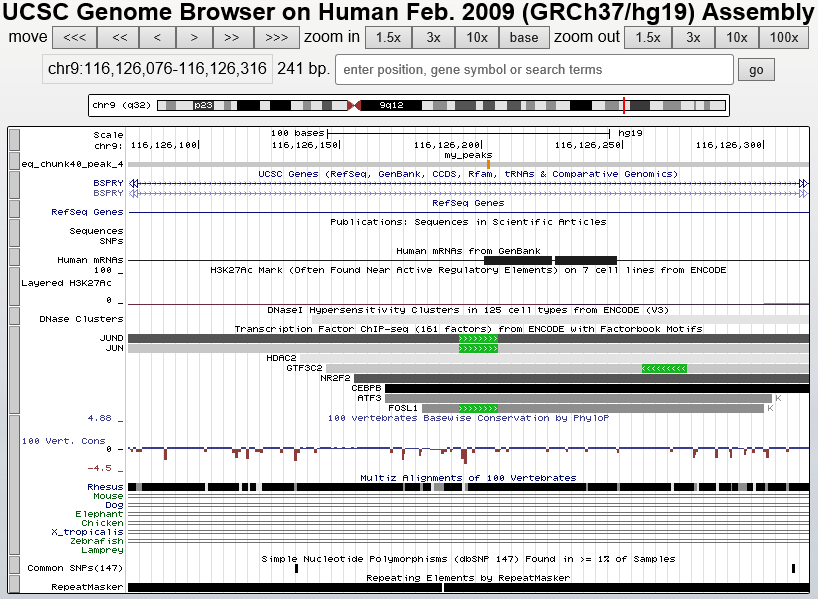
Рис. 10. Расположение пика 4
Поиск сигналов TATA-бокс связывающего белка (TBP) в геноме человека
В данном задании требовалось проанализировать результаты эксперимента ChIP-seq по поиску TATA-box - сайтов связывания белка TBP. Это связывание необходимо и достаточно для инициации транскрипции у эукариот. ТАТА-бокс имеет консенсус T-A-T-A-A/T-A/G.
На странице chi-seq Experiment Matrix был выбран эксперимент на клеточной линии H1-hESC с антителами кролика.
Содержащие ТАТА-box промоторы:
Название: MX1 (Homo sapiens myxovirus (influenza virus) resistance 1)
Расположение: chr21:42,803,998-42,830,685 +
Длина гена: 26,688
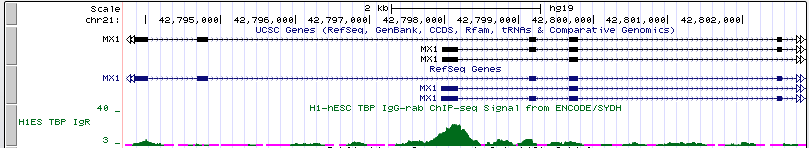
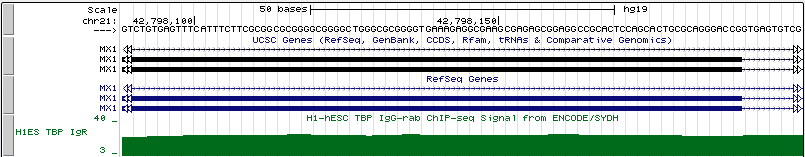
Рис. 11. Промоторная область гена
Название: JAM2 (Homo sapiens junctional adhesion molecule 2)
Расположение: chr21:27011594-27086885 +
Длина гена: 75,293
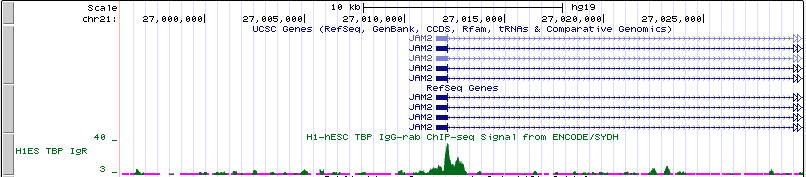
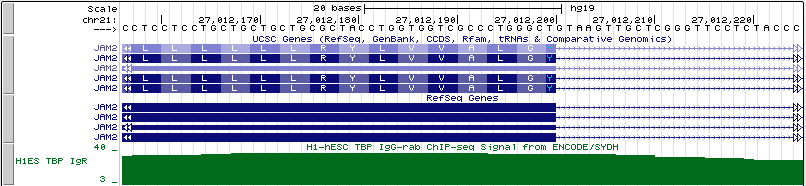
Рис. 12. Промоторная область гена
Название: MRPL39 (Homo sapiens mitochondrial ribosomal protein L39)
Расположение: chr21:26957968-26979801 -
Длина гена: 21,834
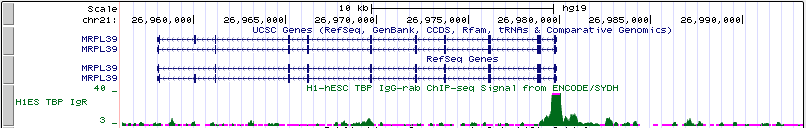
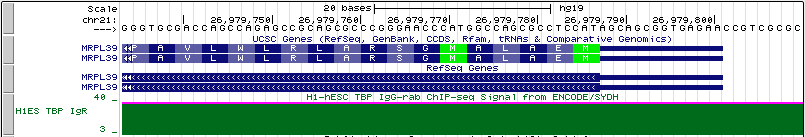
Рис. 13. Промоторная область гена
Не содержащий TATA-box промотер:
Название: LOC339622 (Homo sapiens uncharacterized LOC339622, long non-coding RNA)
Расположение: chr21:26212864-26430056 +
Длина гена: 217,193
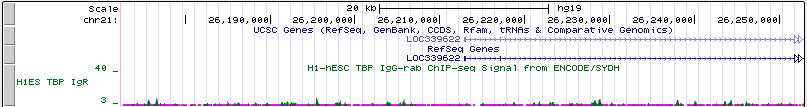
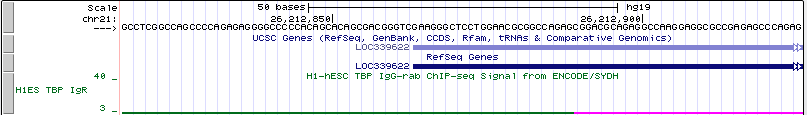
Рис. 14. Промоторная область гена
© Дарья Горбачева | изменено 2.10.2016 |

