| Главная страница | Семестры | О себе | Ссылки |
Построение дерева по нуклеотидным последовательностям
Требовалось построить филогенетическое дерево бактерий, используя последовательности РНК малой субъединицы рибосомы (16S rRNA). Они были получены из базы полных геномов NCBI, в частности из файлов .frn, содержащих все РНК. Файл с последовательностями. Взятые штаммы в таблице 1.
Таблица 1. Организмы, использованные для филогенетического анализа
Название |
Мнемоника |
Штамм |
Bradyrhizobium diazoefficiens |
BRADU |
USDA 110 uid57599 |
Burkholderia cenocepacia |
BURCA |
HI2424 uid5836 |
Ralstonia pickettii |
RALPJ |
12J uid58737/p> |
Pseudomonas aeruginosa |
PSEAE |
PA7 uid58627 |
Enterobacter sp. 638 |
ENT38 |
638 uid58727 |
Escherichia coli |
ECOLI |
SE11 uid5942 |
Yersinia pestis |
YERPE |
Angola uid58485 |
Vibrio fischeri |
VIBFM |
MJ11 uid5890 |
Последовательности были выровнены методом Muscle, по выравниванию в MEGA было построено дерево методом Maximum Likelihood. Оно на рисунке 1.
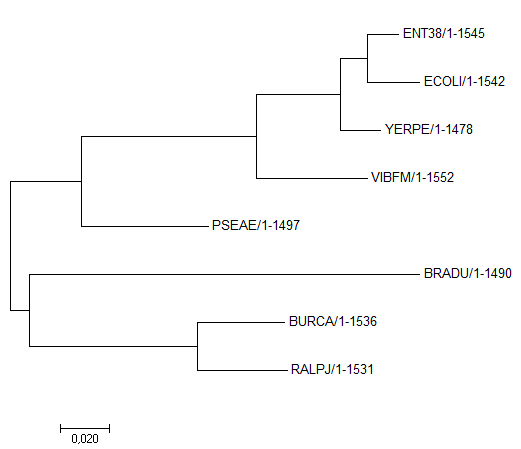
Рис. 1. Дерево на основе 16S rRNA
Дерево получилось идентичным правильному из практикума 1 (однако это не очевидно - деревья по-разному укоренены).
Построение и анализ дерева, содержащего паралоги
Требовалось построить дерево по гомологам белка CLPX_ECOLI.
Чтобы найти гомологов, из директории P:\y14\term4\Proteomes я выбрала протомы своих восьми бактерий, загрузила их себе в директорию и сложила в один файл с помощью команды cat, запустила по нему локальный blast c помощью команд:
makeblastdb -in proteomes.fasta -dbtype prot -out database.fasta |
blastp -query clpx_ecoli.fasta -evalue 0.001 -db database.fasta -outfmt 6 -out findings.fasta |
За исключением самого запроса, blast выдал 33 находки. Они были выровняны c помощью Muscle, по выравниванию в MEGA создано дерево. Оно представлено на рисунке 2.
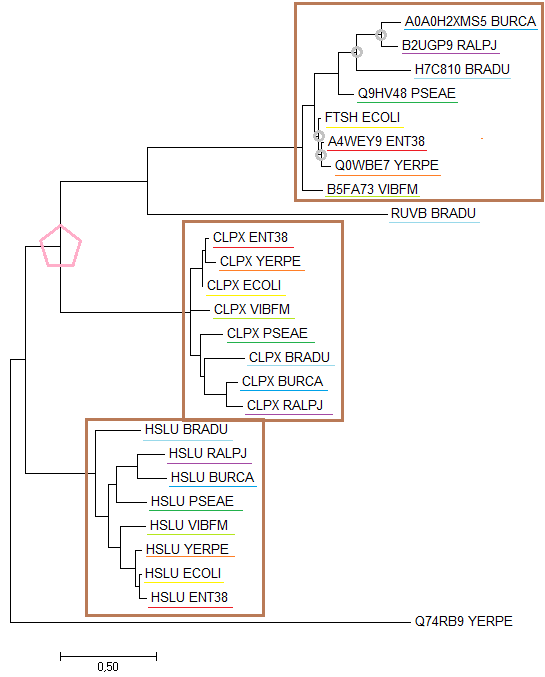
Рис. 2. Дерево из возможных гомологов CLPX_ECOLI
На рисунке 2 введены следующие обозначения:
Коричневые рамки - предполагаемые ортологичные группы
Одним и тем же цветом радуги подчеркнуты возможные паралоги
Серым кружком отмечены примеры разделения путей эволюции белков в результате видообразования
Розовый пятиугольник - пример дупликации гена
© Дарья Горбачева | изменено 11.04.2016 |

