| Главная страница | Семестры | О себе | Ссылки |
Работа с KEGG ORTHOLOGY
Требовалось проверить, являются ли члены разных ортологических рядов KEGG гомологичными белками и проанализировать их филогенетические отношения. Для этого из данных вариантов я выбрала путь биосинтеза лизина (на рисунке 1), а в нем реакцию 1.2.1.31 (отмечена голубым на рисунке 1 и представлена на рисунке 2).
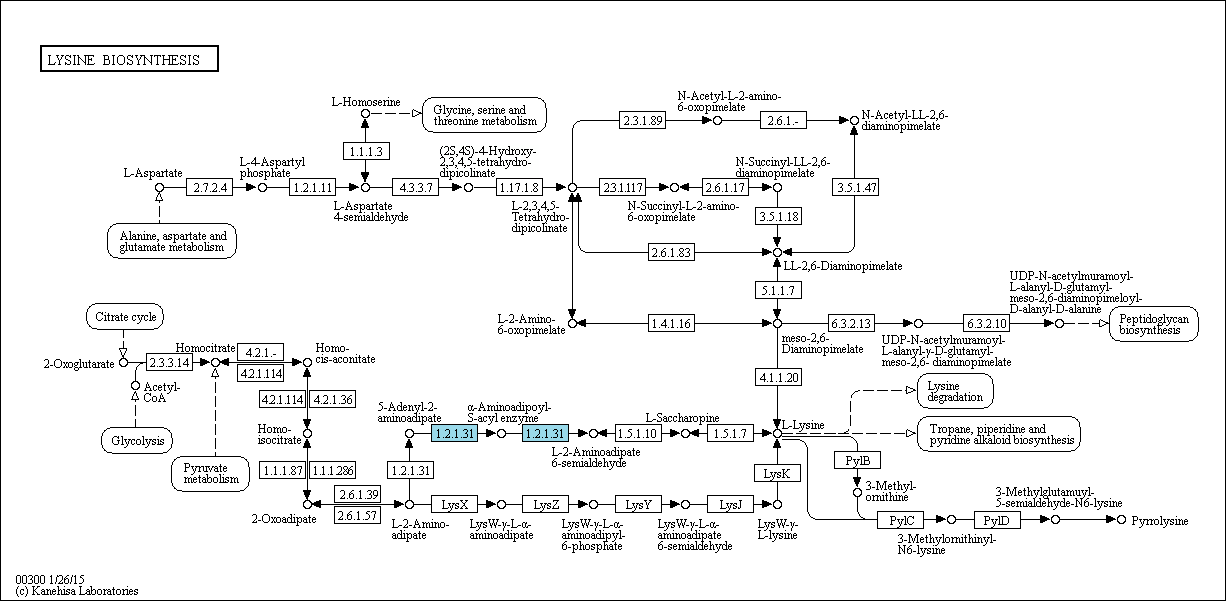
Рис. 1. Путь биосинтеза лизина с отмеченной голубым анализируемой реакцией
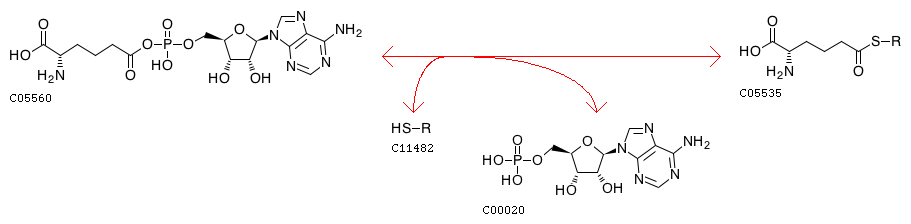
Рис. 2. Катализируемая рассматриваемыми белками реакция
Реакцию катализируют два ортологичных ряда белков (ниже). Из UniProt были получены последовательности этих рядов, к идентификатору каждого белка добавлен идентификатор ортологического ряда.
K00143 (102 белкa из UniProt - последовательности)
K14085 (110 белков из UniProt - последовательности)
Белки были выровнены с помощью Muscle - выравнивание .fasta.
Видно, что белки из одного ряда гомологичны между собой: в выравнивании присутствуют большие блоки с высококонсервативныи позициями. Однако два разных блока мало похожи, хотя наблюдаются некоторые блоки с консервативностью осатков по свойствам. Я думаю, между двумя ортологическими рядами все же есть гомология, но эволюционное расхождение произошло очень давно.
В проекте Jalview помимо исходного выравнивания allalign есть окно allalign_modified с выравниванием, модифицированным как описано ниже.
Из ряда К00143 были удалены:
Плохо выровненные последовательности: C7ZKS8_NECH7, L8GYL8_ACACA, R8BBK6_TOGM
Последовательности с большим количеством гэпов на местах блоков: A8PSDO_MALGO, A9US27_MONBE, C5MF38_CANTT, D4B1F0_ARTBC, D4DCW8_TRNH, E9EM15_METRA, M7SQR7_EUTLA, F8P2A0_SERL9, F0ZCR4_DICPU, C5MF39_CANTT
Из ряда К14085 были удалены:
Плохо выровненные последовательности: A0C8Z9_PARTE, A0DG09_PARTE
Последовательности с большим количеством гэпов на местах блоков: A0A061GLH2_THECC, B6TPU3_MAIZE, B9TE04_RICCO, R7SL24_DICSQ, B9T896_RICCO
По модифицированному выравниванию было построено дерево методом Neighbour Joining cо 100 бутстрэп-репликами - оно на рисунке 3.
Дерево подтверждает серьезное расхождение между двумя ортологическими группами (бутстрэп-поддержка 100). Выделяющихся по длине тривиальных ветвей не замечено.
© Дарья Горбачева | изменено 13.04.2016 |

