| Главная страница | Семестры | О себе | Ссылки |
Для работы я взяла структуру с идентификатором 4GLW - ДНК-лигаза из пневмококка (с этой структурой я работала на первом курсе, и она удовлетворяет всем требованиям). На странице структуры на PDB указаны следующие данные о ней:
Разрешение: 2.0 Å
R-factor: 0.207
R-free: 0.260
Такие параметры хороши, структуру можно использовать в дальшейшей работе.
Изображение электронной плотности вокруг остова полипептидной цепи
В программе PyMOL были получены изображения электронной плотности (ЭП) вокруг полипептидной цепи указанного белка на разных уровнях подрезки (рисунки 1-3).
Для получения изображений использовались команды:
|
load 4glw.pdb, 4glw load 4glw.ccp4, 4glw_map isomesh surface1, 4glw_map, 1.0, name ca+c+n+o and !het, carve=2 isomesh surface2, 4glw_map, 2.0, name ca+c+n+o and !het, carve=2 isomesh surface3, 4glw_map, 3.0, name ca+c+n+o and !het, carve=2 |
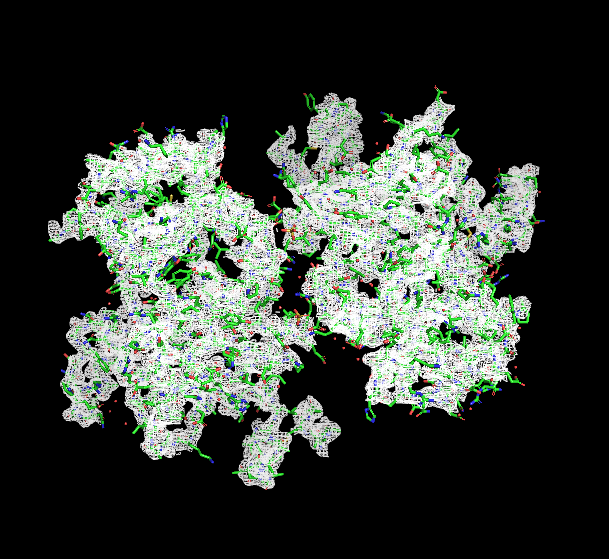
Рис. 1. ЭП на уровне подрезки 1
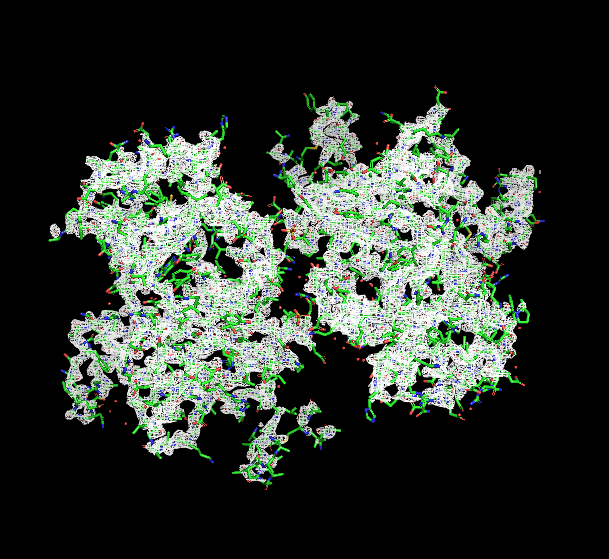
Рис. 2. ЭП на уровне подрезки 2
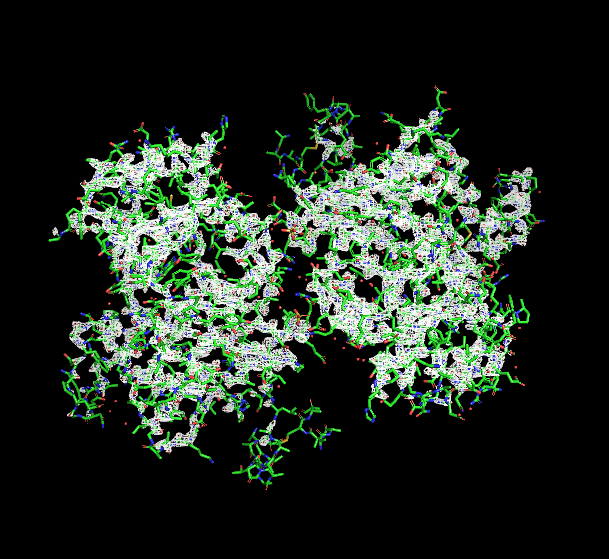
Рис. 3. ЭП на уровне подрезки 3
Видно, что чем выше уровень поверхности уровня, тем меньше атомов вписываются в ЭП.
Изображение электронной плотности вокруг отдельных остатков
Были выбраны остатки 187, 188, 189 цепи А: фенилаланин, аланин, аспарагин. Электронная плотность для них визуализировалась на тех же уровнях подрезки 1, 2 и 3, соответствующие изображения на рисунках 4-6.
Для получения изображений использовались команды:
|
select residues, chain A and resi 187-189 hide everything, not residues zoom residues isomesh surface1_res, 4glw_map, 1.0, name ca+c+n+o and !het and resi 187-189 and chain A, carve=2 isomesh surface2_res, 4glw_map, 2.0, name ca+c+n+o and !het and resi 187-189 and chain A, carve=2 isomesh surface3_res, 4glw_map, 3.0, name ca+c+n+o and !het and resi 187-189 and chain A, carve=2 |
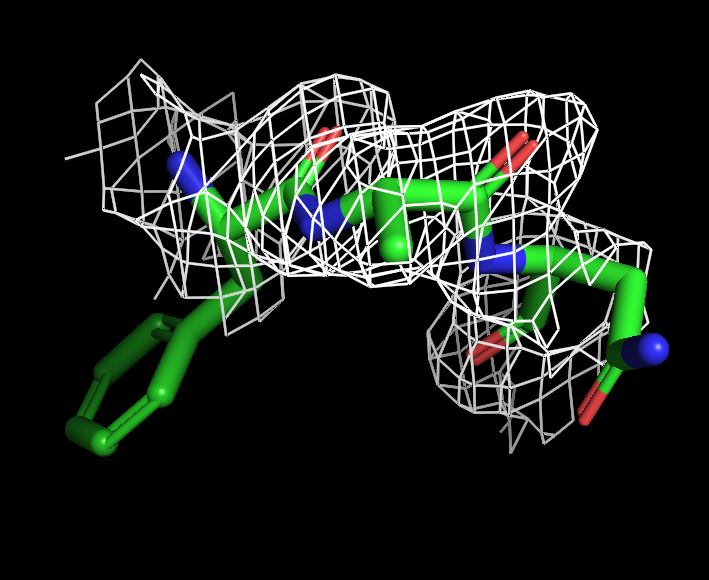
Рис. 1. ЭП трех остатков на уровне подрезки 1
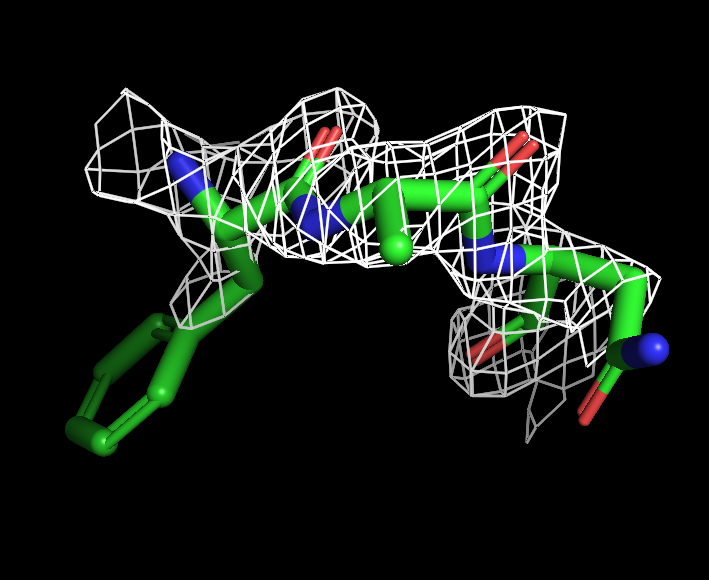
Рис. 2. ЭП трех остатков на уровне подрезки 2
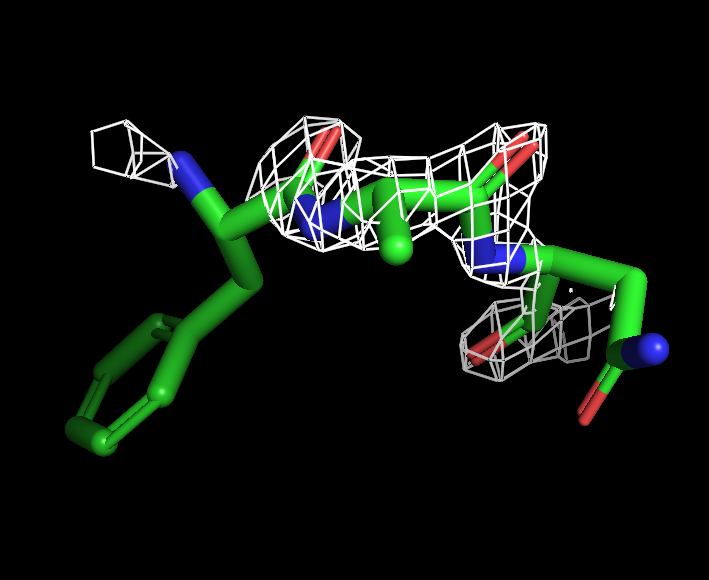
Рис. 3. ЭП трех остатков на уровне подрезки 3
Видя, что не все атомы вписались в ЭП на уровне подрезки 1, я изобразила ЭП на уровне подрезки 0.1 (рисунок 7), но это не помогло вписать все атомы.
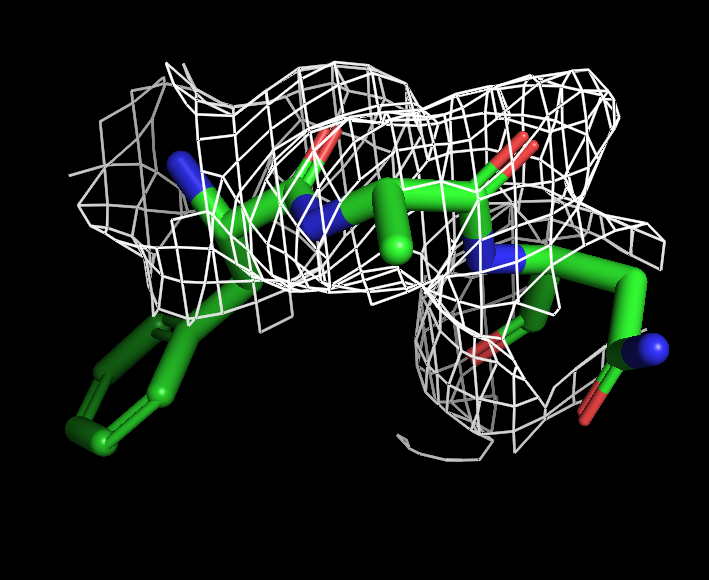
Рис. 3. ЭП трех остатков на уровне подрезки 0.1
Видимо, ЭП плохо отображает структуру белка в случае данной структуры. Не зная последовательности, эту структуру вряд ли можно было бы создать.
© Дарья Горбачева | изменено 6.11.2017 |

