| Главная страница | Семестры | О себе | Ссылки |
В этом задании я работала с рибосомальным белком L30 из Thermococcus celer (Идентификатор PDB 1H7M), с ним я работала в практикуме по ЯМР. Как видно из рисунке 1, в белке 5 α-спиралей и 4 тяжа β-листа.
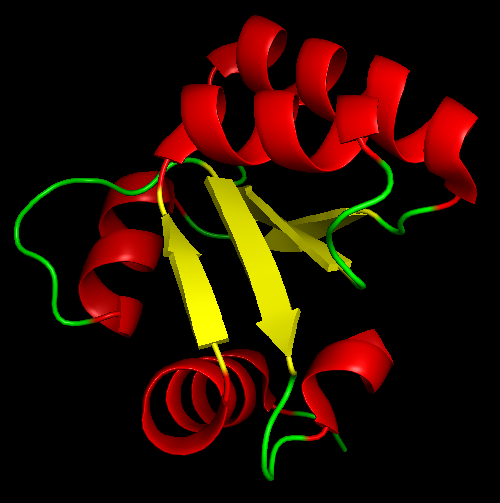
Рис. 1. Структура 1H7M, раскраска по структуре: красный - α-спирали, желтый - β-тяжи, зеленый - неструктурированные фрагменты
В pdb-файле содержится такая информация об элементах вторичной структуры:
HELIX 1 1 ASP A 2 GLY A 14 1 13 HELIX 2 2 GLY A 19 MET A 29 1 11 HELIX 3 3 ARG A 42 GLY A 57 1 16 HELIX 4 4 THR A 66 LEU A 74 1 9 HELIX 5 5 ARG A 92 LEU A 96 5 5 SHEET 1 AA 4 LYS A 15 MET A 18 0 SHEET 2 AA 4 ALA A 82 ASP A 87 -1 O ALA A 84 N VAL A 17 SHEET 3 AA 4 LEU A 34 ALA A 38 -1 O LEU A 34 N VAL A 85 SHEET 4 AA 4 VAL A 60 PHE A 63 1 O TYR A 61 N VAL A 37 |
Для предсказания вторичной структуры использовалась программа DSSP, запущена на kodomo с помощью команды:
|
mkdssp -i 1h7m.pdb -o 1h7m.dssp |
Выдача программы по формату отличается от приведенных выше строк из pdb-файла: для каждого остатка прописана принадлежность к определенному элементу вторичной структуры (а в файле со структурой, наоборот, для спирали или тяжа прописаны составляющие их остатки).
Информация из PDB и предсказания DSSP собрана в таблице 1. Видно, что все β-тяжи предсказаны абсолютно точно, границы α-спиралей предсказываются с ошибкой в 1-2 остатка, но это незначительно.
Таблица 1. Соотнесение информации о вторичной структуре 1H7M из PDB с предсказанием DSSP
Структура |
PDB |
DSSP |
α-спираль |
2-14 |
4-14 |
α-спираль |
19-29 |
21-29 |
α-спираль |
42-57 |
43-56 |
α-спираль |
66-74 |
67-73 |
α-спираль |
92-96 |
- |
β-тяж |
15-18 |
15-18 |
β-тяж |
82-87 |
82-87 |
β-тяж |
34-38 |
34-38 |
β-тяж |
60-63 |
60-63 |
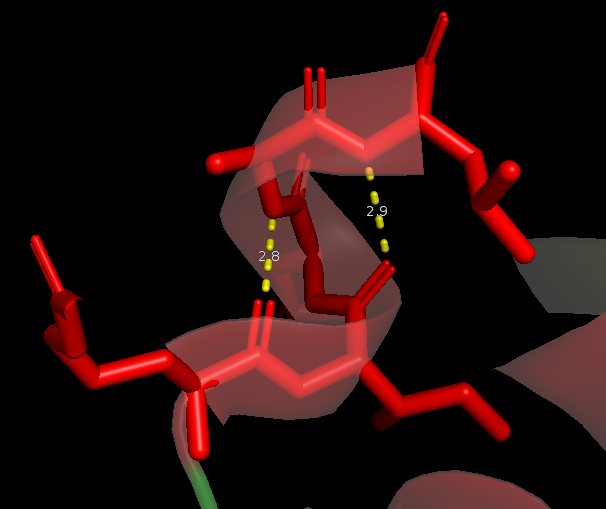
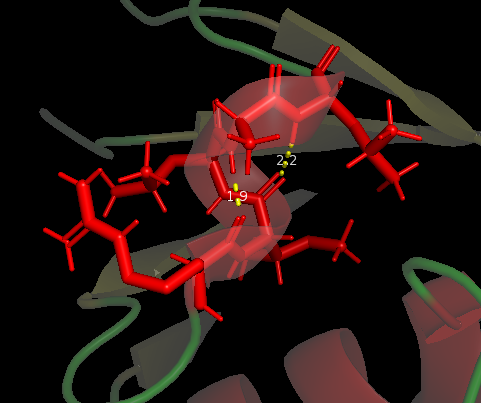
Одна α-спираль не была предсказана DSSP. Так как эта спираль маленькая и находится на конце последовательности, я подумала, что она вообще может быть обозначена в pdb-файле неверно, а поддерживающие ее связи - артефакт кристаллизации. На рисунке 2 видно, что спираль поддерживается всего двумя Н-связями. Однако там же представлена ЯМР-структура для этого белка 1GO0 (усредненная по всем моделям), на ней эта спираль и эти две Н-связи тоже представлены, что опровергает мое предположение. Значит, DSSP не справился с предсказанием всех эементов вторичной структуры.
|
|
|
Рис. 2. Непредсказанная α-спираль на РСА-структуре (слева) и на усредненной ЯМР-структуре (справа)
© Дарья Горбачева | изменено 11.12.2017 |