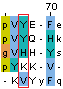
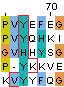
| Главная страница | Семестры | О себе | Ссылки |
Совмещение структур белка и его гомологов
Для этого задания решила работать с рибосомальным белком L30 из археи Thermococcus celer (РСА-структура 1H7M), ранее работала с ним в практикуме №4, посвященном сравнению ЯМР и РСА. Для поиска сходных структур использовался сервис PDBeFold, дефолтные параметры. По результатам поиска было отобрано 4 структуры, описанные на рисунке 1.
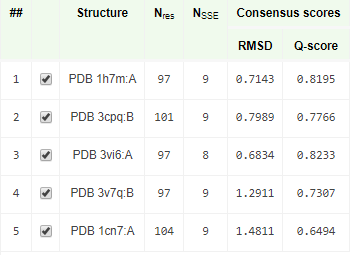
Рис. 1. Параметры отобранных для выравнивания находок
На рисунке 2 общий вид пространственного выравнивания. Видно, что визуально все структуры сходные.
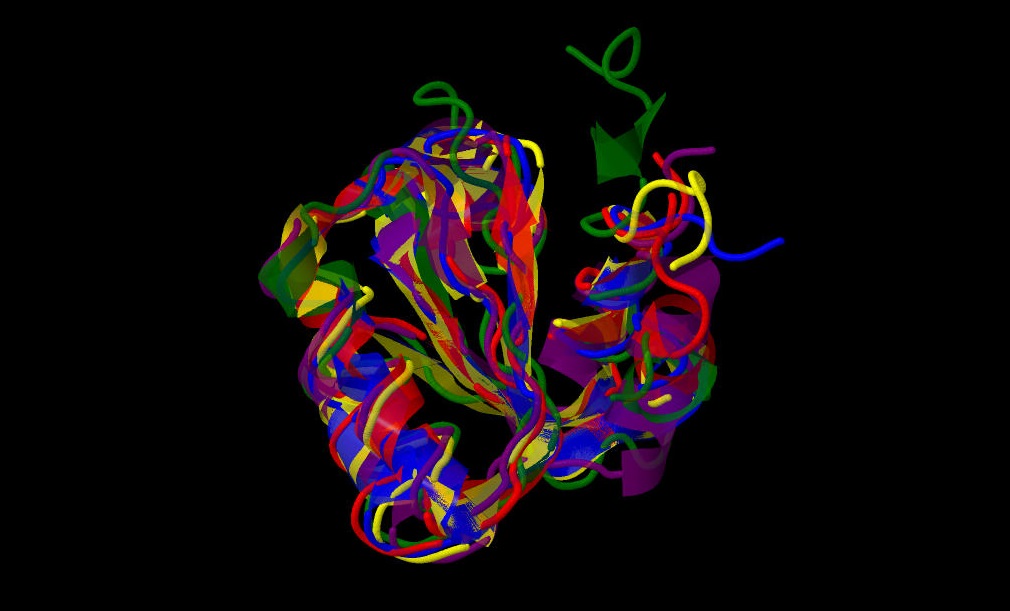
Рис. 2. Структурное выравнивание, разные модели - разными цветами
Сервис выдал выравнивание по структуре в текстовом формате. С помощью muscle в JalView было построено выравнивание по последовательности. Выравнивания представлены также на рисунках 3 и 4.

Рис. 3. Выравнивание по структуре

Рис. 4. Выравнивание по последовательности (с помощью muscle)
Видно, что выравнивания очень сходны, но по некоторым позициям все же отличаются, особенно в С-концевой части, где выравнивание по последовательности в целом компактнее. В качестве примера можно привести колонку 67, которая была не так воспроизведена выравниванием по последовательности (см. рисунок 5). На рисунке 6 изображено соответствующее позиции 67 выравниваний место на пространствнном выравнивании.
|
|
|
Рис. 5. Участок несовпадения выравниваний по структуре (слева) и по последовательности (справа). Красным выделены остатки, выровненные по структуре
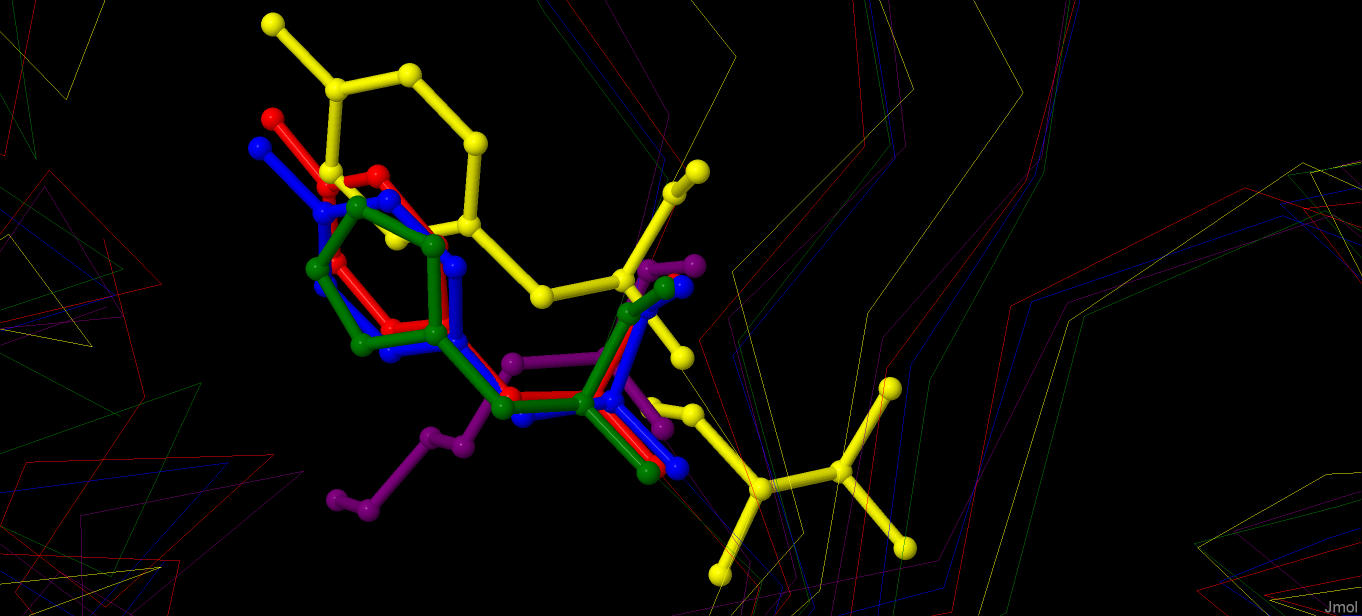
Рис. 6. Участок структуры, соответствующий спорной позиции
По рисунку 6 видно, что ни одно из двух текстовых выравниваний "не право". На текстовом выравнивании, основанном на совмещении структур, колонке 67 соответствуют 4 хорошо накладывающиеся друг на друга нежелтые остатка и желтый валин (67 остаток структуры 1CN7). Видно, что при автоматическом переводе структурного выравнивания в текстовый формат произошла ошибка и на самом деле в этой позиции должен стоять 68 тирозин 1CN7, как и реализовано в выравнивании по последовательности. Однако muscle допустил неточность (относительно выравнивания структур) в положении тирозина модели 3V7Q.
Совмещение по заданному выравниванию
Для задания были выбраны две структуры в составе константного домена Т-клеточного рецептора: регион D:118-206 из 1QRN из цепи α (на рисунке 7), регион E:119-247 из 1MI5 из цепи β (на рисунке 8).
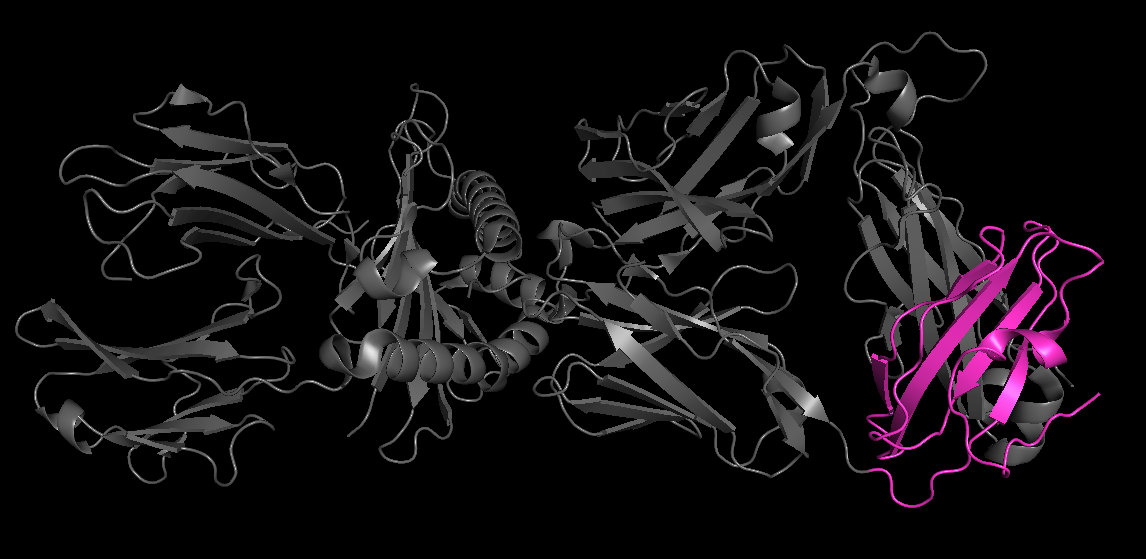
Рис. 7. Структура 1QRN c выделенным участком D:118-206
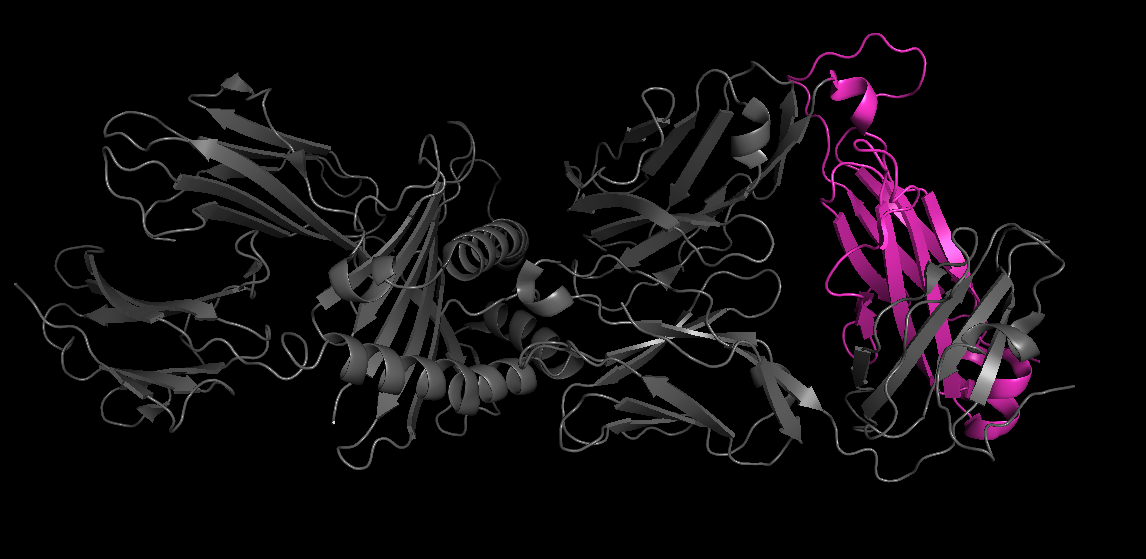
Рис. 8. Структура 1MI5 c выделенным участком E:119-247
На рисунке 9 выравнивание выбранных участков стандартной программой align в PyMol. Видно, что структуры выравниваются плохо. Визуально кажется, что одну из структур следовало бы повернуть на 180o слева направо.
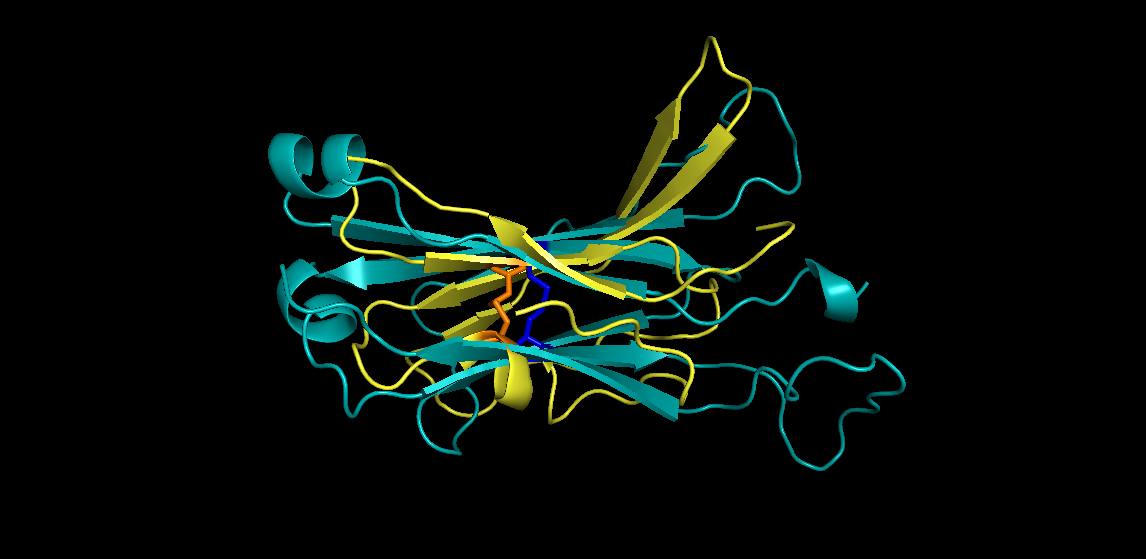
Рис. 9. Выравнивание выбранных участков при помощи PyMol. Более ярким цветом выделены цистеиновые мостики: для каждой структуры нашлось по одному
При помощи сервиса SheeP были составлены карты β-листов этих двух регионов, они на рисунках 9 и 10.
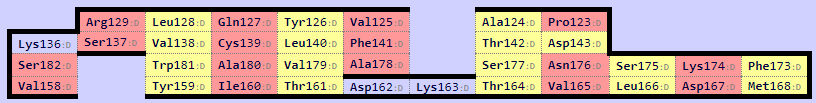
Рис. 10. Карта β-листов для α-цепи Т-клеточного рецептора (участок D:118-206 1QRN)

Рис. 11. Карта β-листов для β-цепи Т-клеточного рецептора (участок E:119-247 1MI5). Нас интересует верхний лист
На основе цистеиновых мостиков были составлены следующие команды для выравнивания:
|
select A, 1qrn and chain D and resi 126-128+138-140+180-182 and name CA select B, 1mi5 and chain E and resi 129-131+147-149+193-195 and name CA pair_fit A, B |
Получившееся выравнивание представлено на рисунке 12, а также в файле. Как видно, цистеиновые мостики действительно выровнялись, но это не улучшило общую картину, структуры по-прежнему выровнены очень плохо. Все еще кажется, что для лучшего выравнивания одну из структур стоит повернуть относительно другой, но я не знаю, как реализовать это.
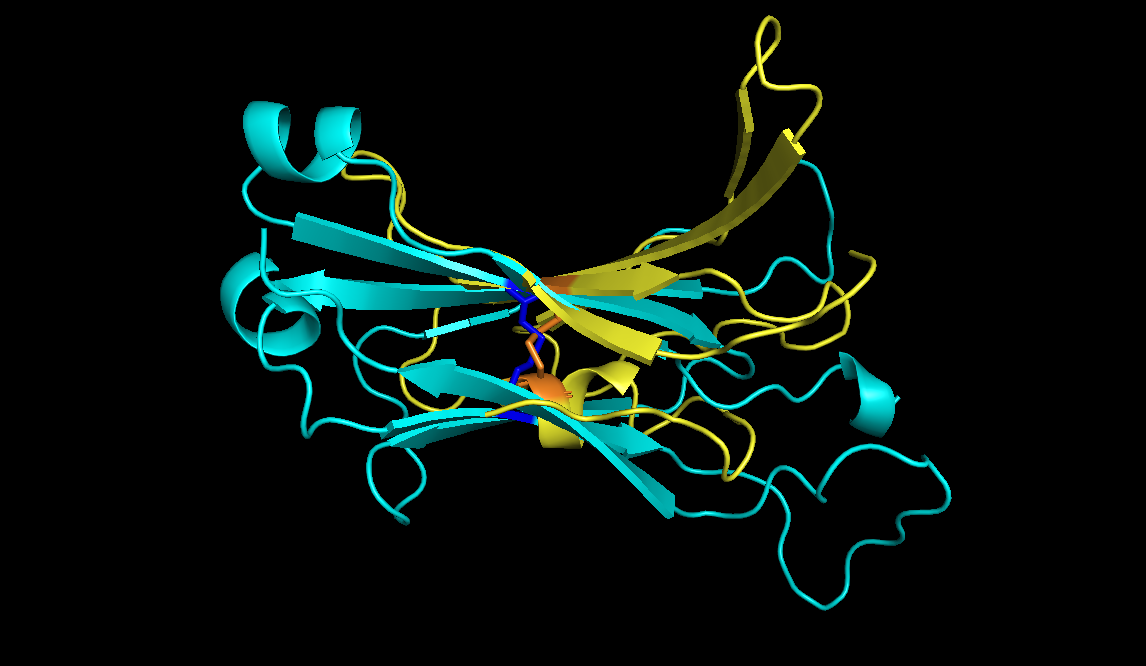
Рис. 12. Выравнивание выбранных участков с помощью приведенных выше операций
© Дарья Горбачева | изменено 6.01.2018 |