Построение карт локального сходства
Буду искать геномы на сайте NCBI, и там же, используя blast для 2 последовательностей, строить карты локального сходства, используя разные алгоритмы (megablast и blastn). Решил взять Escherichia coli, потому что самая изученная бактерия, значит будет много хороших сборок для разных штаммов. Интересно узнать насколько отличаются разные штаммы в пределах одного вида. Выбрал ST2747, он был с геномным уровнем сборки. Сравню с другими штаммами с хромосомным уровнем сборки и выше. Более менее богатая событиями карта сходств получилась при сравнении со штаммом VH1
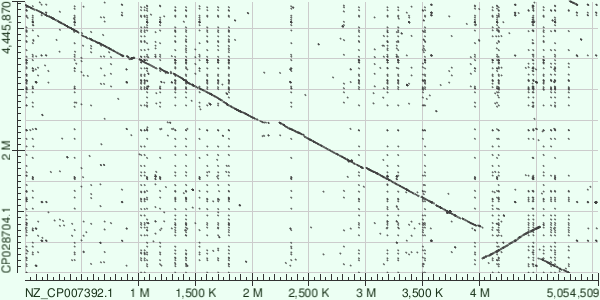
Основная линия на карте наклонена в другую сторону, это значит, что геномы были прочитаны начиная с одного места, но в разные стороны. Геном штамма ST2747 (по горизонтали) длиннее на 600 000 нуклеотидов, чем геном штамма VH1 (по вертикали). Видно, что у VH1 есть делеции в следующих областях: 1.7М, 2.5М, 3.4М, 3.5М, 3.9М (М — миллион нуклеотидов). На одном из участков 0.25-0.75M у ST2747 или 4-4.5М у ST2747 произошла крупная инверсия.
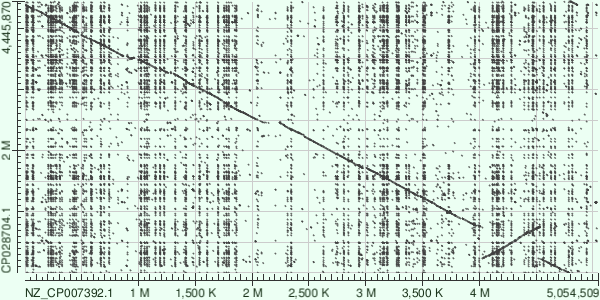
Как известно blastn более чувствительный алгоритм, поэтому помимо того, что было на карте построенной megablast, видим множество дополнительных точек на карте, насколько я понимаю, это какие-то небольшие повторы в геноме.