Сборка генома de novo
Сборка будет происходить на основе чтений, полученных по технологии Illumina с кодом доступа SRR4240356. Для их загрузки использовалась следующая команда
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/006/SRR4240356/SRR4240356.fastq.gz
Подготовка чтений программой trimmomatic
Были вырезаны адаптеры, которые попали в чтения в ходе секвенирования
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240356.fastq.gz SRR4240356_trimmed.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7
И удалены чтения с качеством ниже 20 и чтения длина которых меньше 32 нуклеотидов
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240356_trimmed.fastq.gz SRR4240356_trimmed2.fastq.gz TRAILING:20 MINLEN:32
В итоге после первой команды было выброшено 2% чтений
Input Reads: 7511529 Surviving: 7358438 (97.96%) Dropped: 153091 (2.04%)
После второй еще 4% от того, осталось
Input Reads: 7358438 Surviving: 7053346 (95.85%) Dropped: 305092 (4.15%)
А файл стал весить 155 Мб вместо 167 Мб
Сборка генома программами velveth и velvetg
velveth подготавливает k-меры заданной длины исходя из чтений и складывает их в папку название которой, указано в первом аргументе
velveth assembly 31 -short -fastq.gz SRR4240356_trimmed2.fastq.gz
velvetg собирает k-меры из папки assembly в последовательности
velvetg assembly
Информация о получившихся в ходе сборки контигах содержится в файле stats.txt, а их последовательности в config.fa. Чтобы найти самые длинные использовалась команда
cat stats.txt | sort -nrk2
| ID | Длина | Покрытие |
|---|---|---|
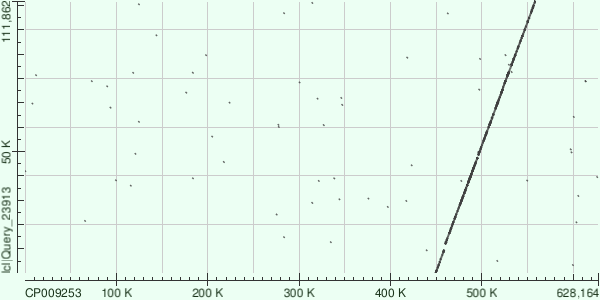
| 8 | 111962 | 38.66 |
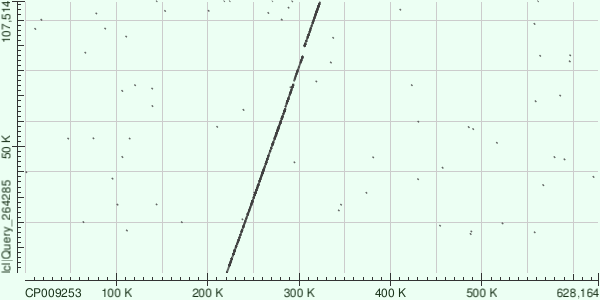
| 6 | 107488 | 34.17 |
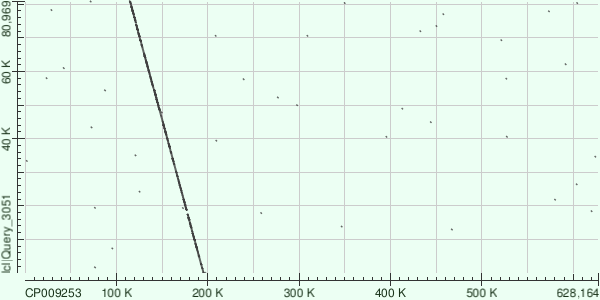
| 10 | 80939 | 37.52 |
Параметр N50 получившейся сборки — 65554 нт (было получено из текстовой выдачи velvetg), было много контигов длиной 1 k-мер, некоторые из них с аномально большим покрытием, например — 267 000 у ID 64, 1134 у ID 127
Сравнение получившихся контигов с исходной хромосомой
Для сравнения использовался алгоритм blastn с параметрами по умолчанию (почему-то при выборе megablast сайт не запускался, а только перезагружался, а с blastn работало..).
Координаты участка хромосомы, соответствующего контигу и характеристики выравнивания или выравниваний (число однонуклеотидных различий, число гэпов) можно найти в файлах Hit table