Мотивы и сигналы - 3
1. Консервативный мотив в выравнивании последовательностей гомологичных белков
Искал домен для анализа на сайте Pfam по ключевому слову Endonuclease. Выбрал Endonuclease_NS (PF01223), число последовательностей seed – 56. Скачал выравнивание, открыл в Jalview, убрал последовательности совпадающие более чем на 80%, осталось 49. Далее, чтобы найти участок имеющий наибольшее информационное содержание (IC) ориентировался на параметр conservation, наиболее консервативной областью оказалась 140-146 нуклеотидов (нт), выберу её для проверки. Также консервативными были области 173-177, 309-317, 236-241 (упорядочены по убыванию IC).

Для описания выбранного мотива был составлен следующий паттерн: [VLIMFWYC][DAQN]RGH[LMQI].[PRA] (не нашел можно ли обозначить гидрофобные АК одной буквой, поэтому в первой позиции пришлось их просто перечислить). Было найдено 35 соответствий.
Затем с использованием сервиса MyHits был произведён поиск найденного мотива в базе данных SwissProt. В результате получено 83 находки, лишь часть из них относились к нуклеазам, много находок было среди НАДН-хинон оксидоредуктаз, остальные принадлежали другим семействам. Полагаю, это из-за того, что паттерн не особо специфичный оказался.
2. Поиск мотива специфичного для клады
В JalView было построено филогенетическое дерево с помощью NJ. Выделил кладу из 10 последовательностей, удалил пустые колонки в выравнивании, из-за чего номерация мотивов немного сдвинулась. Тот мотив, что я выбрал в предыдущем пункте (140-146 нт) сдвинулся и немного расширился — 102-111 нт.
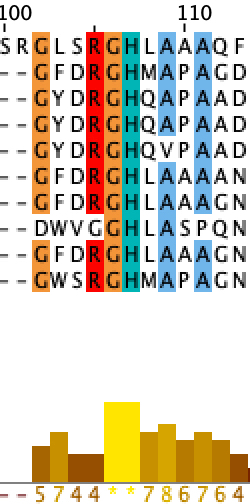
Паттерн мотива – [GD][LFWY][DAQN]RGH[LMQI][AV].[AP] , по нему было найдено 11 последовательностей (только одна не из клады). В виду того, что в паттерне, я указал буквы, которые встречались в определенной позиции в выравнивании клады всего один раз ([GD][AV][AP]), можно было ожидать неспецифичной находки вне клады, как раз содержащей эти вариации. В итоге можно сделать вывод, что мотив специфичен для данной клады.
3. PSI-BLAST
Выбрал Q7VDL2. Probable septum site-determining protein MinC — белок длиной 221 аминокислоты, выделен из Prochlorococcus marinus, ингибитор клеточного деления, который блокирует образование кольцевых перегородок, путем предотвращения полимеризации белка FtsZ.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 146 | Q9AG20 | 0,005 | A8GFG7 | 0,005 |
| 2 | 188 | B6JKXO | 7,00E-08 | - | - |
| 3 | 188 | Q9ZM51 | 2.00E-12 | A7H8E6 | 0,014 |
| 4 | 189 | A8MHK8 | 0,001 | A7H8E6 | 0,013 |
Основное кол-во находок появилось после второй итерации. На четвертой, судя по E-value, добавился лишний белок. Это подтверждается, если посмотреть информацию о нем, он выполняет другую функцию и принадлежит другому семейству белков
4. Проверка числа TA в геноме бактерии
Для проверки использовал геном той же бактерии, что и в прошлом практикуме: E.coli K-12 MG1655. Всего в геноме 4 641 652 нт, GC-состав буду считать равным 0.52, в таком случае частоты А и Т равны по 0.24 . Тогда ожидаемое число ТА составляет 267 359 нт. А фактическое число их получилось 212 024 нт. Количество сайтов TA в геноме имеет биномиальное распределение, но его можно приблизить к нормальному, т.к. кол-во сайтов очень велико. В таком случае, можно использовать классический z-test для проверки нулевой гипотезы о том, что фактическое и ожидаемое число сайтов были получены из одного распределения. Данное отличие является статистически значимым, так как p-value практически равен нулю.