Нахождение гомологов белка CLPX_ECOLI
Среди всех белков бактерий, выбранных в практикуме 1, буду искать гомологи белка CLPX_ECOLI. Далее использую bash и локальный BLAST на сервере kodomo
Объединил файлы с геномами бактерий
cat *.fasta >> bacteria.fasta
Создал базу данных из полученного набора белков:
makeblastdb -in bacteria.fasta -dbtype prot
Запустил blastp c порогом на E-value 0,001
blastp -task blastp -query CLPX_ECOLI.fasta -db bacteria.fasta -evalue 0.001 -out blast_results.txt
В итоге было получено 20 находок.
| Sequences producing significant alignments: | Score (Bits) | E-value |
|---|---|---|
| CLPX_STRAW | 535 | 0.0 |
| CLPX_RHOJR | 534 | 0.0 |
| CLPX_NOCSJ | 528 | 0.0 |
| Q1AVT0_RUBXD | 524 | 0.0 |
| CLPX_MYCVP | 523 | 0.0 |
| CLPX_MYCTU | 519 | 0.0 |
| Q47MU4_THEFY | 516 | 0.0 |
| Q1AU05_RUBXD | 52.0 | 4e-07 |
| Q0S6Y7_RHOJR | 47.8 | 1e-05 |
| Q0S8C7_RHOJR | 47.0 | 2e-05 |
| Q47MZ2_THEFY | 43.9 | 2e-04 |
| Q1AY82_RUBXD | 43.5 | 2e-04 |
| A1TG29_MYCVP | 43.1 | 3e-04 |
| Q82QV8_STRAW | 41.6 | 5e-04 |
| A1SDV1_NOCSJ | 42.0 | 6e-04 |
| Q47KU4_THEFY | 42.0 | 7e-04 |
| Q82EE9_STRAW | 41.6 | 7e-04 |
| A1TG43_MYCVP | 41.6 | 8e-04 |
| Q82EB8_STRAW | 41.6 | 0.001 |
| FTSH_MYCTU | 41.2 | 0.001 |
Скачал из Uniprot последовательности белков. Реконструировал дерево найденных гомологов программой FastME cо следующими параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "Tree Refinement" — No refinement, 100 бутстреп реплик (использовал сайт NGPhylogeny.fr).
Newick формула дерева. Построено с использованием веб-приложения iTOL
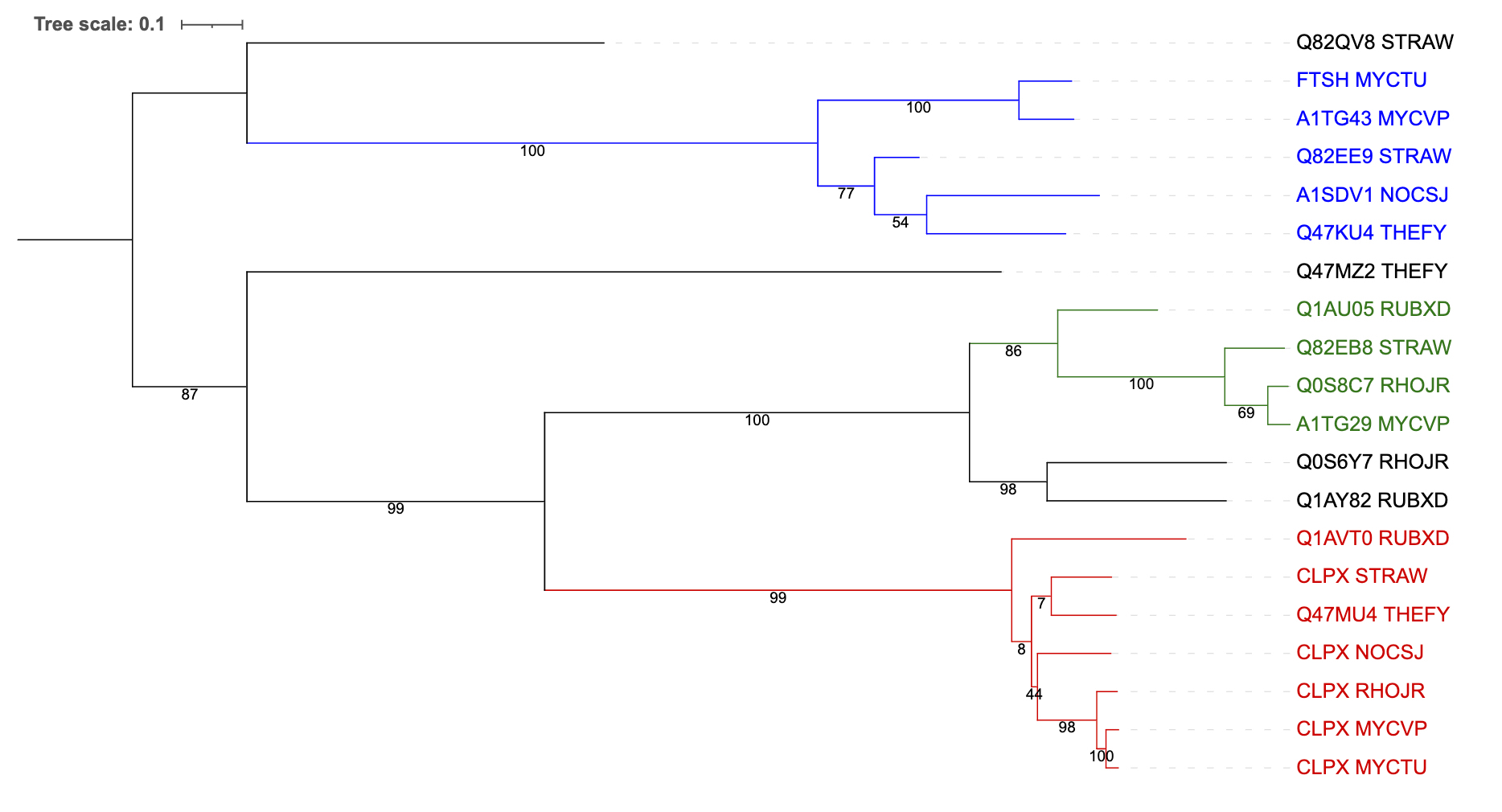
Дерево было переукоренено в среднюю точку, для ветвей добавлены значения бутстреп‑поддержки. Группы ортологических белков выделены цветами. Красным отмечена группа белков, принадлежащих семейству сериновых протеаз Clp, зеленым — группа ААА белков (АТФ-азы ассоциированные с различными клеточными aктивностями), синим — группа цинковых АТФ-зависимых металлопротеаз.
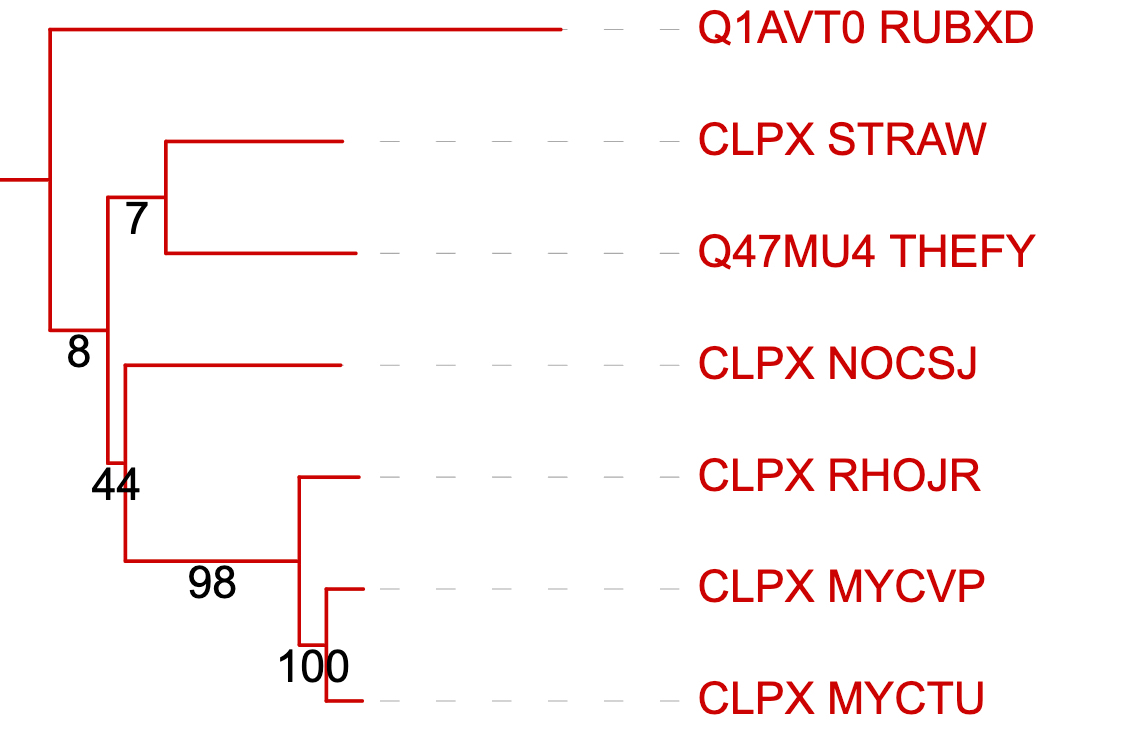
Ветвь ((((CLPX_MYCTU, CLPX_MYCVP), CLPX_RHOJR), CLPX NOCSJ), (CLPX_STRAW, Q47MU4 THEFY)) совпадает с филогенией бактерий за исключением положения NOCSJ. Предполагаю, что в эволюции сначала от общей ветви отделился вид RUBXD, а вместе с этим обособился белок Q1AVT0, потом в ходе расхождения бактерий STRAW и THEFY, белок CLPX последнего претерпел изменение и стал Q47MU4
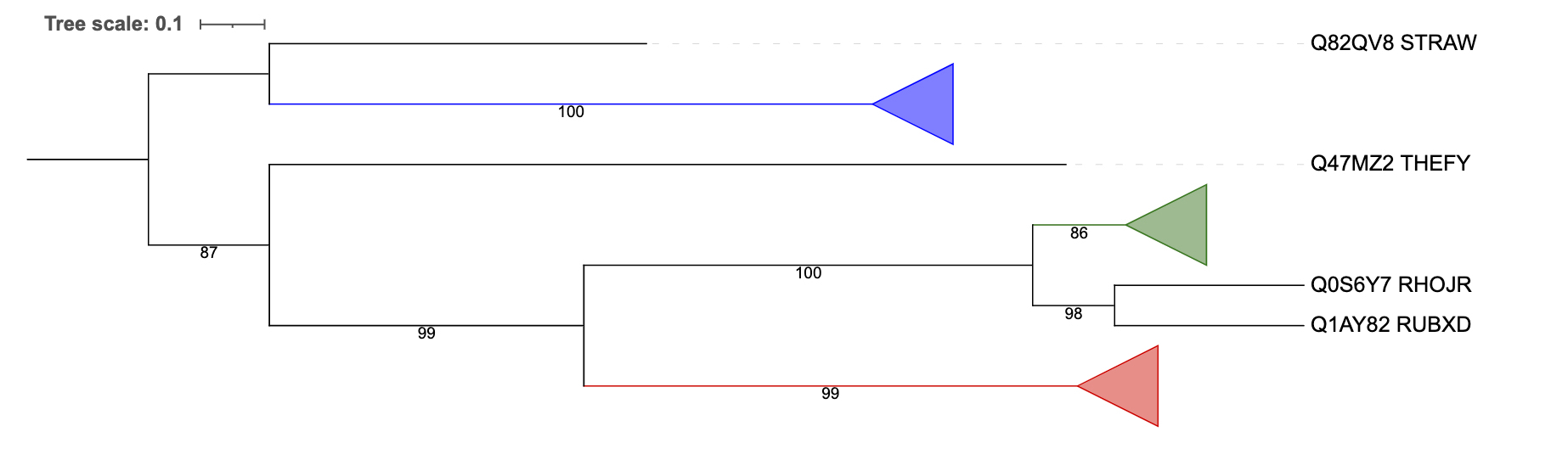
Исходя из построенного дерева среди белков можно найти ортологичные пары:
- CLPX STRAW, Q47MU4 THEFY
- Q0S8C7 RHOJR, A1TG29 MYCVP
- Q0S6Y7 RHOJR, Q1AY82 RUBXD
и паралогичные:
- Q1AY82 RUBXD, Q1AU05 RUBXD
- Q47MZ2 THEFY, Q47MU4 THEFY
- CLPX STRAW, Q82EB8 STRAW