Практикум посвящен предсказанию структуры белка по последовательности с помощью нейросети AlphaFold2 (ColabFold v1.5.5).
Задание A. Амилоиды
Сравним структуры белка-амилоида с последовательностью YGGFMTSEKSQTPLVTLFKNAIIKNAYKKGE, полученные экспериментально, со структурами предсказанными нейросетью.
 |
 |
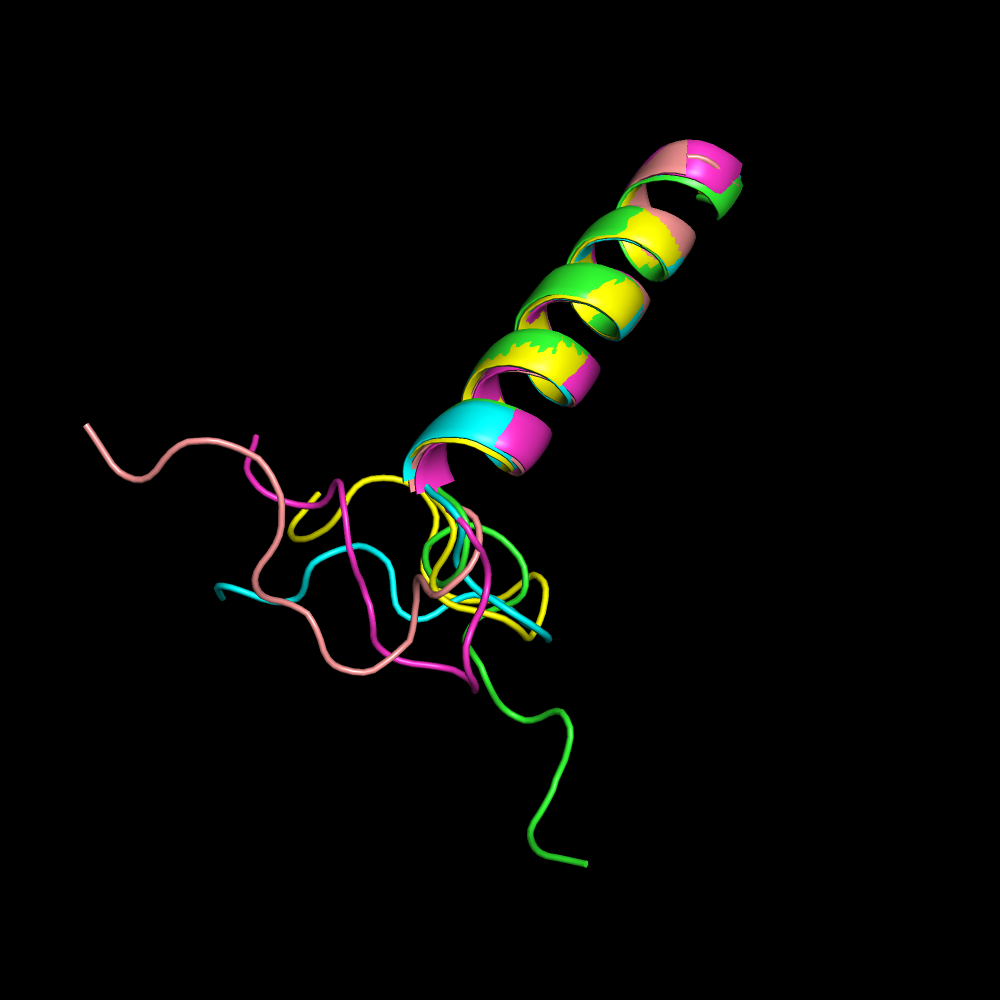
Слева 5 предсказанных структур с наибольшим pLDDT (predicted Local Distance Difference Test). Они пространственно выровняны друг на друга. Альфа-спираль у всех совпадает и предсказывается c высокой степенью уверенности (pLDDT около 90), а отличаются структуры подвижной боковой цепью (pLDDT около 60). Справа для сравнения структура мономера белка-амилоида 6TUB с той же последовательностью, полученная экспериментально методом ЯМР. Чтобы для последовательности найти похожую структуру в банке PDB использовался blastp с настройками по умолчанию. Эту же структуру можно найти просто введя последовательность в поисковик на сайте rcsb. 6TUB является гексамером амилоидных фибрил бета-эндорфина.
Предсказание имеет вторичную структуру альфа-спираль, тогда как на самом деле мономер содержит бета-слои, предсказание неверное. Но сравнивать предсказание для одной цепи и мономер комплекса из 6 цепей друг с другом не корректно, не учитывается влияние цепей друг на друга. А еще стоит отметить, что структура в банке получена методом ЯМР и является фибриллярной, а нейросеть обучалась на структурах полученных методом РСА и в основном на глобулярных.
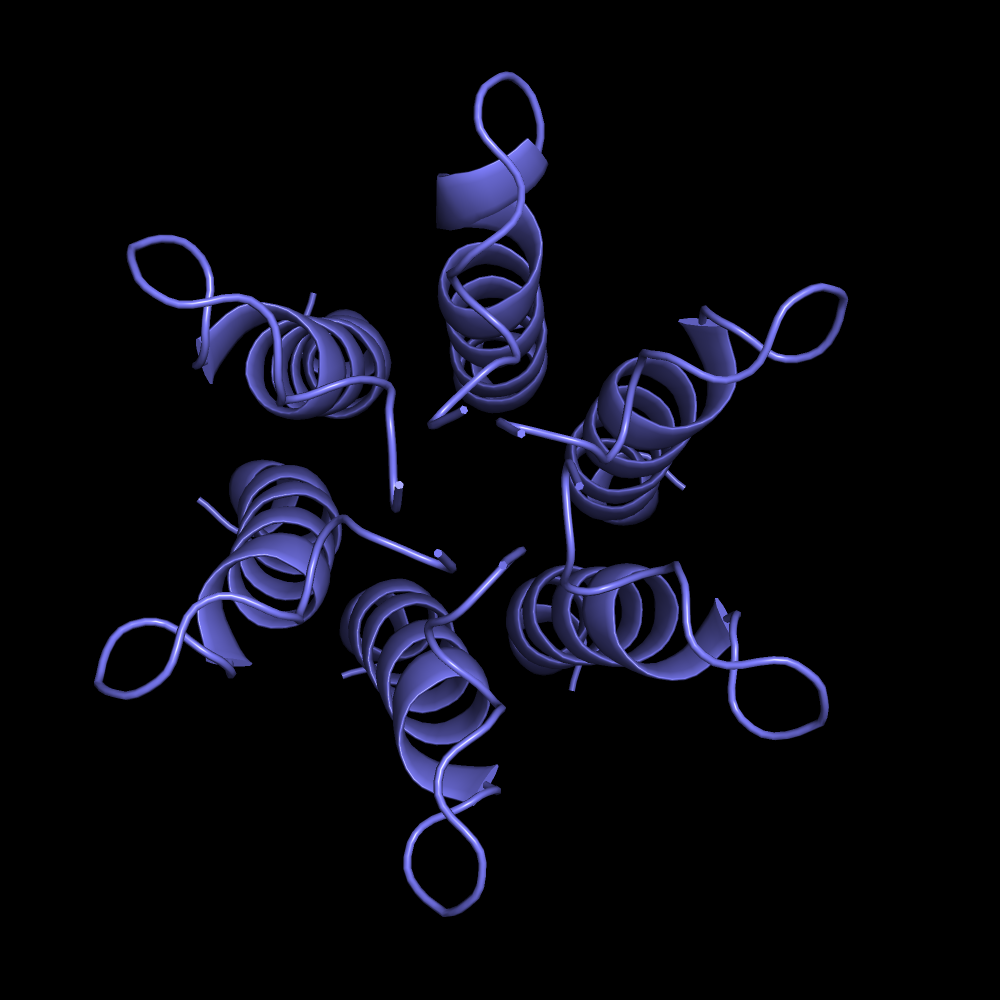
Попробуем предсказать структуру для гексамера
 |
 |
Предсказание мультимера из 6 цепей белка не изменило результат, Alphafold всё еще предсказывает альфа-спирали для всех цепей. pLDDT для каждой из цепей находится в диапазоне от 20 до 50, то есть теперь нейросеть присваивает своим прогнозам более низкую степень достоверности.
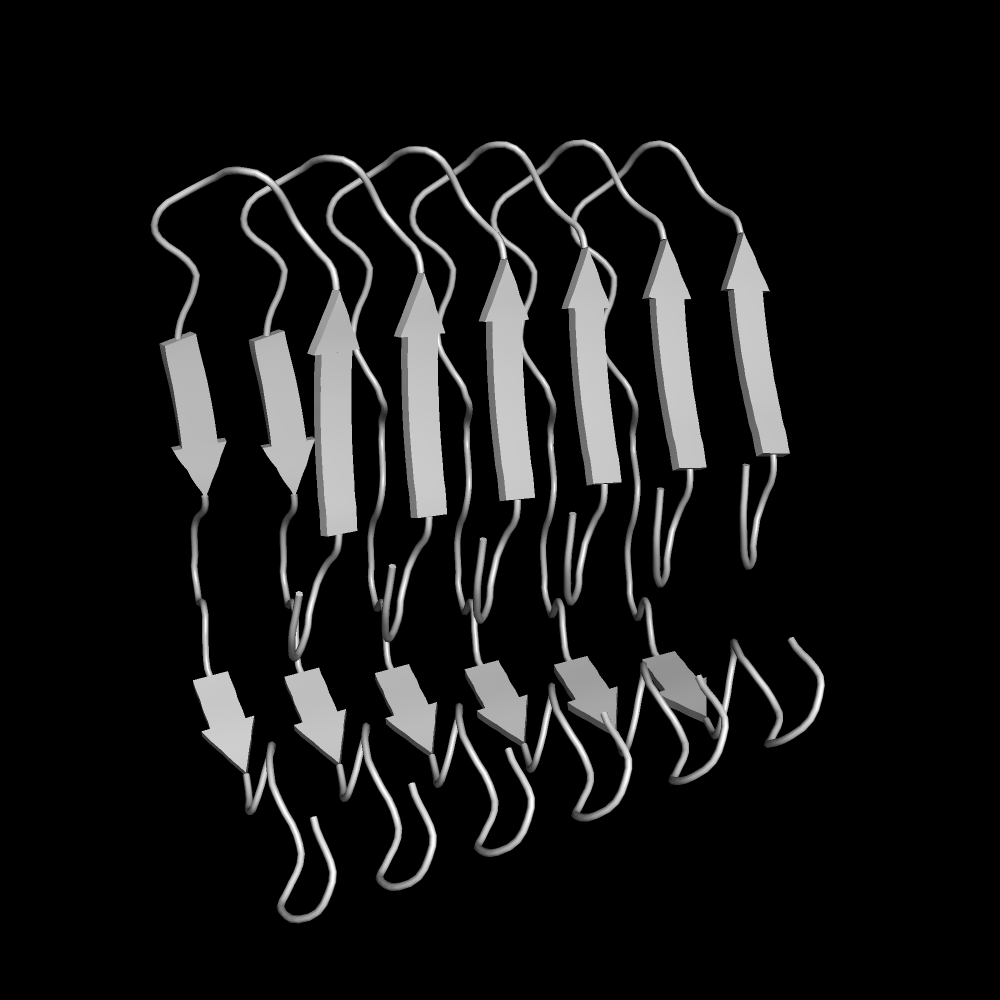
Попробуем повторить для 10 цепей
Все еще альфа-спирали. А pLDDT для каждой из цепей не поднимается даже выше 40. Думаю, это связано с тем, что при попытке соединить всё большее количество цепей друг с другом в одну структуру, каждую из них приходится всё больше деформировать и потому достоверность такой структуры падает.
Вывод: нейросеть Alphafold без дополнительного обучения не подходит для точного предсказания структуры амилоидов.
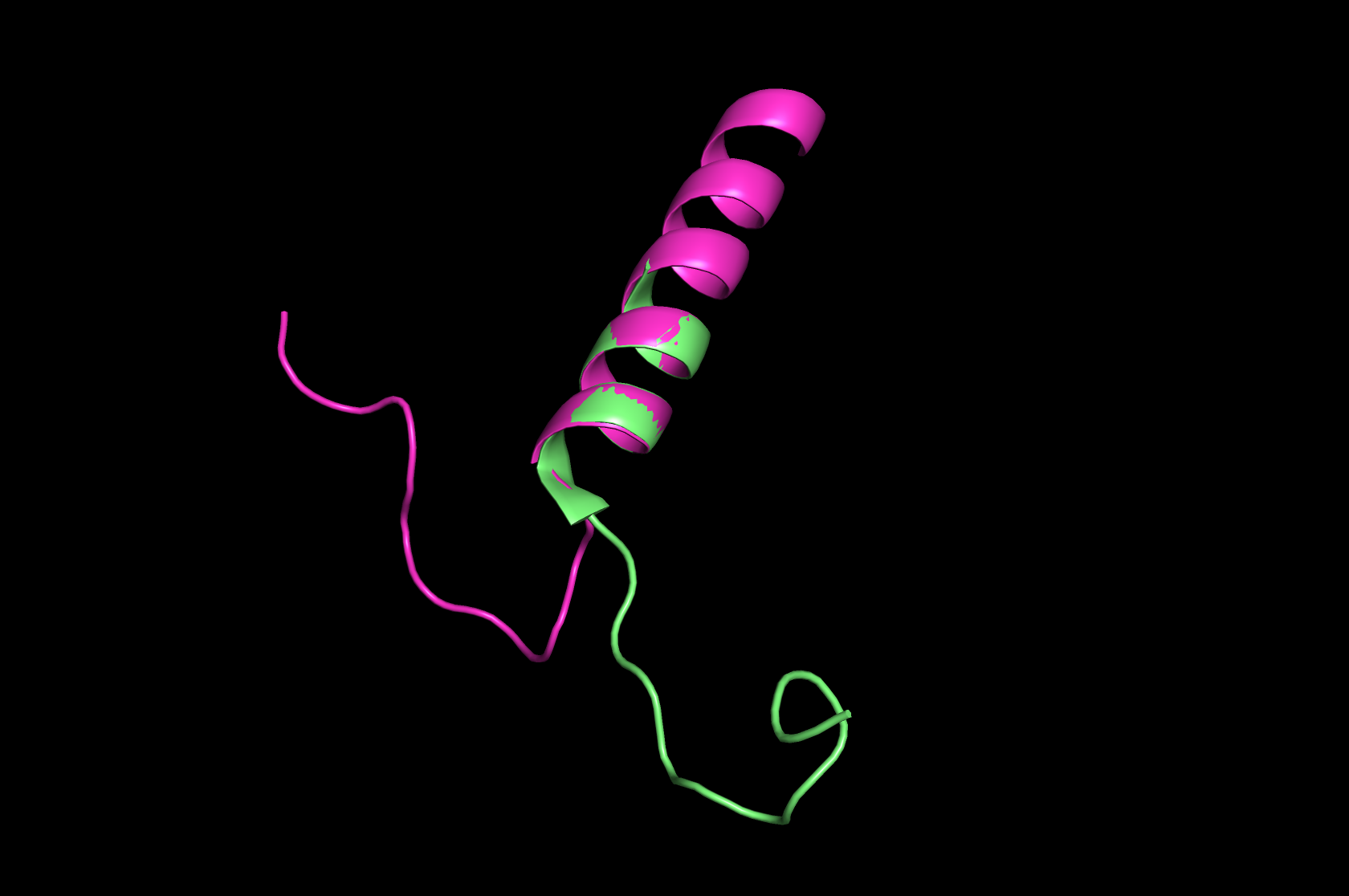
Можно попробовать сравнить предсказания со структурой обыкновенного неамилоидного бета-эндорфина. Он был взят из модели 8F7Q
 |
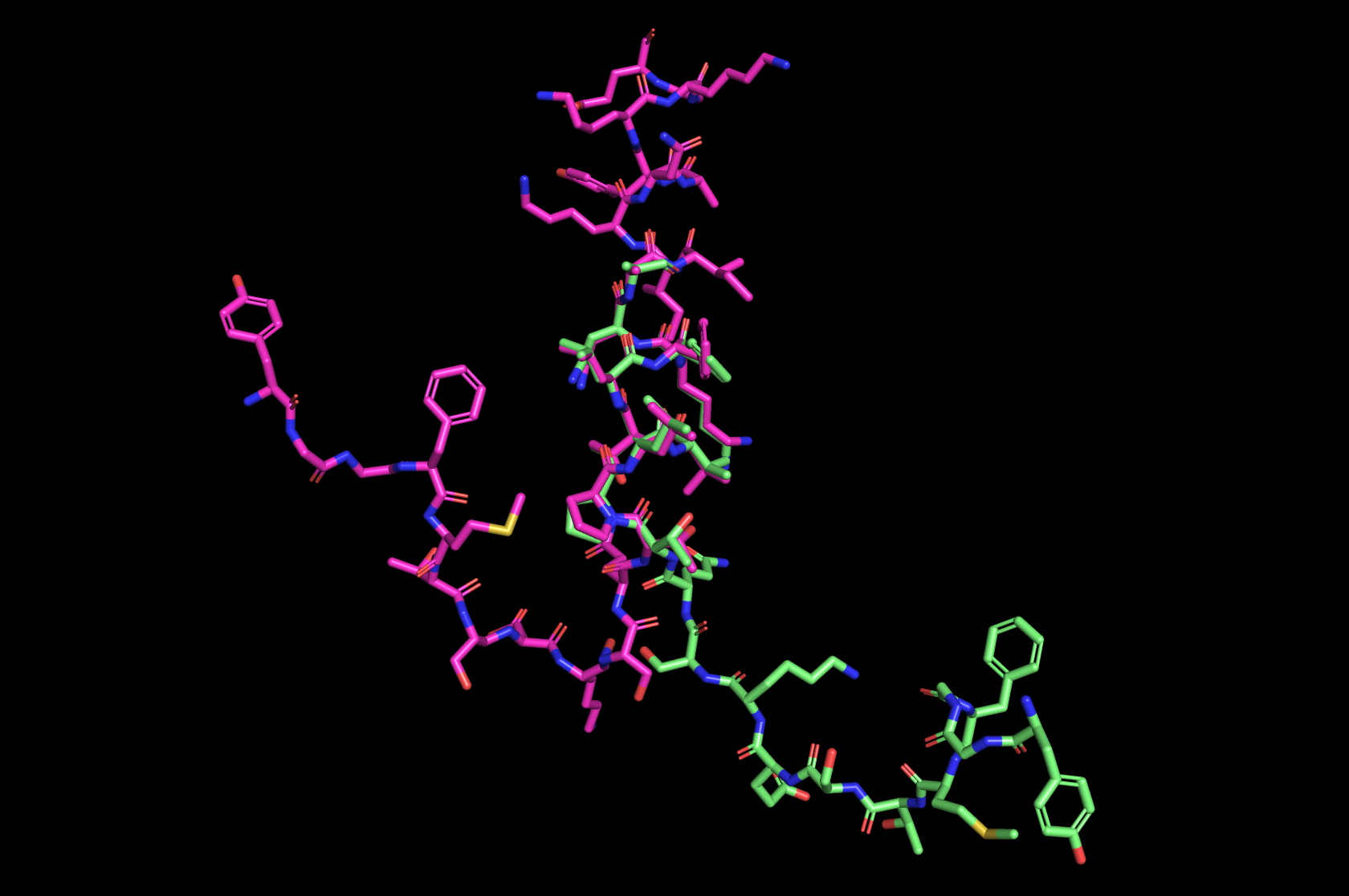 |
Выровнял пространственно по альфа-спирали, бета-эндорфин и предсказание совпадают (у бета-эндорфина в модели просто не расшифрованы остатки для половины спирали). Получается, что с предсказанием нативной конформации Alphafold справляется неплохо.
Pymol сессия: taskA.pse