PDB ID: 6K2K (ЯМР), 6M2C (РСА)
Задание 1
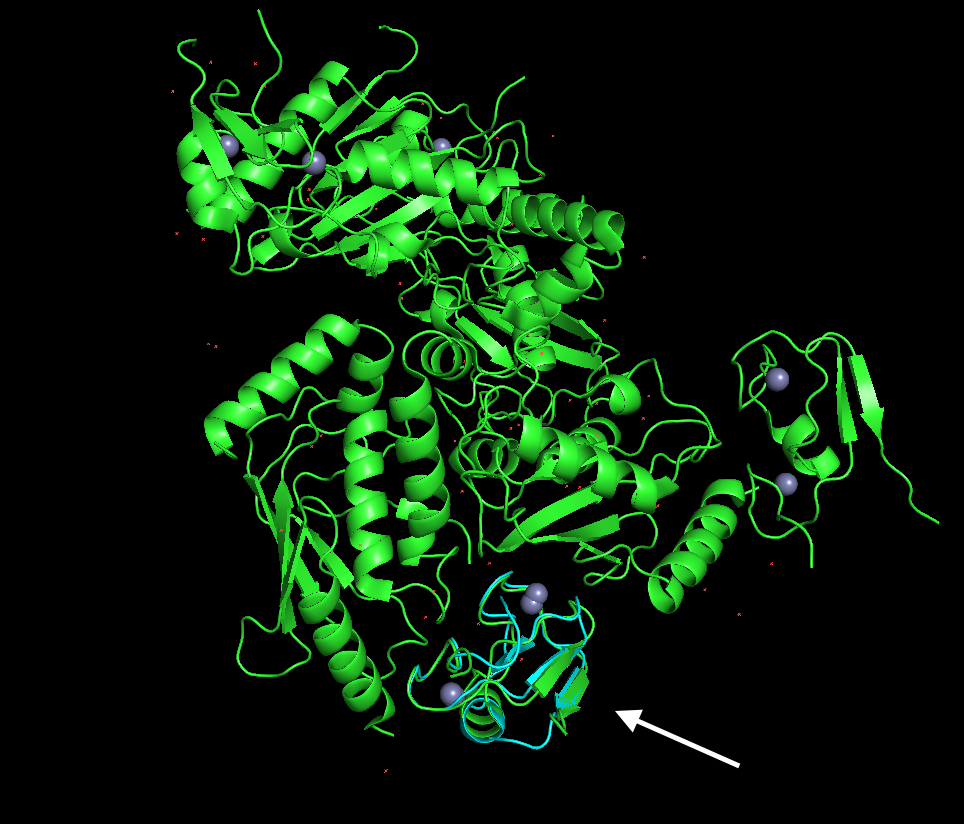
Даны две структуры одного и того же белка, полученные разными методами — рентгеноструктурным анализом (РСА) и ядерно-магнитным резонансом (ЯМР). Так как структура полученная ЯМР это только небольшой домен всего белка, а выбрать надо в пересечении, выбор связей был небольшой.
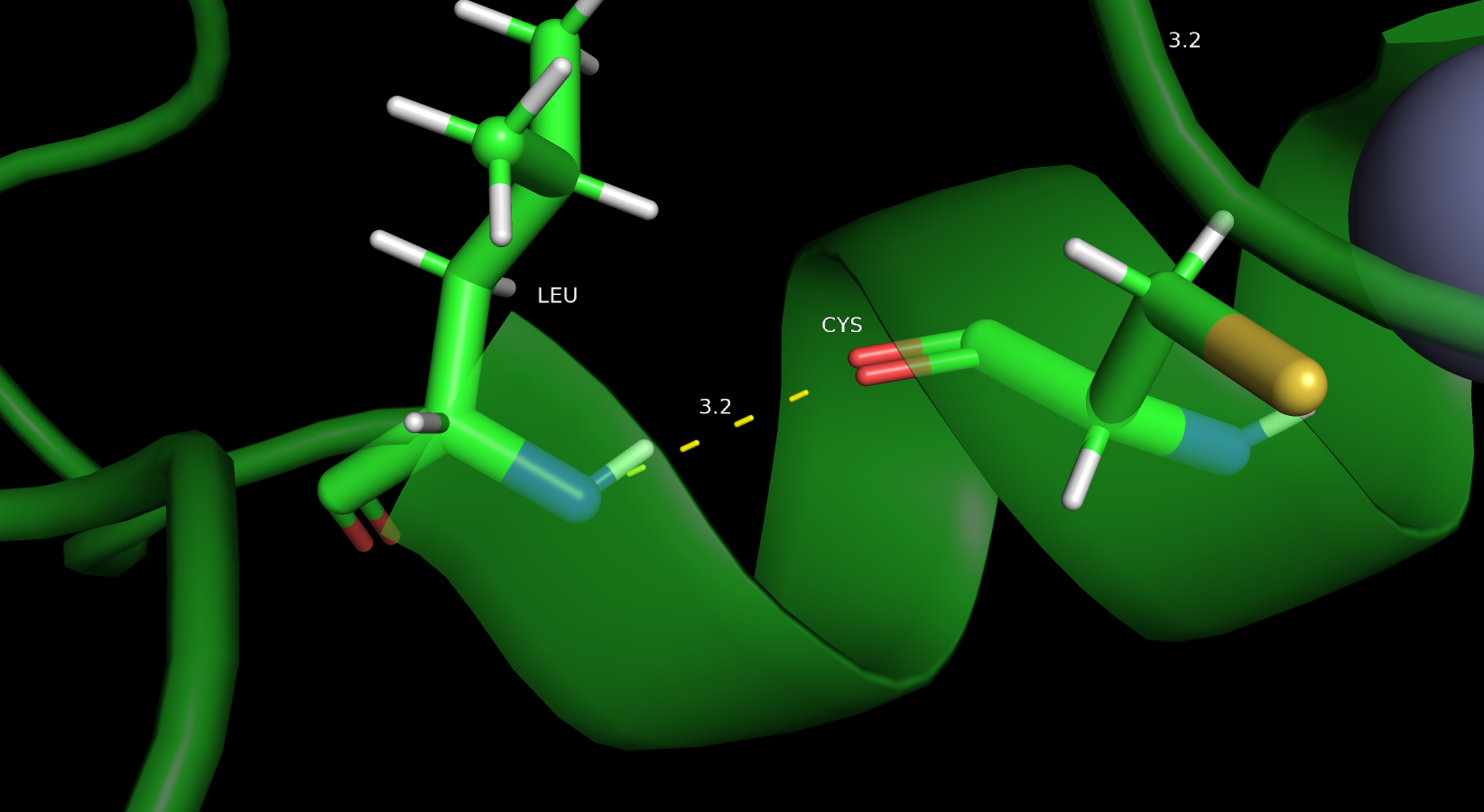
Водородная связь между атомами остова в ядре белка 6M2C. У домена, связи в котором я выбираю только одна небольшая спираль и два бета-листа и все они на повехности . Выбрал контакт в спирали со стороны ядра
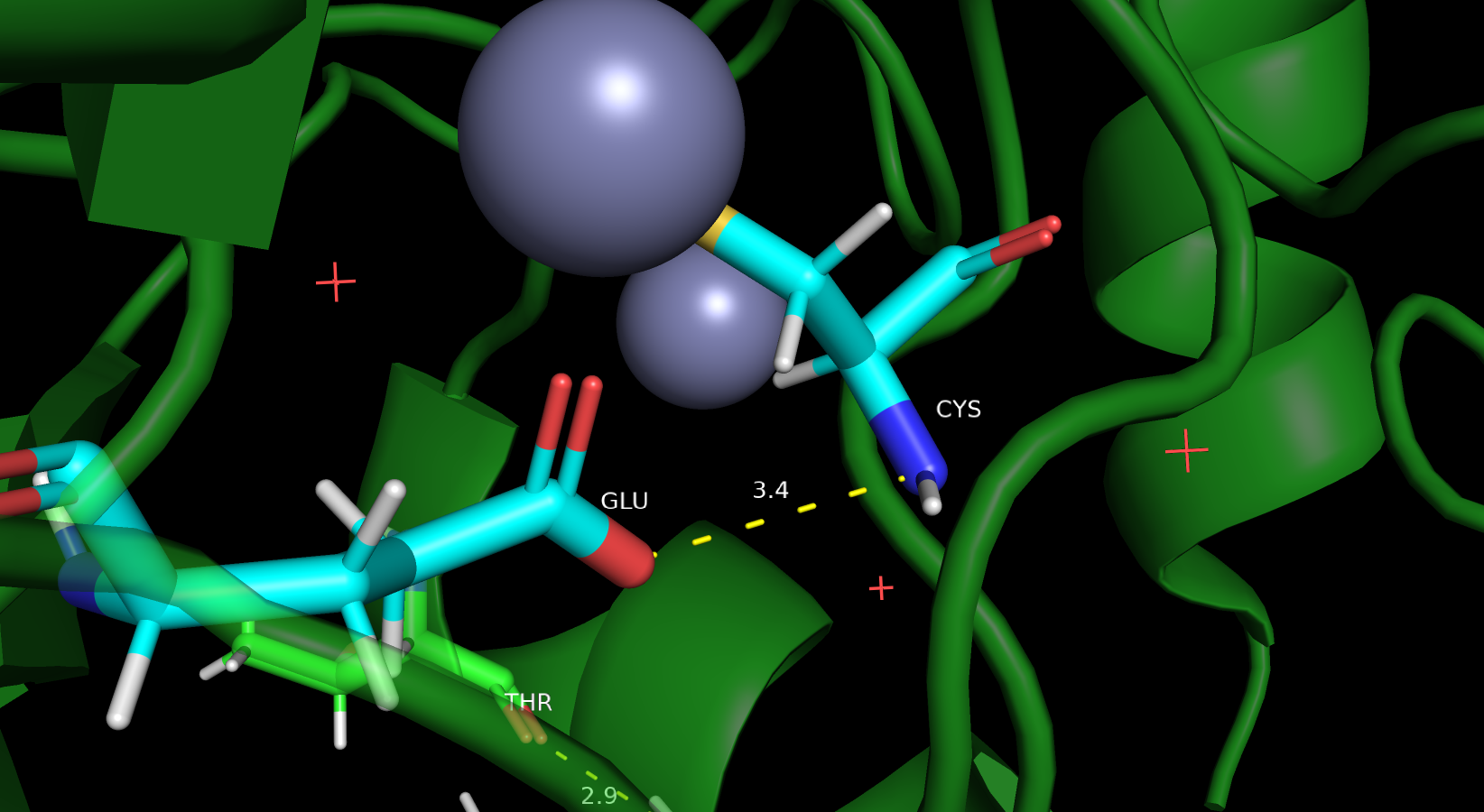
Водородная связь боковых цепей в ядре белка 6M2C. Не удалось найти связь между двумя боковыми цепями аминокислот, выбрал связь между боковой цепью и остовом. Что интересно, оказалось, что эта связь есть во всех 20 моделях ЯМР, но в РСА модели глутамат смотрел в противоположную сторону. Можно предположить, что эта связь по какой-то причиние устойчива в растворе, но не в кристалле
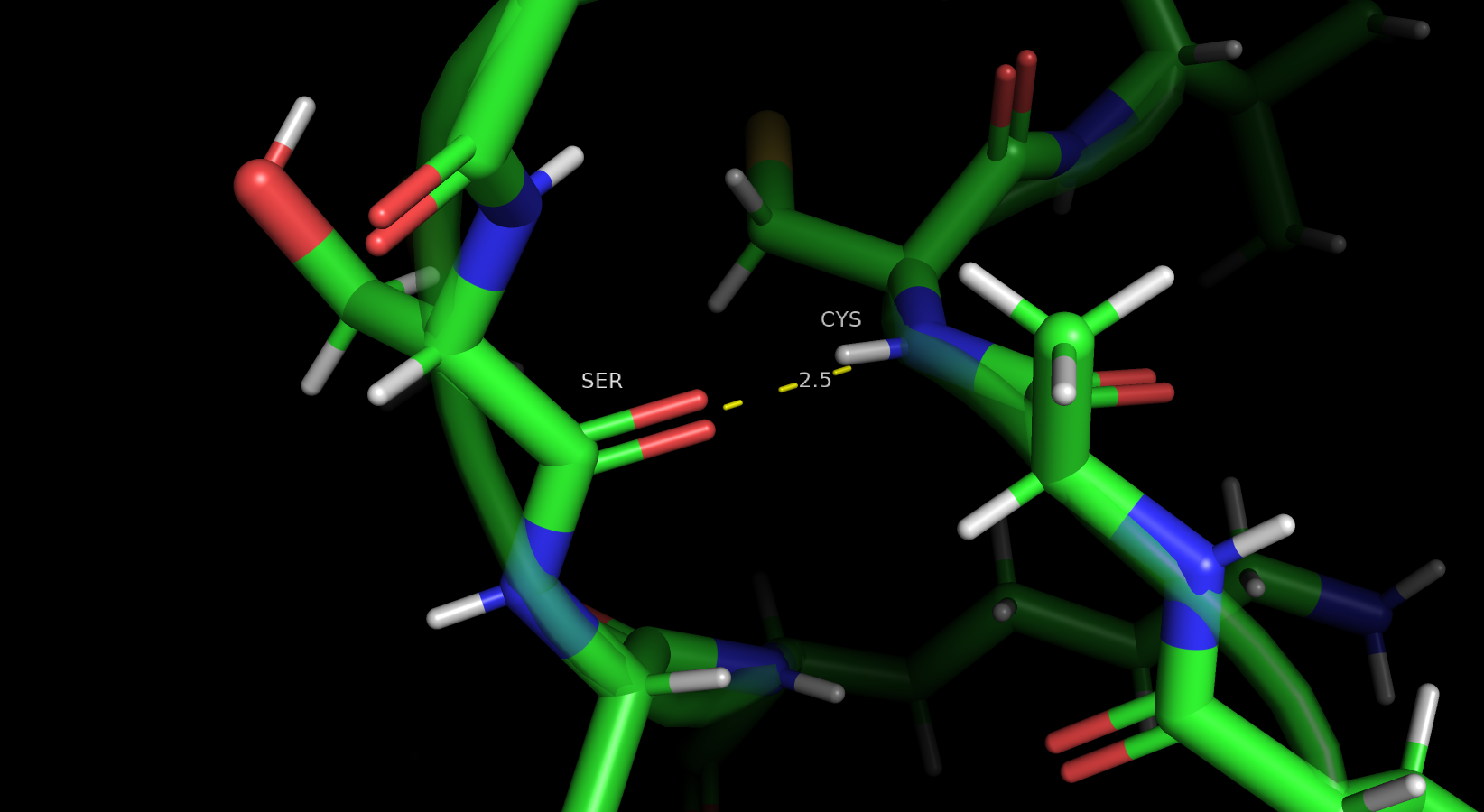
Водородная связь в петле, выходящей на поверхность глобулы 6M2C. Выбрал связь между остовами.
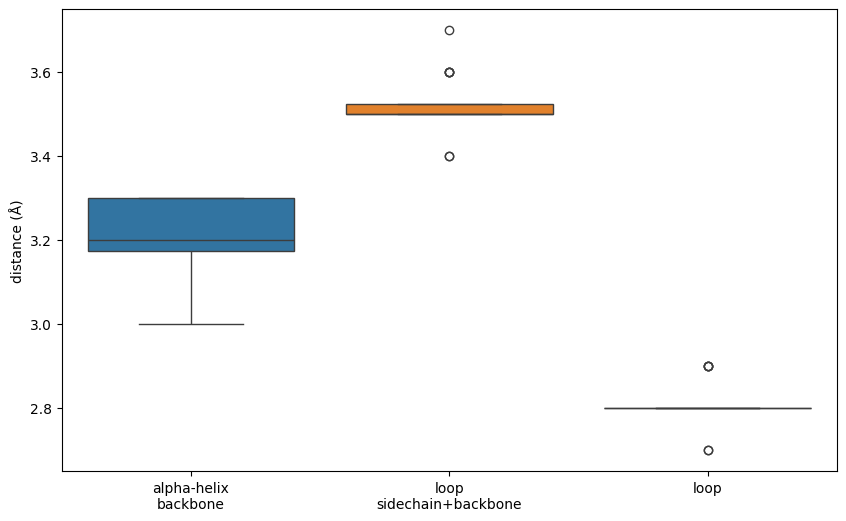
Результат анализа 20 моделей для ЯМР-структуры приведен в следующей таблице:
| Leu330-Cys326 | Glu316 – Cys336 | Ser308 – Cys302 | |
|---|---|---|---|
| Атомы | N—O | N-OE | N—O |
| Вторичная структура | α-спираль | — | — |
| На поверхности | - | - | + |
| Расстояние РСА | 3.2 | 13.1 | 2.5 |
| Доля моделей ЯМР со связью | 100% | 100% | 100% |
| Медиана (ЯМР) | 3.2 | 3.5 | 2.8 |
| Минимальное (ЯМР) | 3.0 | 3.4 | 2.7 |
| Максимальное (ЯМР) | 3.3 | 3.7 | 2.9 |
В теории я должно было наблюдаться увеличение отклонения в расстоянии между донором и акцептором от первой выбранной водородной связи к третьей. Но получилось, что третья связь, которая в петле, наоборот, почти во всех моделях имело одинаковую длину, а первая связь, образующая альфа-спираль в половине моделе отличалась на 0.1-0.2 ангстрема. Полагаю, это потому что единственная альфа-спираль, из которой я выбрал связь сама по себе находится на поверхности белка.
Задание 2
Чтобы примерно понять, в какой мере ансамбль моделей в записи PDB, полученной методом ЯМР, действительно можно принять за отражение подвижности белка, возьму для сравнения значения B-факторов из модели РСА и построю график зависимости одного от другого. Чтобы значения в двух массивах попарно соответствовали одному и тому же остатку, в модели РСА взял только цепь Н
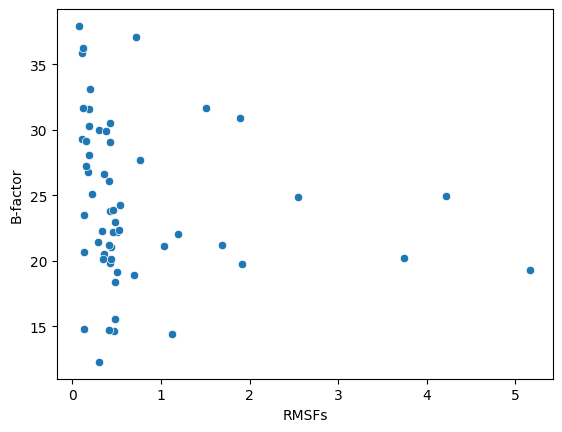
Значимой корреляции нет, получается в данном случае диапазон ЯМР структур не может отражать подвижность белка. В ноутбуке отдельно проверил, что сравниваемые последовательности совпадают.
Ссылка на ноутбук с кодом: task2.ipynb