Задание 1
Сперва посмотрел 1y0l на сайте PDB, это структура каталитического элиминирующего антитела 34E4 в комплексе с гаптеном. Это антитело, которое способно не только связываться с антигеном, но и катализировать его разрушение (элиминацию). Комплекс с гаптеном позволяет изучить механизм каталитической активности антитела, так как связывание позволяет стабилизировать его активную форму. В PDB нумерация аминокислотных остатков в антителах нередко меняется, на всякий случай лучше иметь копии .pdb файлов локально и работать с ними, поэтому в PyMOL загружаем файл не методом fetch, а версию файла с kodomo с помощью load
load "https://vsb.fbb.msu.ru/share/sbinf2022/2022.T1/1y0l.pdb"
# Лиганд имеет 3-буквенный код НAN, отделим его от загруженного белка, поменяем цвет на красный (контраст с остатками окружения, которые у меня будут зелеными)
select ligand, resn HAN
create lig, ligand
util.cba(12,"lig",_self=cmd)
remove ligand and 1y0lЛиганд окружен антипараллельными β-листами.

Сперва выделяем аминокислотные остатки, которые потенциально могут взаимодействовать с лигандом. Обычно рассматривается радиус в пределах 4-6 Å, это позволяет захватить все значимые нековалентные взаимодействия [1] [2]
create nearby_residues, byres 1y0l within 6 of lig
hide cartoon
show_as sticks, nearby_residues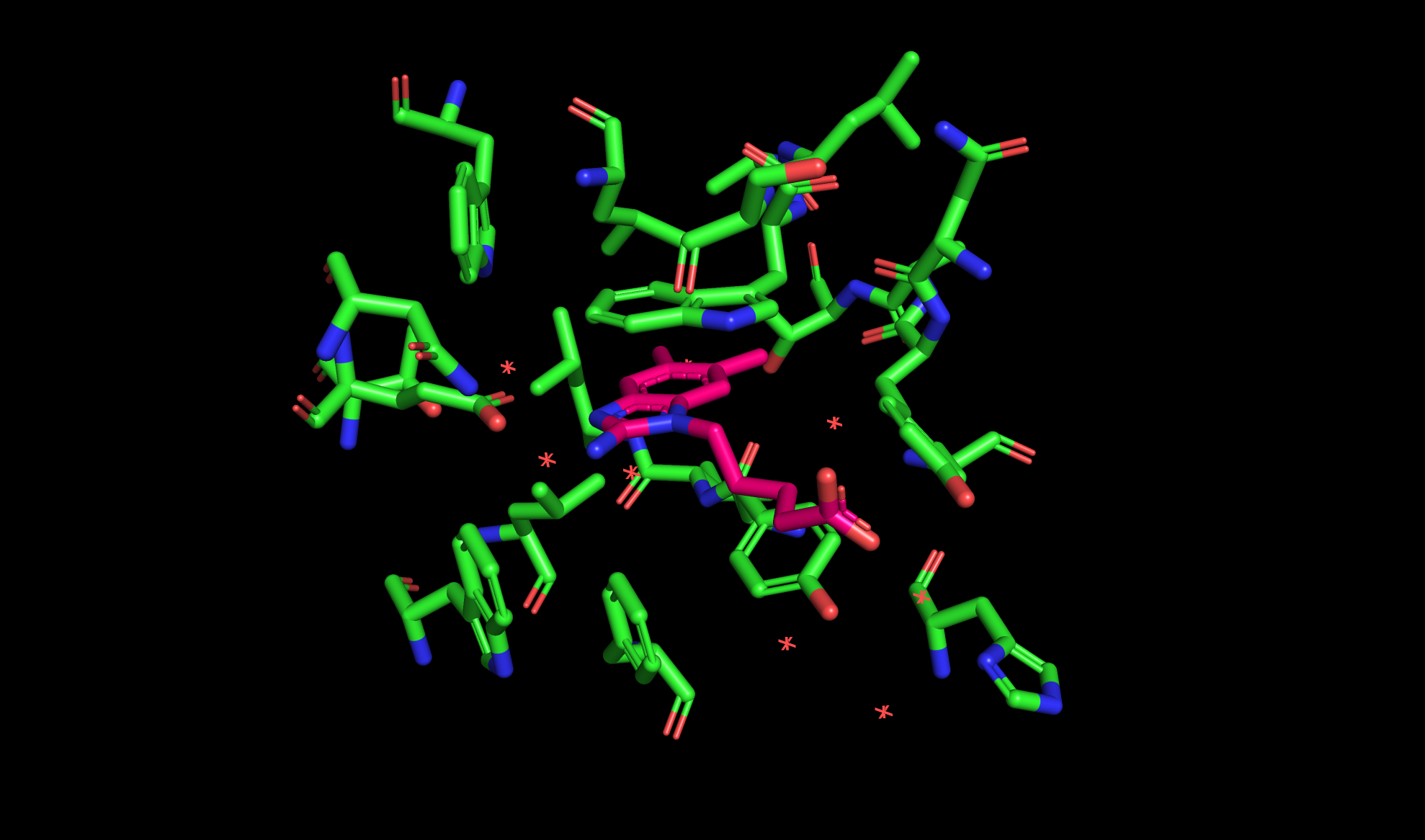
Видим две водородных связи с глутаматом и два стекинга между пятичленным кольцом лиганда с Trp91 и с Tyr100. Еще можно заметить, что две метильные группы шестичленного кольца лиганда окружены гидрофобными остатками (лейцин, валин..). Вода, судя по всему, не участвует во вз-иях, скрываем её для удобства обзора.
wizard measurement
cmd.get_wizard().set_mode("pairs")
dist hb1, /lig/J/A/HAN`601/N3, /nearby_residues/D/B/GLU`50/OE1, mode=0
dist hb2, /lig/J/A/HAN`601/N12, /nearby_residues/D/B/GLU`50/OE2, mode=0
# стекинг вручную указал
hide everything, solvent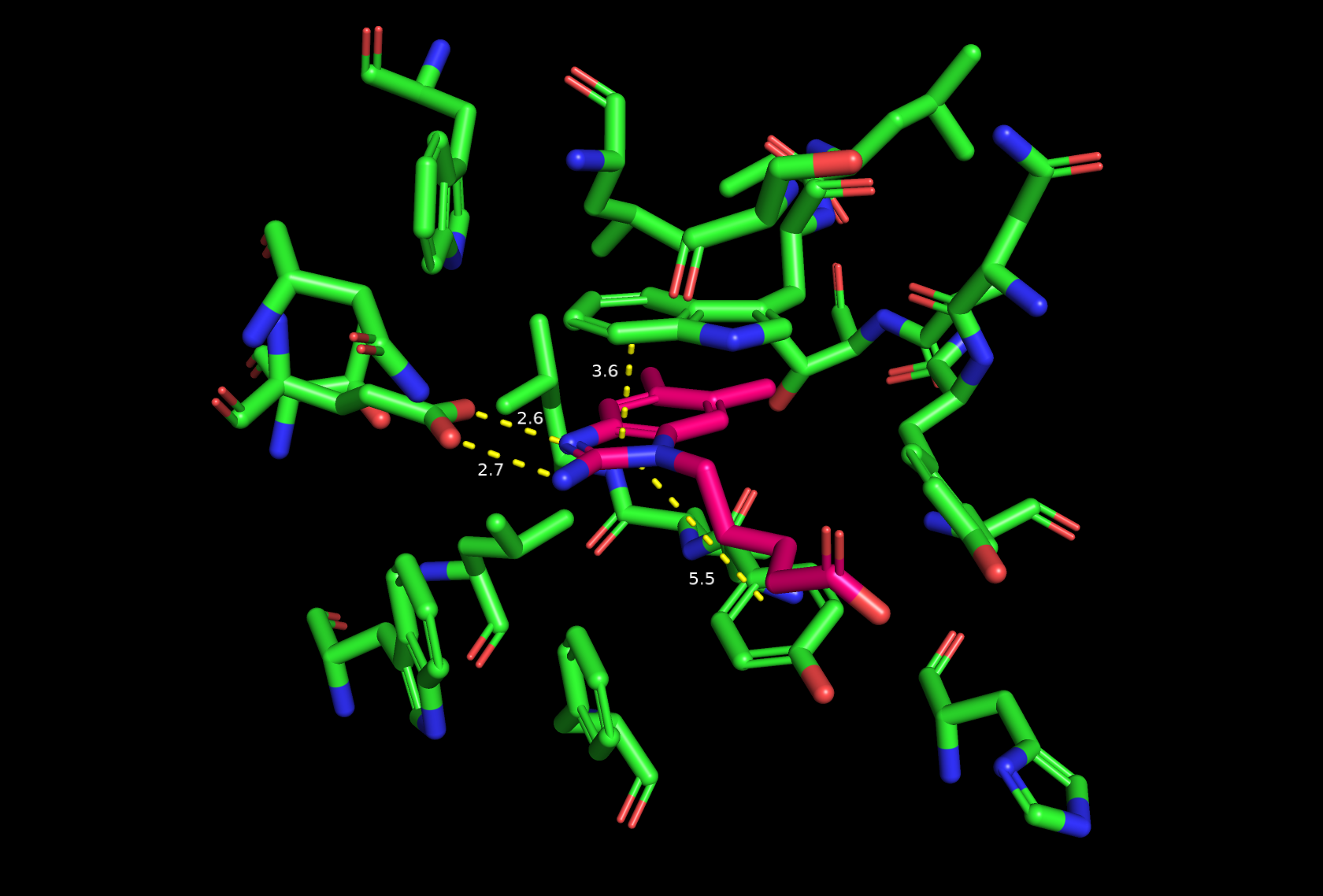
Пунктирные линии обозначают нековалентные взаимодействия.
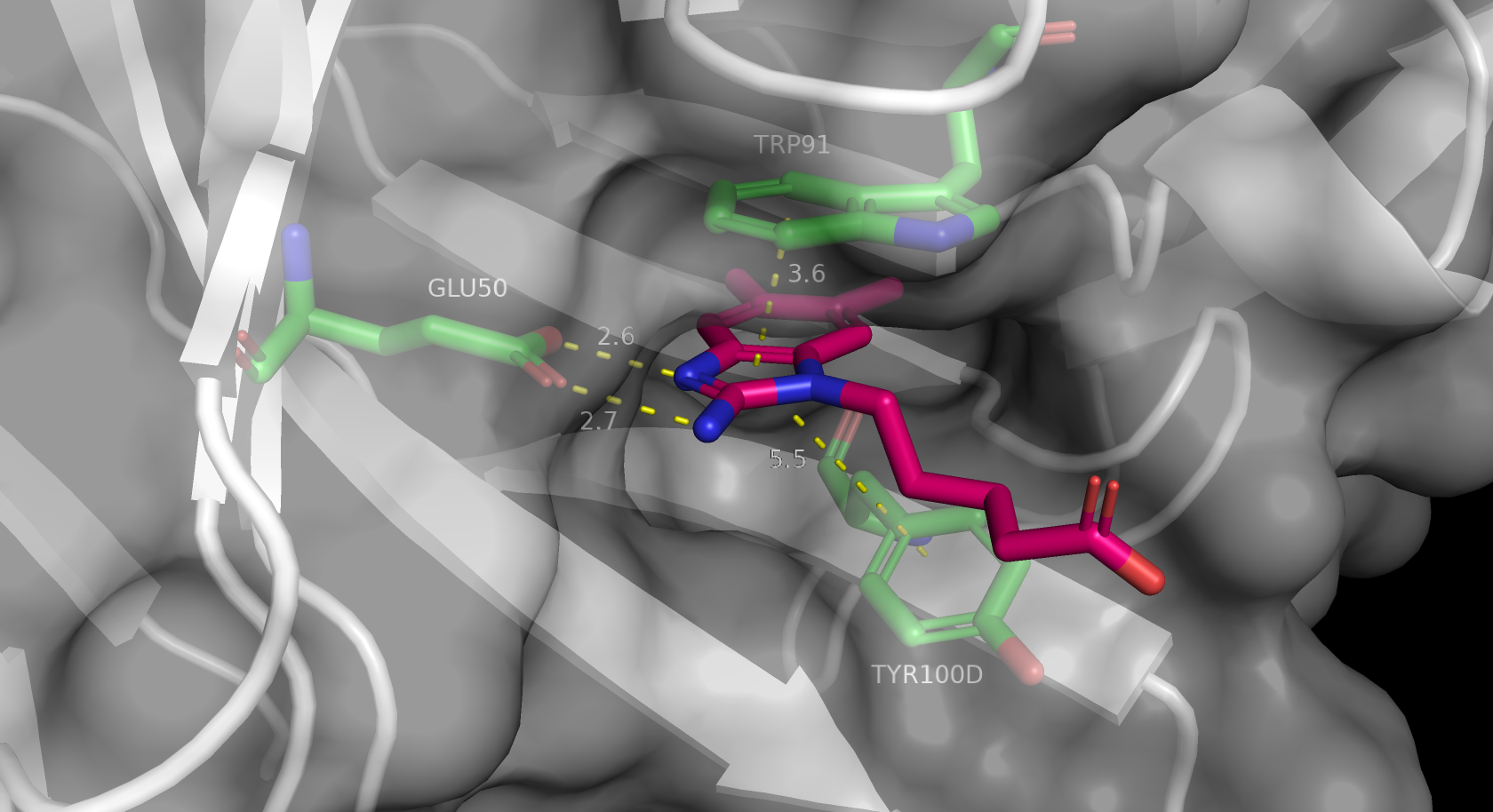
Теперь скроем не участвующие во взаимодействиях остатки, добавим полупрозрачное изображение поверхности антитела и силуэт вторичной структуры в месте связывания.
create interact_res, (resi 91 & chain A | resi 50+100D & chain B) & nearby_residues
show_as sticks, interact_res
show_as surface, 1y0l
cmd.color_deep("white", '1y0l', 0)
set transparency, 0.4
sele alpha_C, name CA and interact_res
label alpha_C, "%s%s" % (resn, resi)
set label_size, -0.4
# перерисовываю кратность связей COOH группы глутамата, чтобы на рисунке доноры с акцепторами подходили друг другу
unbond /interact_res/D/B/GLU`50/OE2, /interact_res/D/B/GLU`50/CD
bond /interact_res/D/B/GLU`50/OE2, /interact_res/D/B/GLU`50/CD, order=2
unbond /interact_res/D/B/GLU`50/OE1, /interact_res/D/B/GLU`50/CD
bond /interact_res/D/B/GLU`50/OE1, /interact_res/D/B/GLU`50/CD, order=1
Задание 2
fetch 1y0l, type=2fofc
isomesh eDens, 1y0l_2fofc, 1, lig or interact_res, carve=2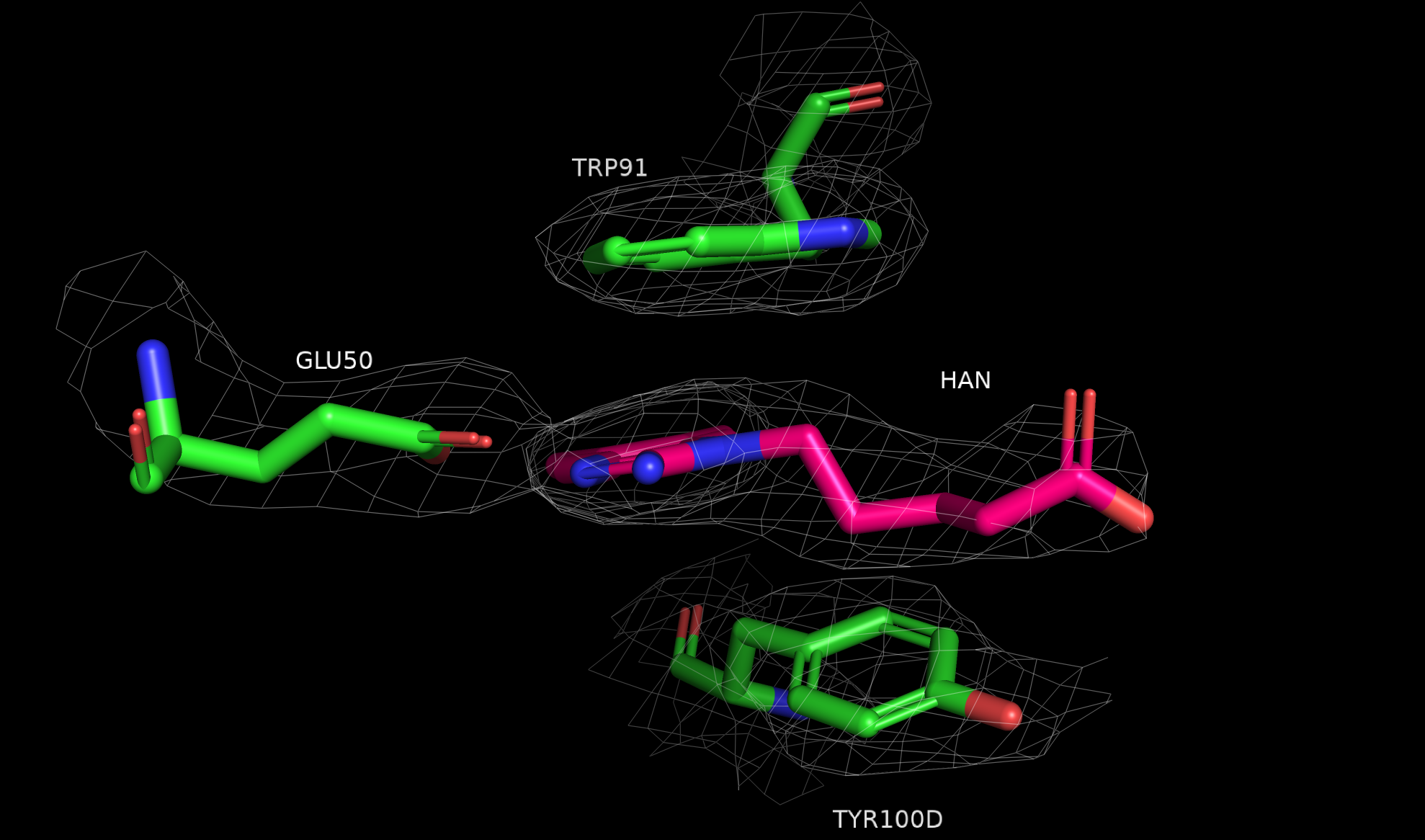
На уровне подрезки 1, значимой разницы между покрытием электронной плотностью лиганда и аминокислоты белка нет (часть глутамата не покрыта просто потому что в край ячейки попала).