1.AC:pf07992; ID:Pyr_redox_2; название: Pyridine nucleotide-disulphide oxidoreductase; число последовательностей: 412,453.
2.AC: pf14721; ID: AIF_C; название: Apoptosis-inducing factor, mitochondrion-associated, C-term; число последовательностей: 442. .
3.Архитектура: ID:L8DIH5_9NOCA; AC: L8DIH5; название: Uncharacterized NAD(FAD)-dependent dehydrogenase; число последовательностей: 294 в Pfam и 436 в UniProt. .
Белки полученной выборки использовались в построении HMM профиля. Последовательности белков из выборки были выравнены в программе JalView с помощью Muscle. Выравнивание было обработано и сохранено в формате fasta. С помощью команд "hmm2build pr9.hmm pr9.fasta" и "hmm2calibrate pr9.hmm" hmm профиль был построен и откалиброван. Затем из базы данных uniprot были получены последовательности 1253 белков в fasta формате. С помощью команды " hmm2search -E 0.1 -T 0 pr9.hmm uniprot-PF14721.fasta.gz > hmm_results.fasta " был совершен поиск по этим белкам. Были взяты последовательности с e-Value ниже 0.1 и они были проанализированы в excel. Были получены графики score и кривая ROC.
Ссылка на выравнивание Ссылка на результат hmm2search Ссылка на таблицу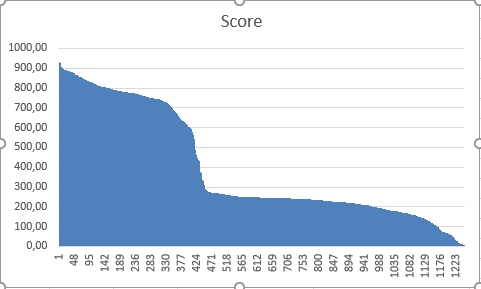
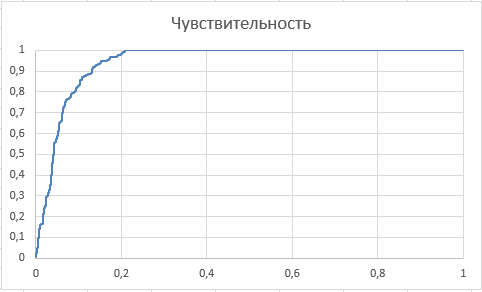
Было посчитано максимальное значение F1-score: 0,711297 . Ему соответствовала 287 позиция в таблице В результате был определён оптимальным порог score по соотношению чувствительности и точности: 749.30 Также все найденные последовательности с нашей архитектурой были приняты тестом. Это говорит о высокой консервативности и, возможно, специфичности данной архитектуры.