Определение функции и таксономии нуклеотидной последовательности
Для выполнения этого задания была использована нуклеотидная последовательность, полученная из хроматограммы в 6 практикуме: СсылкаДля определения того, кому может принадлежать эта последовательность, необходимо было воспользоваться сервисом BLAST.
Нужно было выбрать, по какому из алгоритмов blastn начать поиск. Так как мы ничего не знаем о том, какие функции выполняет эта последовательность и кому она принадлежать(и консервативная ли она), я не вижу смысла использовать megablast. Именно из-за этого я решил использовать blastn. Параметры запуска я предпочел оставить дефолтными:
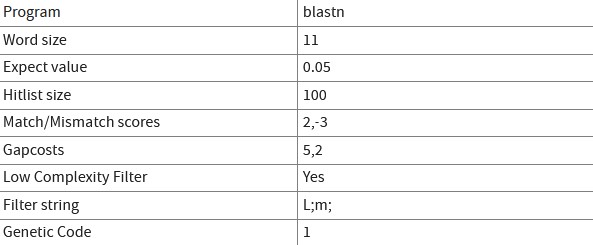
Выдача:
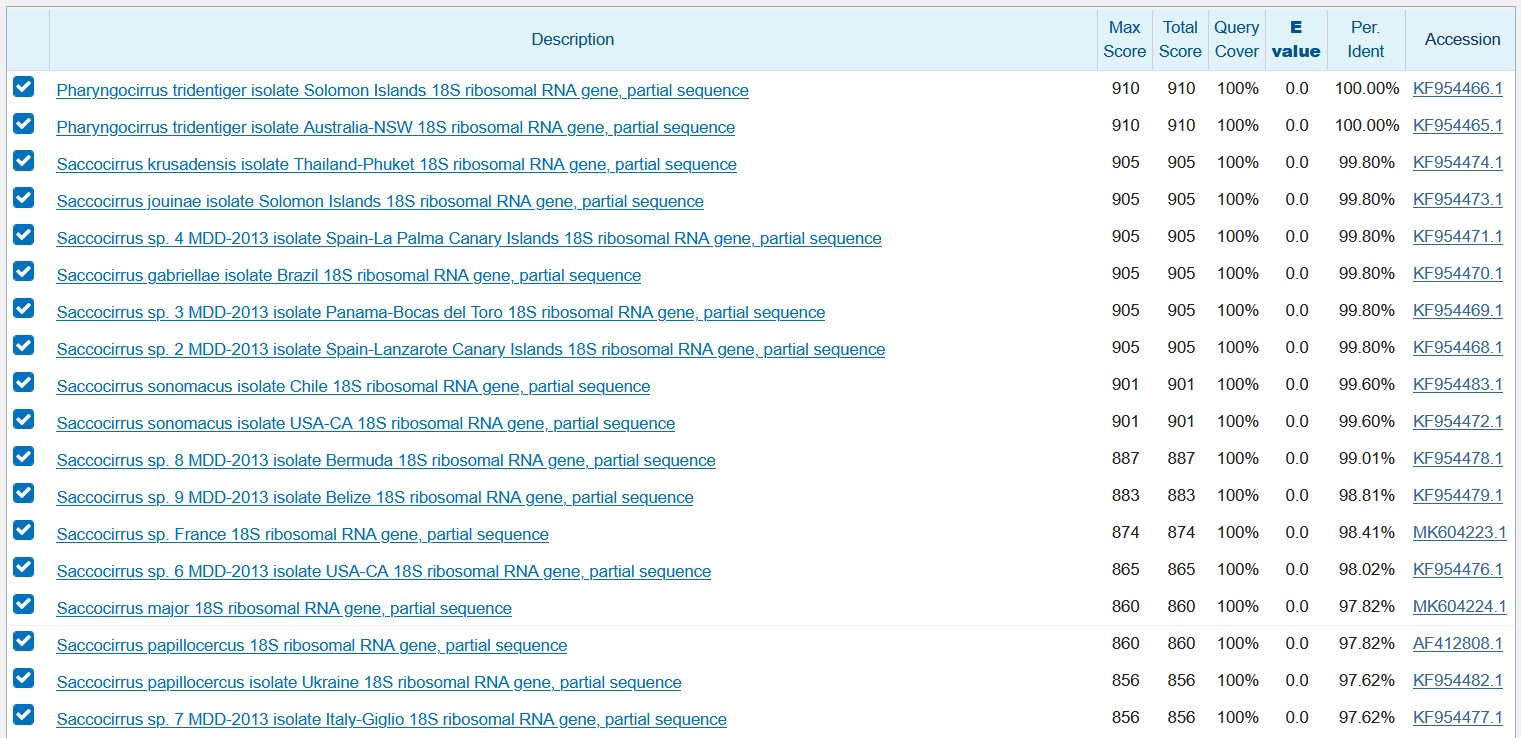
Я посчитал, что для аннотации достаточно первых 8 находок.
Судя по выдаче, консенсусная последовательность с большой вероятностью принадлежит Pharyngocirrus tridentiger и является частью гена, кодирующего 18S рРНК. Я поинтересовался, к какому типу рибосом она принадлежит, и с помощью википедии узнал, что 18S rRNA образует вместе с белками малую субъединицу 80S рибосомы (встречается, как я знаю, только у эукариот). Pharyngocirrus tridentiger - принадлежит к многощетинковым червям(полихетам).
Ближайшие находки (Saccocirrus krusadensis и Saccocirrus jouinae) также принадлежат к Polychaeta, и даже принадлежат к одному семейству - Saccocirridae.
Первые 8 находок, полученные с помощью BLAST
Поиск генов белков в неаннотированной нуклеотидной последовательности
Я взял контиг Gallus gallus (Красная джунглевая курица) Ссылка на контигФотография Gallus gallus, взятая из википедии:

Я провёл поиск по базе данных swissprot. Это было сделано из соображений того, что там меньше белков и поиск пройдёт быстрее, а при малом числе находок можно провести поиск по Non-reduntant protein sequences. Но, как мне показалось, выдача достаточно достоверная и с достаточным числом белков. Выдачу Blastx вы можете посмотреть, перейдя по ссылке
Рассмотрев выдачу Blast, можно сделать предположение, что в контиге содержался ген, или участок гена (скорее всего это целый ген , так как в Uniprot написано, что этот белок имеет в своем составе около 420 аминокислот, а выравнивания blastx проиходили по длине 415-420 аминокислот), кодирующий белок нуклеопорин P42.
Интепретация карты локального сходства гомологичных хромосом двух бактерий
Необходимо было выбрать геномы 2 бактерий, принадлежащих к одному роду, но разных видов. Я взялгеномы Alteromonas australica и Alteromonas naphthalenivorans.
AC записей: NZ_CP008849 и NC_015554 соответственно.
Карта локального сходства, полученная при сравнении геномов:
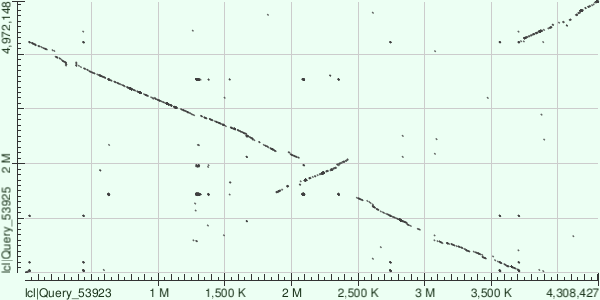
Достаточно интересный результат. Получить что-то интересное вышло не сразу, но всё же повезло.
На карте достаточно хорошо видно, что произошло минимум две инверсии(по координатам: 2M-2,5M и 3,8M-4,3M относительно бактерии "по оси X") . Следует отметить, что разрыв на карте не играет большой роли в сравнении хромосом, так как они кольцевые, а не линейные. Гораздо важнее, что этот "разрыв" повернут на 180 градусов относительно того, что мы могли бы ожидать.
Меня заинтересовала инверсия "посередине" карты:

Дело в том, что здесь, как я думаю, было минимум 2 инверсии, причем они могли пройти 2 разными путями(зелёная и синяя линии на картинке ниже). В случае, отмеченном зелёным, произошло 2 инверсии на 2 близких участках, тогда как синим отмечена ситуация, когда произошла инверсия на большом участке(≈500кб), и также имела место быть инверсия на меньшем участке внутри большого. Причём во втором случае нельзя сказать (по крайней мере я не смогу точно утверждать) какая из инверсий была первее.
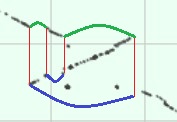
Результат, полученный при сранении, кажется достаточно интересным.