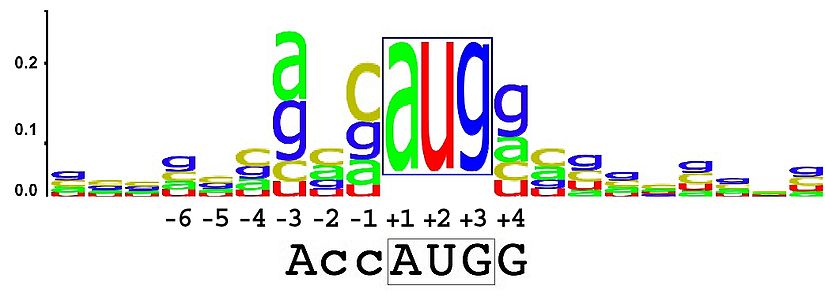Задание 2
Я взял 7 вариант задания. Была построена таблица количества букв для каждой позиции в выравнии, затем по ней таблица частот букв. По ней была создана таблица IC для каждой буквы. Далее были просуммированны IC для столбцов, а затем все столбцы, чтобы вычислить итоговое значение IC. Формула для вычисления IC для каждой ячейки таблицы: IC(b, j) = f(b,j) * log2(f(b,j)/p(b)) ТаблицаТакже было построено LOGO при помощи сервиса

Задание 3
В прошлом практикуме при помощи МЕМЕ я искал сайты регуляции разрывной транскрипции в геноме коронавируса Scotophilus bat coronavirus 512. Наиболее правдоподобным, на мой взгляд, оказался мотив, представленный ниже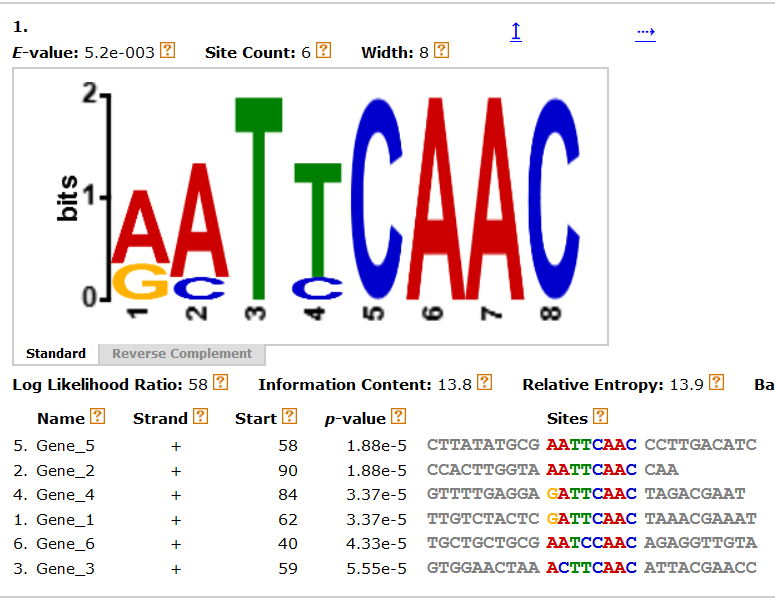 . PWM, соответствующую этому мотиву, я загрузил в программу FIMO. Результат её работы представлен ниже:
. PWM, соответствующую этому мотиву, я загрузил в программу FIMO. Результат её работы представлен ниже: 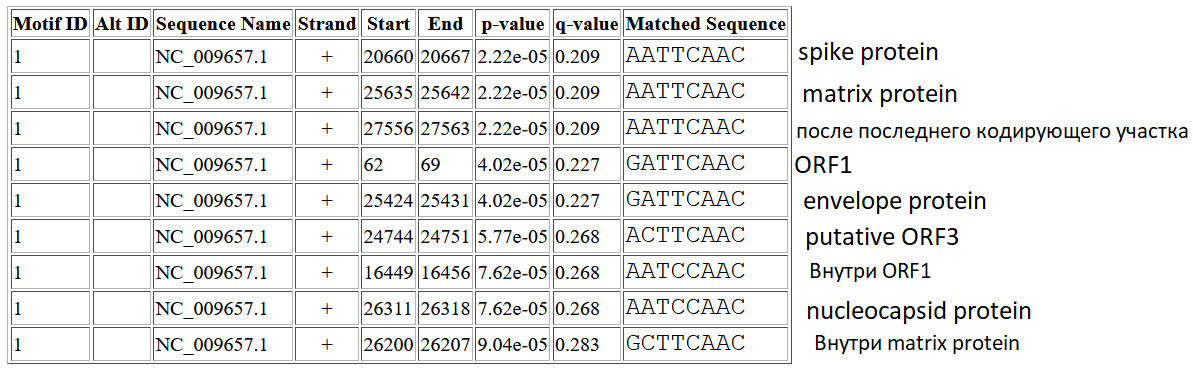 В выдаче было 9 находок, 6 из которых располагались перед генами. 2 другие находки попали внутрь других генов, и 1 располагалась после последней из кодирующих последовательностей. Найденный мотив имеет самую
консервативную часть длиной 6 нуклеотидов (TTCAAC) (Core Sequence). До начала гена ORF1 полипротеина есть ровно 1 сигнал. Перед каждым из поздних генов на участке -100 - -1 от старта трансляции есть ровно 1 сигнал.
Вероятность случайно найти мотив с такими свойствами мала (хорошие значения p-value).
В выдаче было 9 находок, 6 из которых располагались перед генами. 2 другие находки попали внутрь других генов, и 1 располагалась после последней из кодирующих последовательностей. Найденный мотив имеет самую
консервативную часть длиной 6 нуклеотидов (TTCAAC) (Core Sequence). До начала гена ORF1 полипротеина есть ровно 1 сигнал. Перед каждым из поздних генов на участке -100 - -1 от старта трансляции есть ровно 1 сигнал.
Вероятность случайно найти мотив с такими свойствами мала (хорошие значения p-value).
Поиск сигнала в близкородственных видах
К сожалению, в ncbi отсутствуют геномы других штаммов этого коронавируса, как и геномы вирусов, принадлежащих к тому же подроду, поэтому я взял два других коронавируса рода Alphacoronavirus. В качестве первого я выбрал Alphacoronavirus Bat-CoV/P.kuhlii/Italy/3398-19/2015, выдача FIMO представлена ниже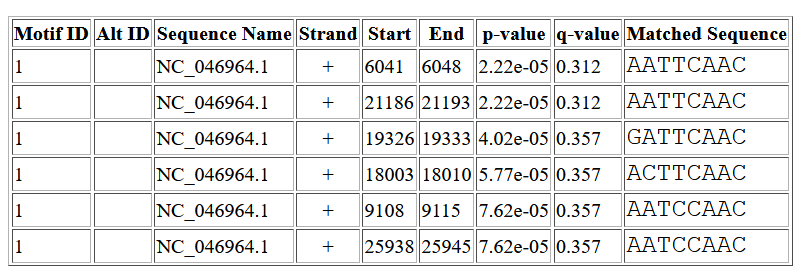 Найденные мотивы не расположены в upstream областях,
данный мотив не встречается перед геном, кодирующим полипептид, поэтому я не думаю, что в данном вирусе он регулирует разрывную транскрипцию. В качестве второго представителя рода Alphacoronavirus
я выбрал Rousettus bat coronavirus HKU10. Выдача FIMO представлена ниже:
Найденные мотивы не расположены в upstream областях,
данный мотив не встречается перед геном, кодирующим полипептид, поэтому я не думаю, что в данном вирусе он регулирует разрывную транскрипцию. В качестве второго представителя рода Alphacoronavirus
я выбрал Rousettus bat coronavirus HKU10. Выдача FIMO представлена ниже: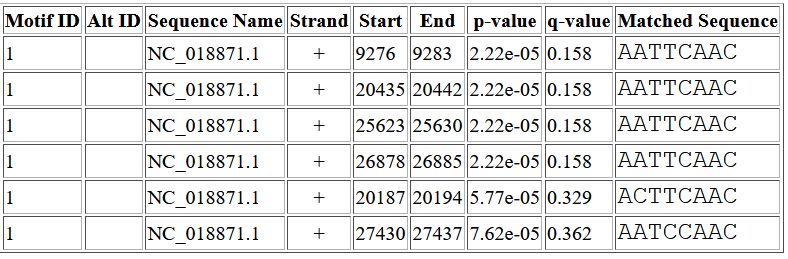 . Для этого вируса найденные мотивы так же не расположены в upstream областях генов,
и данного мотива нет перед геном полипептида, поэтому я не думаю что для этого вируса он регулирует разрывную транскрипцию. Таким образом, данный мотив скорее всего не является специфичным для рода,
а для более близких родственников мы не можем делать выводов, так как в ncbi отсутствуют такие геномы.
. Для этого вируса найденные мотивы так же не расположены в upstream областях генов,
и данного мотива нет перед геном полипептида, поэтому я не думаю что для этого вируса он регулирует разрывную транскрипцию. Таким образом, данный мотив скорее всего не является специфичным для рода,
а для более близких родственников мы не можем делать выводов, так как в ncbi отсутствуют такие геномы.
LOGO для последовательностей Козак поздних генов
Для построения LOGO я вырезал участки генома расположенные на +-5 нуклеотидов он старт-кодона (с позиций -5 до 8 от начала генов), и затем воспользовался сервисом MEME. Если сравнивать его с LOGO для последовательностей Козак генов человека, то окажется, как и у человека в позиции -3 у вируса чаще встречается аденин.
Если сравнивать его с LOGO для последовательностей Козак генов человека, то окажется, как и у человека в позиции -3 у вируса чаще встречается аденин.