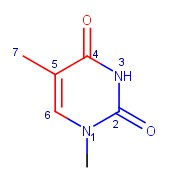
Рисунок 1. Тимин
В сторону большой бороздки обращены атомы: C4, O4, C5, C7, C6, H6.
В сторону малой бороздки обращены атомы: N1, C2, O2, N3, H3.
Соответственно заданию были построены содели структур А, В и Z форм ДНК с помощью инструментов пакета 3DNA. Последовательности молекул состоят из 20 пар нуклеотидов, которые были заданы следующим образом: для А и В форм это 5 раз повторенные куски "GATC", для Z-формы это 10 раз повторенный участок "GC", так как иных вариантов последовательности для этой формы в данном пакете мне сделать не удалось.
Ссылки на файлы: А-форма, B-форма, Z-форма.
 Рисунок 1. Тимин |
Полученная модель В-формы была открыта с помощью JMol. Требовалось определить для заданного азотистого основания (в моем случае -тимина), какие атомы смотрят в большую, а какие - в малую бороздку.
Результат можно увидеть на рисунке 1: на нем красным цветом выделены атомы, смотрящие в сторону большой бороздки, а синим - в сторону малой. Также это записано в виде списка, где
атомы тимина пронумерованы согласно файлу gatc-b.pdb, в котором записана структура этой модели:
В сторону большой бороздки обращены атомы: C4, O4, C5, C7, C6, H6. В сторону малой бороздки обращены атомы: N1, C2, O2, N3, H3. |
В этом упражнении требовалось сравнить основные спиральные параметры разных форм ДНК и занести результаты в таблицу - см. таблицу 1.
| А-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (А) | 28,03 | 33,75 | 43,50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7,98 [DC]32:B.P #638 - [DT]3:A.P #44 | 17,21 [DA]26:B.P #515 - [DC]12:A.P #228 | 18,30 [DC]32:B.P #638 - [DC]6:A.P #105 |
| Ширина малой бороздки | 16,81 [DG]13:A.P #247 - [DA]30:B.P #597 | 11,69 [DT]35:B.P #700 - [DA]10:A.P #187 | 9,87 [DC]32:B.P #638 - [DG]13:A.P #247 7,20 [DG]33:B.P #657 - [DG]11:A.P #206 |
Таблица 1. Спиральные параметры форм ДНК
Для наглядности на рисунках 2 -7 представлены эти формы с измеряемыми параметрами. Зеленым выделены атомы фосфора, фиолетовым и красным покрашены разные цепи.
 |  |  |  | |
| Рисунок 2. Шаг спирали, А-форма | Рисунок 3. Ширина бороздок, А-форма | Рисунок 4. Шаг спирали, В-форма | Рисунок 5. Ширина бороздок, В-форма | |
 |  |  Рисунок 7В. Расстояния от цитозина  Рисунок 8В. Расстояние от гуанина |  | |
| Рисунок 6. Шаг спирали, Z-форма | Рисунок 7A. Ширина бороздок, Z-форма (при измерении от цитозина) | Рисунок 8А. Ширина бороздок, Z-форма (при измерении от гуанина) |
Ширина бороздок искалась как локальный минимум в ряду расстояний от одного фосфата для ближайших фосфатов комплементарной цепи.
Интересна ситуация с Z-формой. Дело в том, что в ней различаются эти значения при измерении расстояний от гуанина (рисунки 8) и от цитозина (рисунки 7).
При этом если мерить от цитозина, то удается засечь обе бороздки, если же от гуанина - то только малую (7,20А), потому что значение 0,868 слишком близко к малой бороздке и возникает не из-за перехода в большую бороздку, а из-за зигзагообразной структуры Z-формы (такой же близкий локальный минимум есть и при измерении от цитозина - 11,58А).
| Для этого задания мне были заданы две структуры: tRNA (PDB ID: 2DLC) и DNA (PDB ID: 1HDD). С помощью программ программ find_pair и analyze были получены данные о торсионных углах этих структур (на рисунке 9 показано, какие обозначения углов каким углам в реальности соответствуют). Эти программы дали и другие данные, которыми использовала при выполнении упражнений, а также сами файлы, полученные с помощью программ, можно посмотреть по ссылке (оригинальные данные про торсионные углы содержаться там в таблицах 1 и 6): Далее с помощью Exel были высчитаны средние значения углов и найдены отклонения от этих значений для каждого нуклеотида (из тех, что образуют пары). Отклонение считалось как модуль разности значения угла для данного нуклеотида и среднего значения. В таблицу 2 были занесены эти результаты с соответствующими нуклеотидами, а так же нуклеотид (один на каждую цепь), у которого была максимальна сумма отклонений по углам. |  |
| Значения всех отклонений и всех сумм октлонений можно найти все по той же ссылке в таблицах 2, 3 и 7,8. | Рисунок 9. Торсионные углы нуклеотида |
| tRNA | |||||||
|---|---|---|---|---|---|---|---|
| α | β | γ | δ | ε | ζ | χ | |
| Strand I | |||||||
| средние значения | -42,9 | 57,9 | 50,8 | 85,1 | -136,8 | -64,9 | -166,1 |
| максимально отличающиеся значения | 168,9 | -177,0 | -151,6 | 139,6 | 0,0 | 0,0 | -76,8 |
| модуль отклонения | 211,8 | 234,9 | 202,4 | 54,5 | 136,8 | 64,9 | 89,3 |
| соответствующие нуклеотиды | 555 P | 514 A | 506 G | 519 G | 519 G, 515 G | 519 G, 515 G | 519 G |
| Максимально непохожий нуклеотид - 506 G | |||||||
| Strand II | |||||||
| средние значение | -57,9 | 80,2 | 47,9 | 90,2 | -126,0 | -64,1 | -156,8 |
| максимально отличающиеся значения | 137,8 | -176,4 | -178,6 | 143,4 | 156,8 | 179,6 | -74,6 |
| модуль отклонения | 195,7 | 256,6 | 226,5 | 53,2 | 282,8 | 243,7 | 82,2 |
| соответствующие нуклеотиды | 523 G | 561 C | 523 G | 558 a | 528 A | 558 a | 558 a |
| Максимально непохожий нуклеотид - 523 G | |||||||
| DNA | |||||||
| Strand I | |||||||
| средние значение | -54,7 | 60,7 | 42,1 | 130,8 | -81,0 | -80,0 | -111,7 |
| максимально отличающиеся значения | 147,1 | -178,2 | -174,4 | 79,1 | 178,0 | 165,1 | -159,0 |
| модуль отклонения | 201,8 | 238,0 | 216,5 | 51,7 | 259,0 | 245,1 | 47,3 |
| соответствующие нуклеотиды | 21 A | 18 C | 21 A | 18 C | 18 C | 11 T | 7 C |
| Максимально непохожий нуклеотид - 506 G | |||||||
| Strand II | |||||||
| средние значение | -45,9 | 43,5 | 34,2 | 138,9 | -61,6 | -104,0 | -103,4 |
| максимально отличающиеся значения | 44,0 | -176,7 | -50,8 | 86,2 | 172,0 | 126,3 | -151,5 |
| модуль отклонения | 89,9 | 220,2 | 85,0 | 52,7 | 233,6 | 230,0 | 48,1 |
| соответствующие нуклеотиды | 40 A | 36 T | 40 A | 31 T | 28 T | 34 C | 31 T |
| Максимально непохожий нуклеотид - 506 G | |||||||
Таблица 2. Торсионные углы
Затем мной были так же получены данные о торсионных углах А, В и Z форм ДНК (структур, полученных в задании 1). Таблицы с углами можно найти все на той же странице с таблицами. Затем я посчитала среднее по всем углам (не считая украйних нуклеотидов) по обеим цепям вместе. Полученные значения занесены в таблицу 3.
| α | β | γ | δ | ε | ζ | χ | |
|---|---|---|---|---|---|---|---|
| A | -51,7 | 174,8 | 41,7 | 79,1 | -147,8 | -75,1 | -157,2 |
| B | -29,9 | 136,3 | 31,1 | 143,3 | -140,8 | -160,5 | -98,0 |
| Z | -43,8 | 21,1 | -61,5 | 116,3 | -100,1 | 8,6 | -47,8 |
| tRNA | -54,4 | 72,2 | 50,4 | 89,7 | -134,8 | -66,0 | -165,0 |
| DNA | -50,3 | 52,1 | 38,2 | 134,9 | -71,3 | -92,0 | -107,6 |
Таблица 3. Торсионные углы разных форма DNA и приведенных выше tRNA и DNA
По таблице 3 можно сказать, что приведенная структура tRNA больше всего похожа на A-форму ДНК (сумма модулей разностей по углам между ними минимальна), а выданная ДНК - на В-форму (посчитано все также по сумме модулей разности между углами).
Таблица 4. Пары | Всего в последовательности данной tRNA 76 нуклеотидов, которые нумеруются от 501 до 576:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
В этих 24 парах есть 8 неканонических (пара, в которой есть модифицированный нуклеотид, я считала автоматически неканонической):
Из этих пар 3 не участвуют в формировании стеблей (см рисунок 10, на нем эти пары покрашены желтым):
| 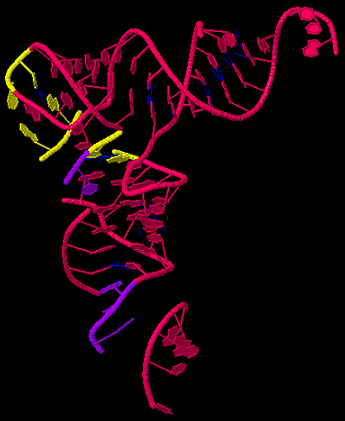 Рисунок 10. tRNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
В этом упражнении искались возможные стэкинг-взаимодействия. Данные о перекрывании азотистых оснований были в файле stacking.pdb - это один из файлов, полученных в задании 2 (см таблицу 9). Максимальная суммарная площадь - у пар tP/ga (структура 13): 14,01. С помощью комманд ex_str и stack2img выбранная структура была вырезана, далее полученный файл был конвертирован в формат png - см рисунок 11. Для сравнения я сделала все то же самое для структур с минимальной площадью (структура 14, площадь 0) и просто маленькой (структура 15, площадь 3,16) - см рисунки 13 и 12.
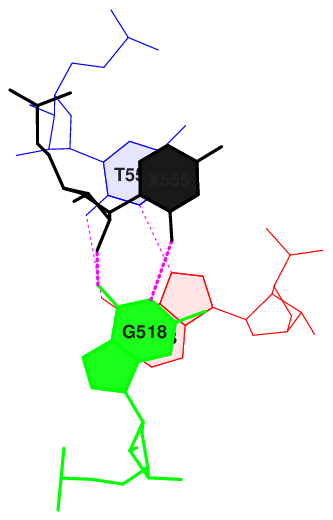 |  | 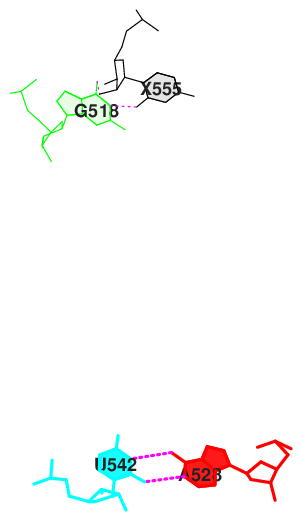 |
| Рисунок 11. Максимальное перекрывание | Рисунок 12. Низкое перекрывание | Рисунок 13. Минимальное перекрывание |
НАЗАД ➜ |
| © Рюмина Екатерина, 2017 |