



 |  |  |  |
Анализ крупных перестроек геномов |
Вернуться на страницу семестра Выравнивание геномовПоиск данных для выравнивания, результаты blast Нужно было выберать три генома бактерий или архей одного вида. В базе данных Genome NCBI ищем бактерии, у котороых есть сборка генома до хромосомы. Хромосомы должны быть собраны полностью, набор контигов или скэффолдов не годятся.
Я выбрала вид Paenibacillus polymyxa и 3 его штамма: SC2 (NC_014622.2), E681 (NC_014483.2), CR1 (NC_023037.2)
Таблица 1. Информация о выбранных штаммах
Программой blast2seq на NCBI были проведены 3 выравнивания геномов. Карты локального сходства приведены ниже. Рис.1. Карта локального сходства SC2 (NC_014622.2) и E681 (NC_014483.2)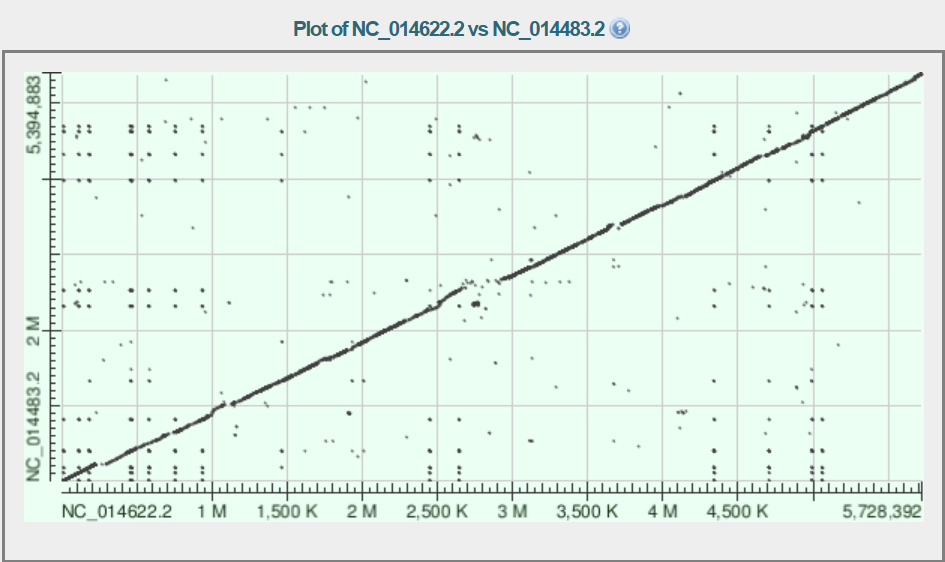
Табличная выдача находок blast Рис.2. Карта локального сходства SC2 (NC_014622.2) и CR1 (NC_023037.2)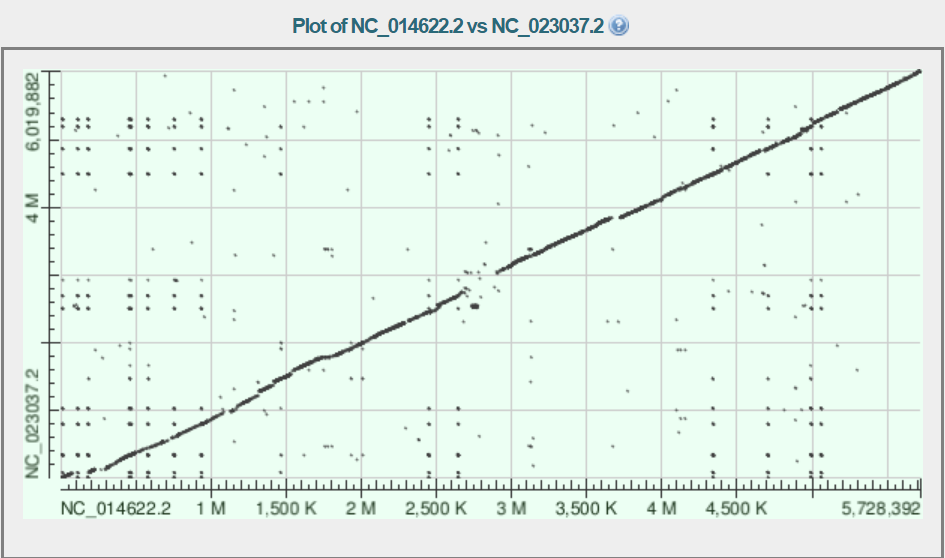
Табличная выдача находок blast Рис.3. Карта локального сходства E681 (NC_014483.2) и CR1 (NC_023037.2)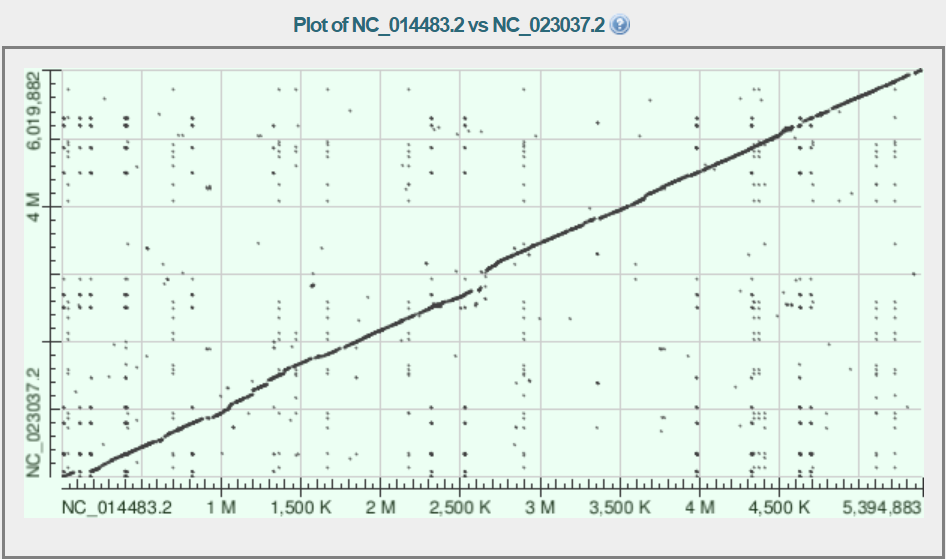
Табличная выдача находок blast Вычисление сходства (identity %) на гомологичных участках геномовДалее вычисляю сходство всех 3 геномов. Таблица 2. Информация о выравниваниях
Из таблицы прикидка: покрытие гомологичных участков около 80 %. Теперь будем работать с картами локального сходства и вычислим приблизительное покрытие гомологичных участков. Составлю таблицу с грубыми приближениями гомологичных участков (границы в килобазах). Таблица 3. Гомологичные участки
Суммарная длина гомологичных участков - 5000 килобаз, это 83,1% от самого длинного генома (CR1). Очевидно, что приближение, очень грубое, теперь нужно его уточнить. Терминология: Ортологи - гомологи в разных организмах Паралоги - гомологи в одном организме Синтения – неологизм (John Renwick, 1971). Изобразим гены в геноме стрелочками. Ортологичные гены пометим одинаковым цветом или еще как-нибудь (одинаковым номером) Предположим, в геноме 1 и 2 найдутся одинаковые последовательности генов или комплементарные. Тогда соответствующие участки геномов называются синтеничными. Конечно, берутся максимальные такие последовательности. Данные таблиц, полученных при выравниваниях, я занесла в данный файл Excel. В практикуме 13 был использован пакет биоинформатических программ bedtools, тк предыдущие расчёты были примитивны, я хочу применить его для подсчёта покрытия.
Исследуйте один или несколько типов крупных перестроекВ рассматриваемом мною примере на картах локального сходства можно увидеть делеции/вставки, а также дупликацию. Так, например, на рис. 1 видна делеция (или вставка) в штамме E681 а участке, соответствующем области примерно с 2,7 до 2,9 млн в штамме SC2. Для конкретных цифр использую сервер NCBI. В SC2 5,495 кодирующих генов, а в E681 4,515 Рис.4. Предполагаемая вставка/делеция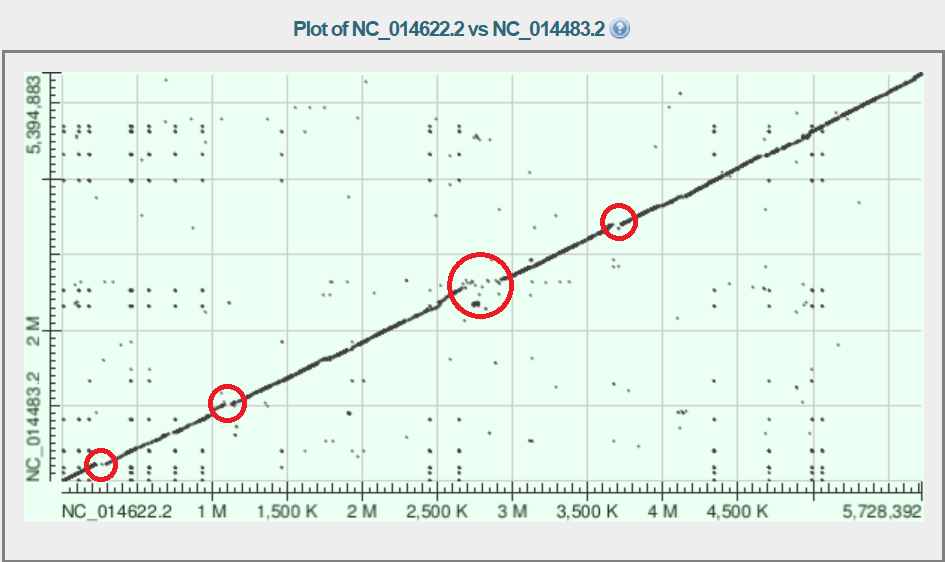
Рис.5. Предполагаемая дупликация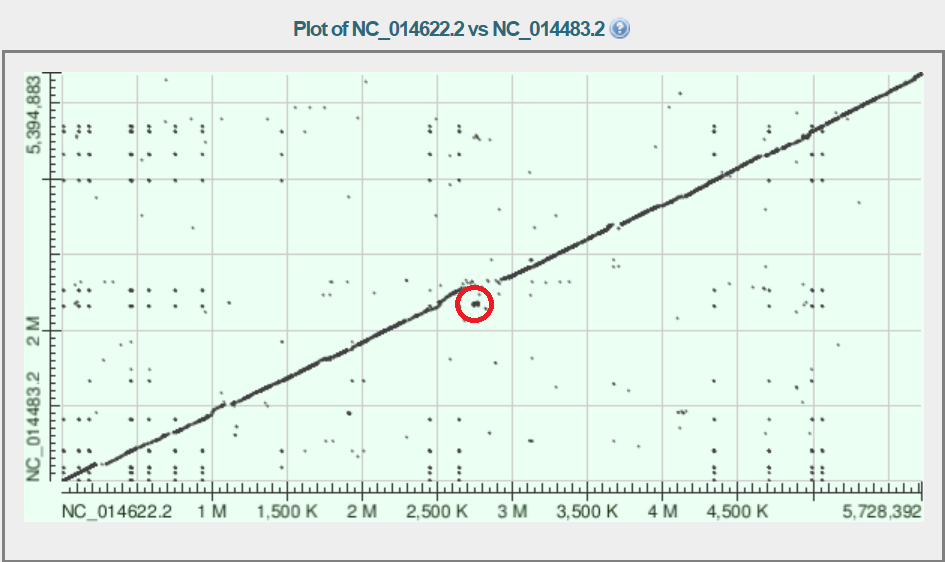
Красным контуром отмечено место предполагаемой дупликации, она небольшая, но всё равно интересная, тк также присутствует с выравнивании SC2 и CR1 (рисунок 2). При этом зона, в которой она находится, это зона предполагаемой делеции/вставки и находится почти на кооринатах разрыва сплошной линии выравнивания. Получается, что в SC2 есть участок, который повторяется трижды: который лежит на линии диагонального выравнивания и дважды в выделенном мною фрагменте. Из-за этого факта я все-таки считаю, что в самом большом гэпе в области 2,7 - 2,9 млн нуклеотидов в SC2 произошла не делеция, а вставка. |
Источники