



 |  |  |  |
Нуклеотидный blast |
Вернуться на страницу семестра Нуклеотидные банки данных1. Определение таксономии и функции прочтенной нуклеотидной последовательности Произведён поиск по нуклеотидному blast
Рис.1. Результат поиска blast (прямая цепь). Лучшие находки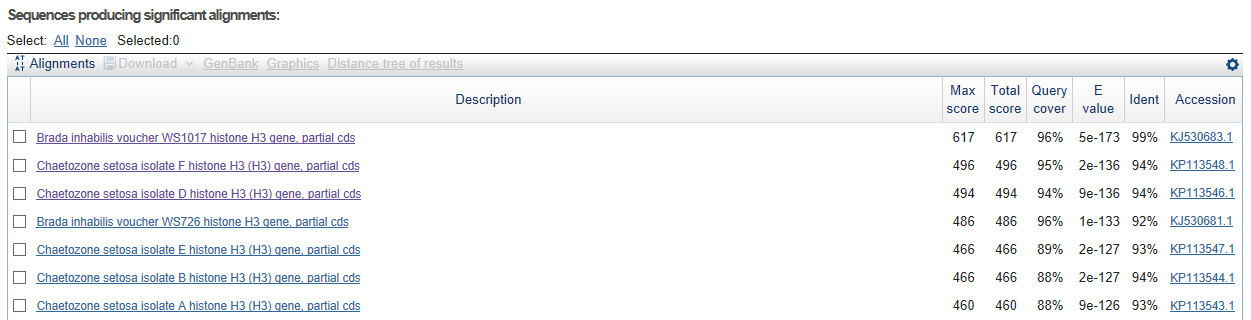
Рис.2. Результат поиска blast (обратная). Лучшие находки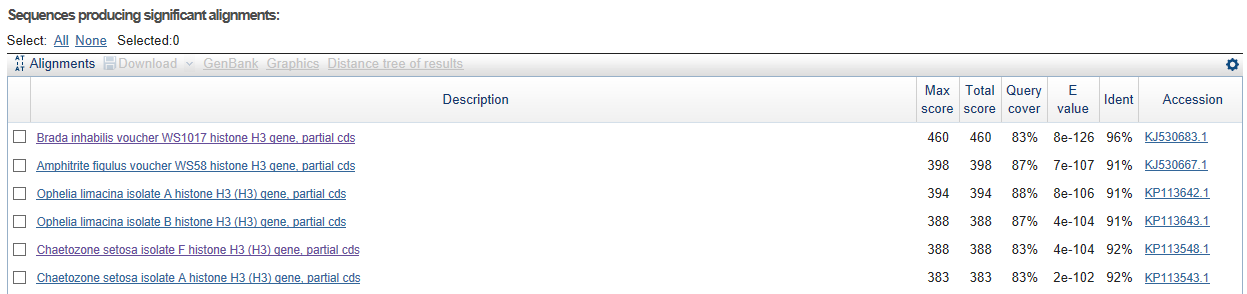
Рис.3. Лучшее выравнивание прямой цепи с Brada inhabilis voucher WS1017 histone H3 gene
Рис.4. Лучшее выравнивание обратной цепи с Brada inhabilis voucher WS1017 histone H3 gene
В поиске и по прямой, и по обратной цепи лучшая находка - выравнивание с Brada inhabilis voucher WS1017 histone H3 gene, partial cds. AC KJ530683. Причем все несовпавшие позиции это N (любой нуклеотид), которые выдал секвенатор. Процент покрытия больше 90%, а E-value очень мало. Поэтому мы считает, что находка это AC KJ530683, частичная кодирующая последовательность гена гистона H3 Brada inhabilis voucher WS1017 Таксономия
Brada inhabilis
Eukaryota; Metazoa; Lophotrochozoa; Annelida; Polychaeta; Palpata;
Canalipalpata; Flabelligerida; Flabelligeridae; Brada.
Рис.5. Brada
Описание вида: Тело короткое и толстое, U-образное, без глаз. Эпидермис густо покрыт круглыми сосочками, усыпанными мелким песком. Размер до 60 мм для 26 сегментов. Обитает на песке, грязи, гравии, смешанных кустах, между камнями. [1] 2. Сравнение списков находок нуклеотидной последовательности 3-я разными алгоритмами blastБанк nr/nt, поиск в таксоне Mollusca, кроме Gastropoda, порог E-value 1.0E-100 ZEZWZ840015 megablast: длина слова 28 megablast: число находок 21 Рис.5. Находки megablast Рис.6 Находки discontiguous megablast Рис.7 Находки blastn Находок в двух последних алгоритмах заметно больше, например, Laevipilina hyalina histone H3 gene, partial cds. Если говорить о верхних позициях, то в megablast не был найден Crassinella lunulata voucher MCZ 379774 histone H3 gene, partial cds Megablast отвечает за поиск очень похожих последовательностей, длина слова в поиске была 28 (более чем в 2 раза длиннее, чем у Discontigous megablast и BlastN). Как раз из-за длины слова находки Crassinella lunulata voucher MCZ 379774 histone H3 gene, partial cds нет в megablast. Также можно увидеть, что расчёт E-value отличается в алгоритмах. Я поставила ограничение на него: менее 1.0E-100, в Megablast E-value увеличивался с какждой находкой быстрее, поэтому и отсеялось больше находок, чем в других алгоритмах. Это объясняет отсутствие Laevipilina hyalina histone H3 gene, partial cds в выводе megablast 3. Проверка наличия гомологов трех белков в геноме одного организмовОрганизм X5 (Amoeboaphelidium protococcarum)Я взяла белки: HSP71_YEAST - шаперон HSP70, белок теплового шока; TBB_NEUCR - тубулин, белок, участвующий в образовании микротрубочек PRPC_EMENI - митохондриальная цитратсинтаза Проверка HSP71_YEAST Проверка TBB_NEUCR Проверка PRPC_EMENI Таблица 1. Результаты поиска гомологов
4. Найти один ген белка, закодированный в одном скэффолде ''Amoeboaphelidium protococcarum''
|
Источники [1]Institute of Oceanology, Brada inhabilis