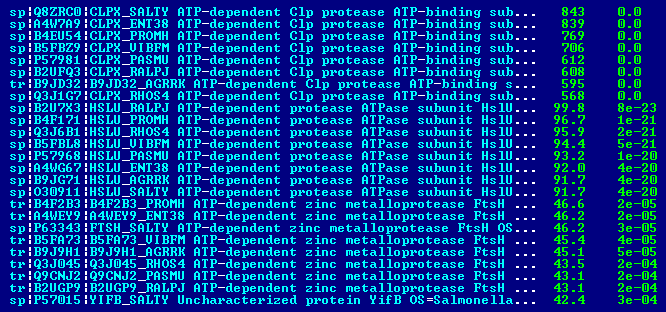
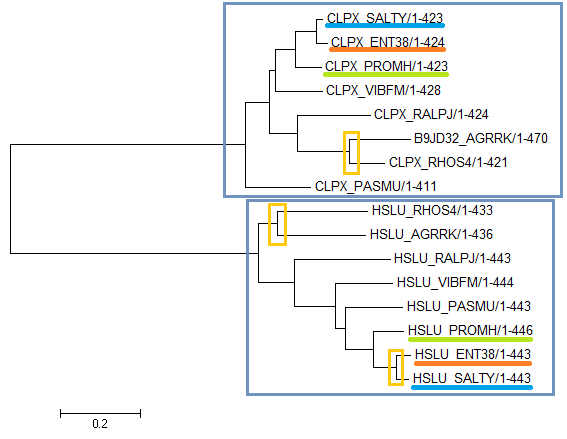
Последовательности 16S рРНК были взяты с сайта NCBI из файлов с расширением .frn
В наличии были последовательности не всех бактерий, поэтому в нескольких случаях пришлось
взять родственные виды или другие штаммы того же вида:
Vibrio fischeri str. ES114 (VIBF1)
Agrobacterium fabrum str. C58 (AGRFC)
Rhodobacter sphaeroides KD131 (RHOSK)
Последовательности были сохранены в файл
16SrRNA_all.fasta
Выравнивание было получено при помощи Muscle.
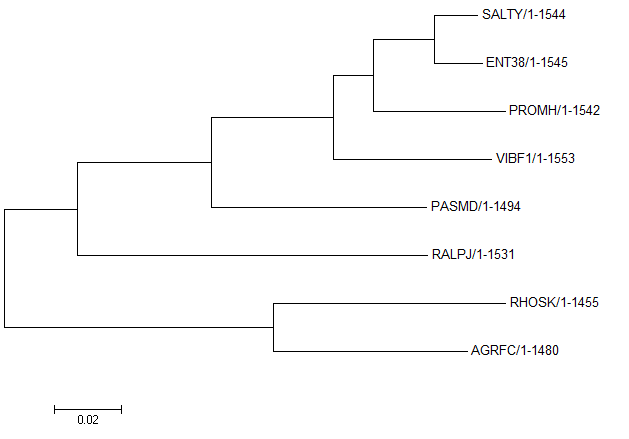
В программе MEGA методом Maximum Likelihood было получено дерево.
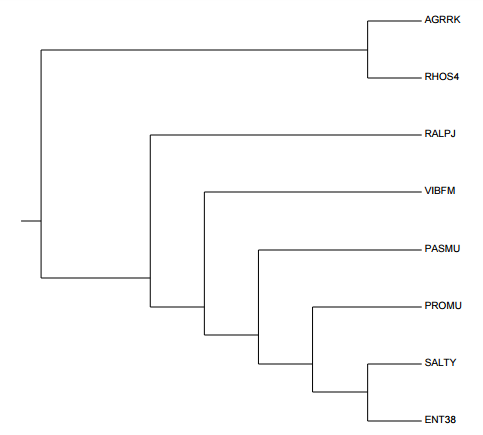
Рис1. Слева: дерево, полученное программой MEGA. Справа: "правильное" дерево, полученное из таксономии.
Эти два дерева отличаются по топологии: VIBF1 и PASMD поменялись местами.