Семестр 3, практикум 2
Назад на учебную страницу Птицыной ЕленыО структуре нуклеиновых кислот
Суть работы сводится к сравнительному анализу канонической ДНК и стеблей тРНК.
Задание 1
С помощью пакета 3DNA, работающего под операционной системой Linux, были построены структуры 3 форм ДНК (А-форма, B-форма, Z-форма). Пример команды: fiber -a -seq=GATCGATCGATCGATCGATC gatc-a.pdb Файлы: gatc-a.pdb, gatc-b.pdb, gatc-z.pdb.
Задание 2
Если рассмотреть ДНК, можно заметить, что в ней можно выделить большую и малую бороздку. Например, у рассмотреного нами в
экспериментальной структуре B-формы ДНК тимина в сторону большой
бороздки обращены атомы a8.c4, a8.o4, a8.c5, a8.c7(c5m), a8.c6 в сторону малой бороздки обращены атомы a8.c2, a8.o2, a8.n1 (Рис. 1, 2).
Далее мы сравнили А, B, Z-формы по различным параметрам.
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
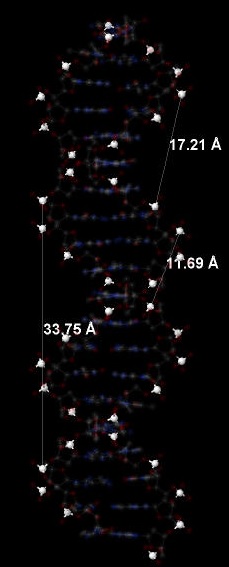
| Шаг спирали (Å) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 16.81 ([DT]31:B.P #618 - [DC]12:A.P #228) | 17.21 ([DC]4:A.P #64 - [DA]34:B.P #679) | 29.25 ([DC]6:A.P #105 - [DG]29:B.P #575) |
| Ширина малой бороздки (Å) | 7.98 ([DT]3:A.P #44 - [DC]32:B.P #638) | 11.69 ([DG]33:B.P #657 - [DC]12:A.P #228 : 11.694147) | 14.27 ([DC]28:B.P #556 - [DG]19:A.P #370) |
| Вид в Jmol | 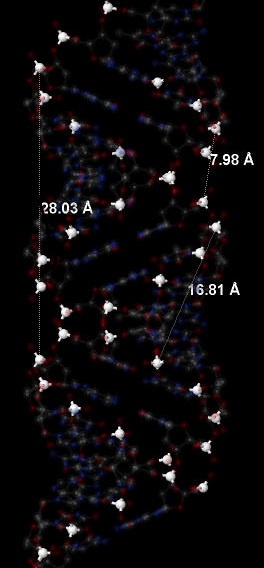
|
 |
 |
Задание 3
В Задании 3 мы определяли параметры структуры нуклеиновых кислот с помощью программ пакета 3DNA.
Упражнение 1.
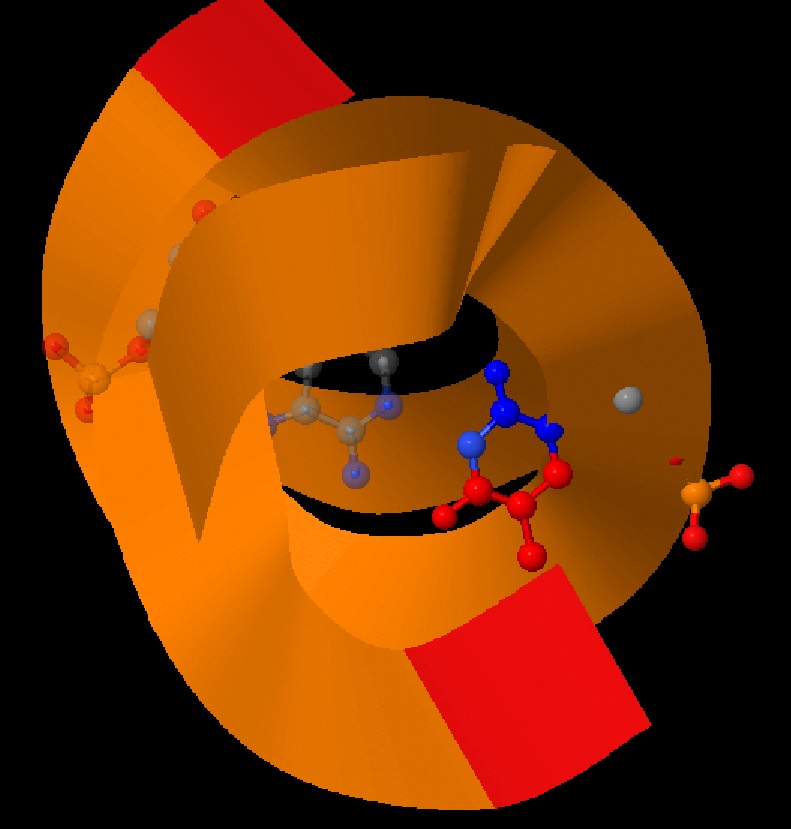
Торсионные углы были определены для A, B, Z-форм ДНК и для 1gtrRNA (структуры ДНК из Задания 1, структура РНК скачана с сайта PDB в pdb-формате, который который перевед в старый pdb-формат командой remediator --old ''1gtr.pdb'' > ''1gtr_old.pdb''). Чтобы определить торсионные углы для молекул нуклеиновых кислот, была использована команда find_pair. Пример для gatc-a.pdb: find_pair -t gatc-a.pdb stdout | analyze . В выходном файле можно найти значения торсионных углов (Рис. 3).
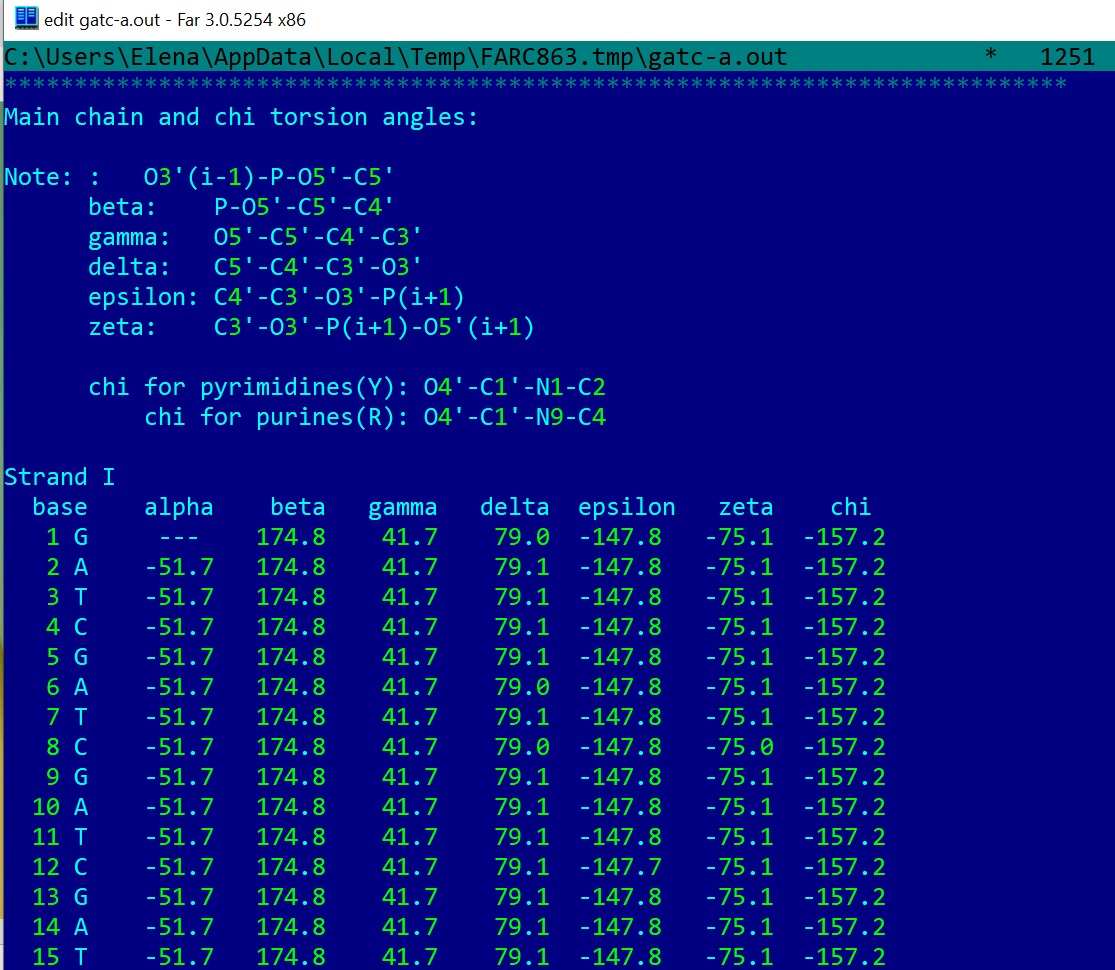
Из каждого файла мы перенесли значения торсионных углов в документ Excel, там посчитали медианные значения торсионных углов для каждой нуклеиновой кислоты (Табл. 1). Файл с расчётом:sravn.xlsx . Из таблицы видно, что по торсионным углам наша РНК наиболее похожа на A-форму ДНК.
Таблица 1. Сравнение торсионных медианных углов.| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| A-DNA | -51,7 | 174,8 | 41,7 | 79,1 | -147,8 | -75,1 | -157,2 |
| B-DNA | -29,9 | 136,3 | 31,1 | 143,3 | -140,8 | -160,5 | -98 |
| Z-DNA | -139,5 | 21,15 | -61,5 | 116,25 | -103,6 | -64,8 | -47,8 |
| 1gtrRNA | -65,2 | 159,2 | 59,6 | 80,7 | -141,9 | -78,1 | -161,8 |
Упражнение 2.
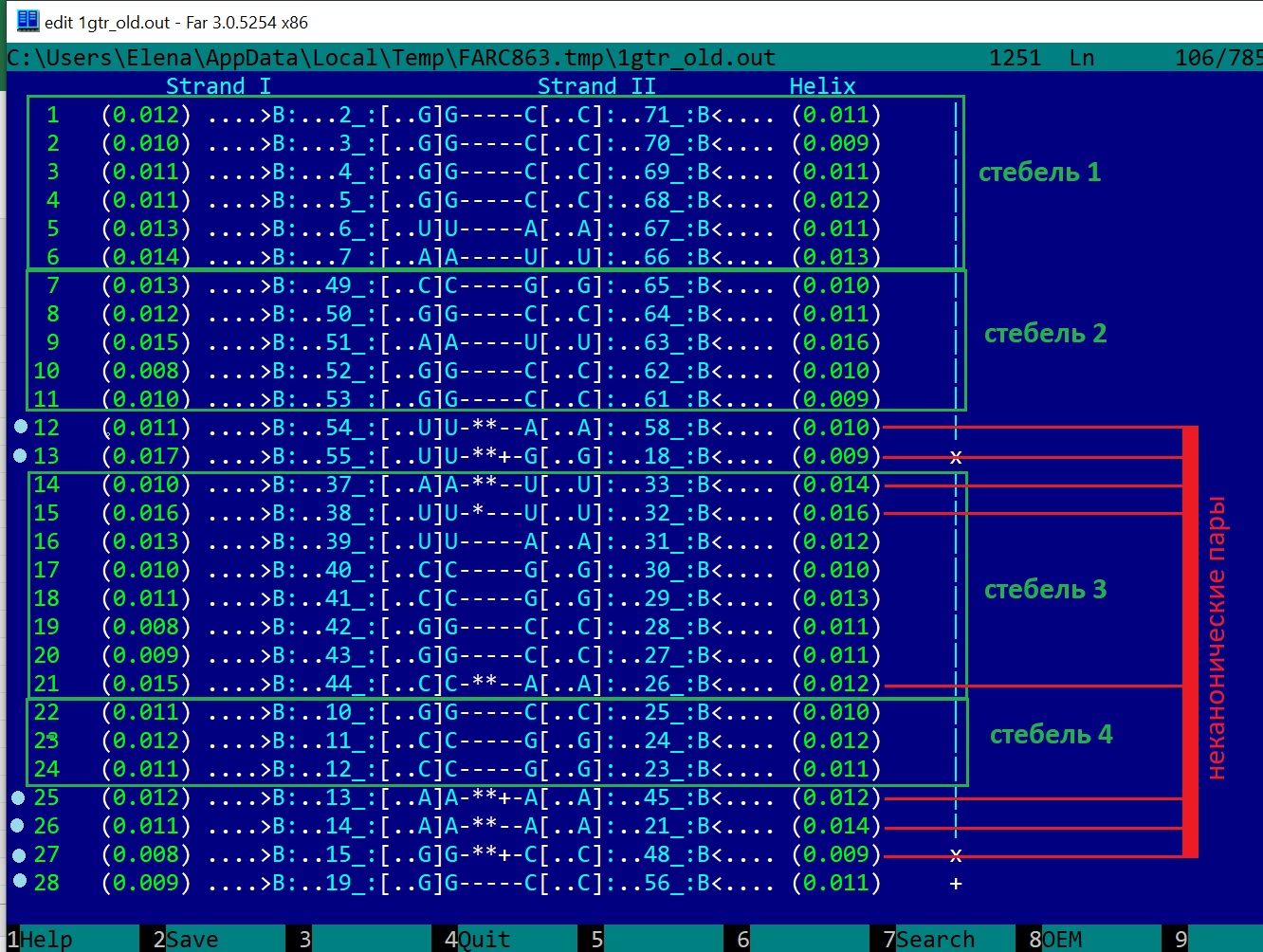
В 1gtrRNA можно найти 4 стебля, 8 неканонических пар, 6 дополнительных водородных связей, стабилизирующих её структуру (Рис. 4).