A- и В- формы ДНК. Структура РНК
Задание 1
Задание 2
Рисунок 1. Сахарофосфатный остов выделен wireframe синего цвета. Атомы N7 выделены cpk зелёного цвета.
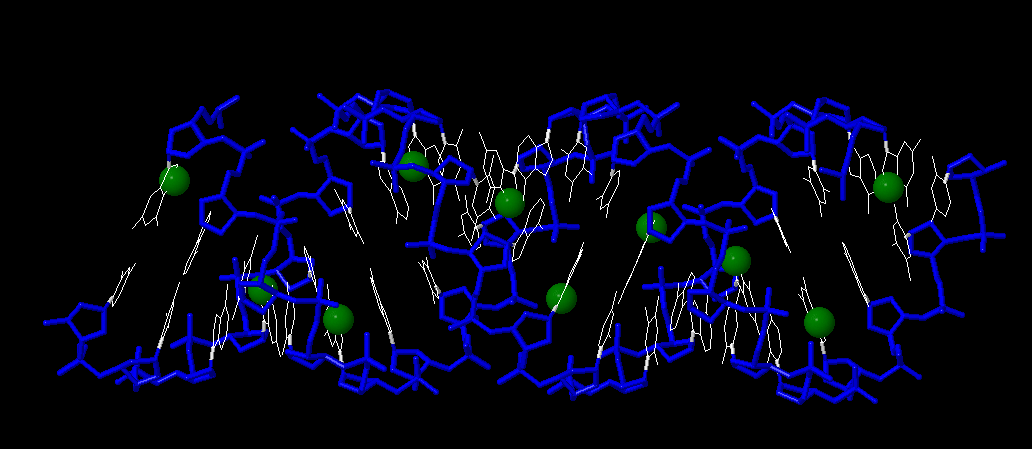
Рисунок 2. Выделение cartoons. Нуклеотиды, содержащие аденин, выделены белым цветом.
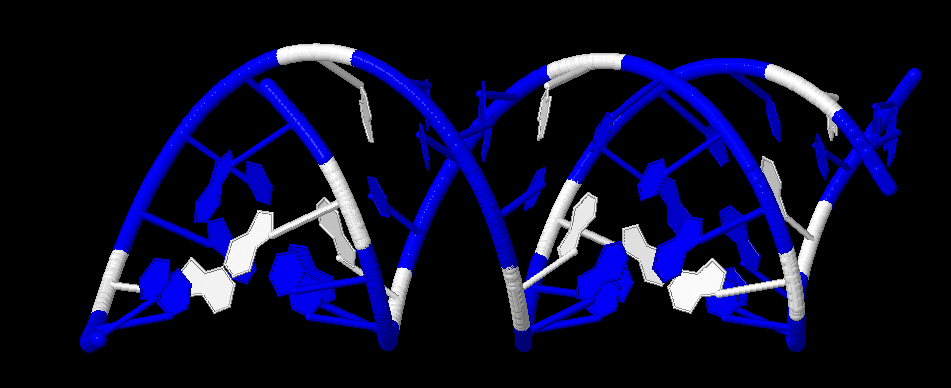
Рисунок 3. В участке молекулы ДНК отсутствуют разрывы.
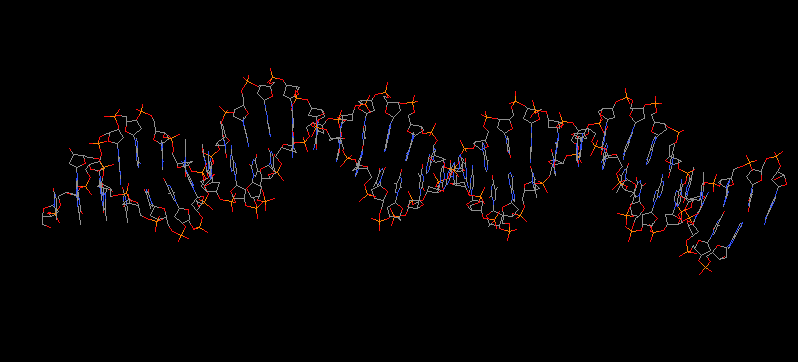
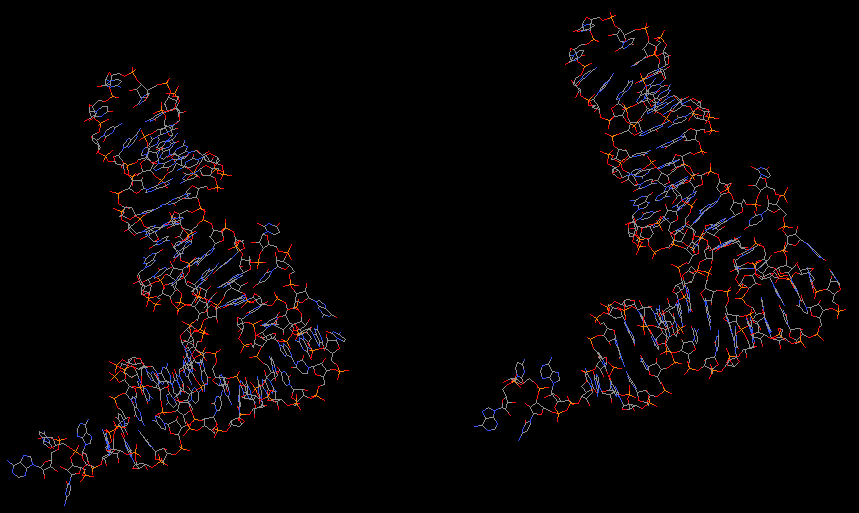
Рисунок 4. В участке молекулы РНК отсутствуют разрывы.
Задание 3
В А-форме в сторону большой бороздки обращены атомы T7.C6, T7.O4, T7.C5M, T7.C5, в сторону малой - T7.C2, T7.O2.
В В-форме в сторону малой бороздки обращены атомы T7.C6, T7.O4, T7.C5M, , T7.C5, T7.C4, в сторону большой - T7.C2, T7.O2.
Рисунок 5. Тимидиловые нуклеотиды.
Основные спиральные характеристики форм ДНК |
A-форма |
B-форма |
Z-форма |
| Тип спирали (правая или левая) | Правая |
Правая |
Левая |
| Шаг спирали (A) | 28,03 |
33,75 |
43,5 |
| Число оснований на виток | 11 |
10 |
12 |
| Ширина большой бороздки (A) | 16.83 ([G]25:B.P - [A]18:A.P) |
17,21 ([C]4:A.P - [A]34:B.P) |
16,08 ([C]14:B.P - [C]4:A.P) |
| Ширина малой бороздки (A) | 7,98 ([T]3:A.P - [C]32:B.P) |
11,69 ([A]26:B.P - [T]19:A.P) |
7.2 ([G]5:A.P - [G]19:B.P) |
Рисунок 6. Торсионные углы в тимидиловом нуклеотиде (А-форма).
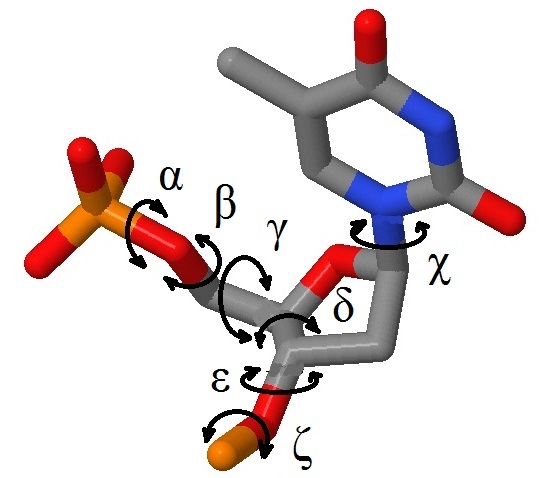
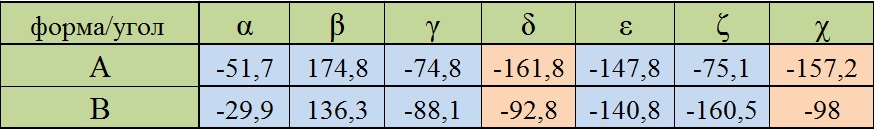
Таблица 1. Результаты измерения торсионных углов в A- и В- формах ДНК.
Задание 4. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Ссылка на таблицу, содержащую информацию о торсионных углах ДНК и РНК
Исходя из информации, представленной в этой таблице, а также из Таблицы 1, я нашла нуклеотид РНК, значения чьих торсионных углов больше всего отличались от средних в РНК (это оказался двенадцатый нуклеотид на первой цепи). Также я сравнила значения торсионных углов в А- и В-форме ДНК и РНК:
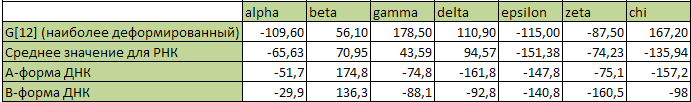
Как видно из таблицы, большее сходство с РНК имеет А-форма ДНК. РНК имеет гораздо более сложное пространственное расположение благодаря водородным связям. С помощью программ find_pair и analyze был получен файл 1G59_old.out, из которого и была получена информация о водородных связях данной РНК:
1) акцепторный стебель состоит из 5'-концевого участка 1-7 и комплементарного ему 3'-концевого участка 66-72;
2) Т-стебель из 49-53 и 61-65 соответственно;
3) D-стебель из 10-12 и 23-25 соответственно
4) антикодоновый стебель из 38-44 и комплементарного ему 26-32 участка.
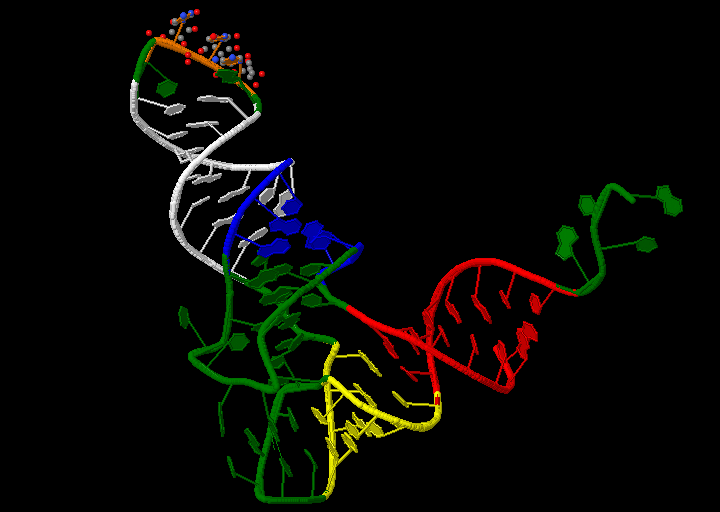
Рис.7. Глутамил-тРНК. Акцепторный стебель выделен красным,
Т-стебель - жёлтым, D-стебель - синим, антикодоновый - белым. В шарнирной модели представлен антикодон к глутамату.
9 (0.006) B:.550_:[..G]G-----C[..C]:.564_:B (0.011)
10 (0.007) B:.551_:[..G]G-----C[..C]:.563_:B (0.010)
11 (0.007) B:.552_:[..G]G-----C[..C]:.562_:B (0.010)
23 (0.015) B:.511_:[..U]U-----A[..A]:.524_:B (0.013)
30 GC/GU 6.86( 4.14) 0.00( 0.00) 0.00( 0.00) 6.65( 4.06) 13.51( 8.20)
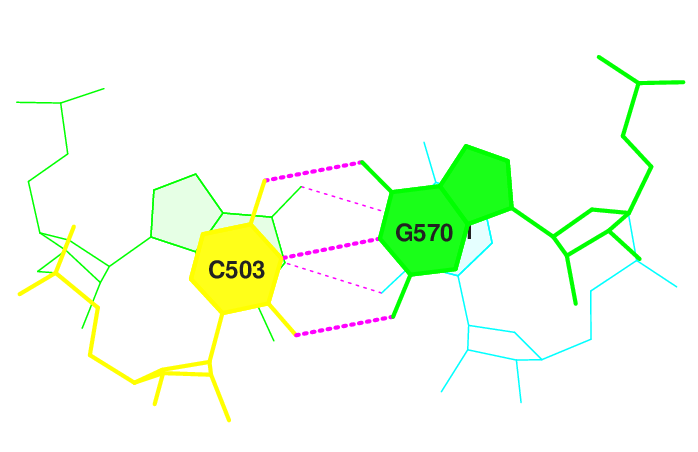
Рис.8. Пара G-C с наиболее сильными стэкинг-взаимодействиями, визуализация stack2img.
Анализируя представленную выше таблицу, считаю необходимым отметить, что наиболее деформированы (в плане наличия нестандартных торсионных углов) гуаниновый нуклеотид в позиции 10 и тимидиновый нуклеотид в позиции 18 цепи 1 (ДНК) и гуаниновый нуклеотид в 12 позиции 1 цепи и цитидиловый нуклеотид в позиции 55 цепи 2 (РНК).