Практикум 4. Паралоги, визуализация
Получение гомологов CLPX_ECOLI: blastp с evalue < 10^-3 и базой данных из протеомов отобранных бактерий.
Множественное выравнивание: в Jalview алгоритмом Tcoffee последовательностей гомологов, полученных из Uniprot.
Дерево: в MEGA алгоритмом NJ с бутстреп-анализом.
Отобранные бактерии
| Name | Mnemonic | Taxonomy |
|---|---|---|
| Clostridium tetani | CLOTE | Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Lactobacillus acidophilus | LACAC | Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactococcus lactis subsp. cremoris | LACLM | Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus; Lactococcus lactis |
| Moorella thermoacetica | MOOTA | Firmicutes; Clostridia; Thermoanaerobacterales; Thermoanaerobacteraceae; Moorella group; Moorella |
| Staphylococcus aureus | STAAR | Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Staphylococcus epidermidis | STAES | Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Streptococcus pneumoniae serotype 4 | STRPN | Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
Скобочная формула дерева (упрощенная)
((((((((((Q899H3_CLOTE/1-603,Q2RM95_MOOTA/1-645),(Q5HRP3_STAEQ/1-700,Q2G0R0_STAA8/1-697)),(FTSH_LACLA/1-695,(FTSH_STRPN/1-652,Q5FMA3_LACAC/1-718))),Q891B9_CLOTE/1-624),Q2RJJ8_MOOTA/1-494),Q898D1_CLOTE/1-576),Q2RLP6_MOOTA/1-415),Q2RLR4_MOOTA/1-370),((CLPL_STAA8/1-701,A0A0H2UNL3_STRPN/1-701),((A0A0H2USJ7_STRPN/1-810,Q5FM98_LACAC/1-825),(Q5FHW6_LACAC/1-709,CLPE_LACLA/1-748)))),((((HSLU_STAA8/1-467,HSLU_STAEQ/1-467),HSLU_LACAC/1-466),Q2RJP5_MOOTA/1-461),(((CLPX_LACLA/1-411,CLPX_STRPN/1-410),Q5FKR6_LACAC/1-420),((CLPX_CLOTE/1-431,CLPX_MOOTA/1-419),(CLPX_STAEQ/1-420,CLPX_STAA8/1-420)))),(Q895L6_CLOTE/1-524,(Y1413_STAA8/1-263,Y979_STAEQ/1-263)));
Изображение дерева
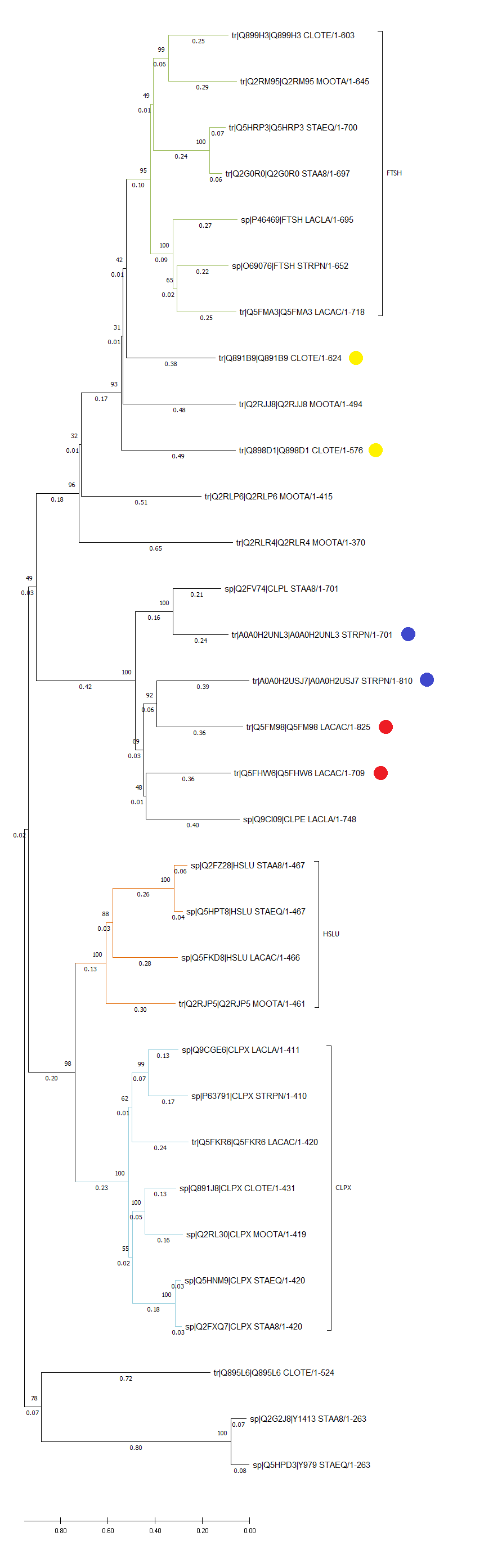
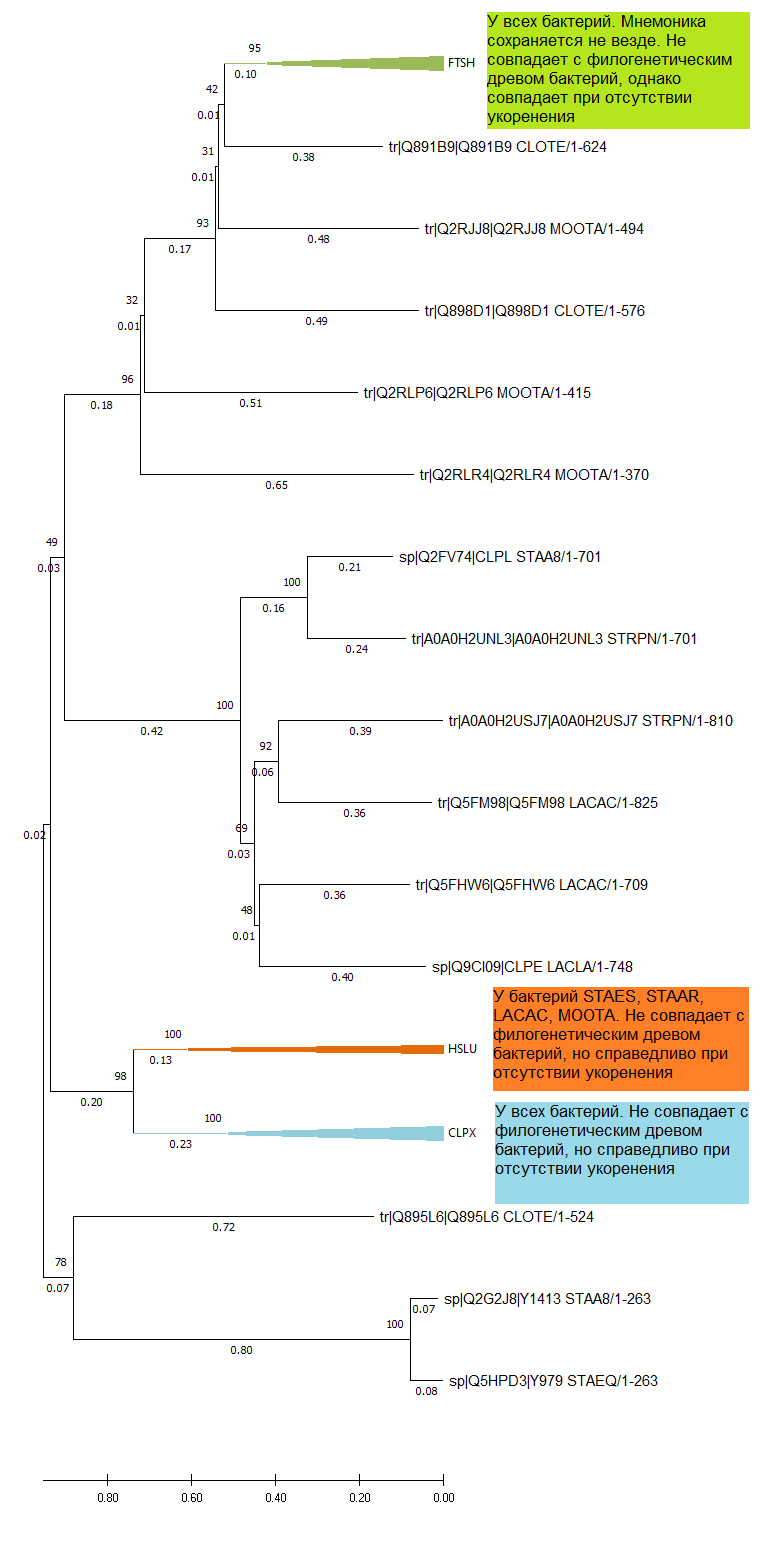
На изображениях указаны 3 крупные ортологичные группы. Несмотря на то, что дерево для филогенетического положения бактерий (с учетом укоренения, обусловленного систематическим положением бактерий [1]) не совпадает с ветвями двух полных ортологичных ветвей, идентичность структуры этих ветвей, вероятно, может говорить о достоверности построенных отношений родства ортологов.
Также указаны три пары достаточно близких паралогов. Кроме того, можно сказать, что 2 ортологичных группы (FTSH и CLPX), в каждой из которых есть белки всех 7 отобранных бактерий, соответствуют 7 парам паралогов.









