Реконструкция эволюции доменной архитектуры
Выравнивание представителей домена Pfam белков с разной доменной архитектурой
Был выбран домен PDEase_I (ID: PDEase_I, AC: PF00233) - домен, обладающий фосфодиэстеразной активностью, то есть способностью гидролизовать 3'-5' фосфодиэфирную связь циклических нукдеотидов.
В выбранном семействе 5152 последоватальности, которые принадлежат 711 видам. Домен входит в 77 архитектур (ссылка). Для 564 последовательностей имеется 3D структура.
Были выбраны две различные доменные архитектуры, включающие в себя домен PDEase_I (зеленый), характеристика которых приведена в таблице 1 ниже:
| Таблица 1. Характеристика доменных архитектур | |||
|---|---|---|---|
| № | Домены, входящие в архитектуру | Число последовательностей | Изображение доменной архитектуры |
| 1 | PDEase_I | 3037 | |
| 2 | PDEase_I, GAF (x2) | 645 |
Характеристика доменов:
GAF (красный) - домен, содержащийся в цГМФ фосфодиэстеразах, аденилат циклазах; имеет ту же каталитическую активность.
Для получения информации об архитектуре всех последовательностей, содеражщих домен PDEase_I, был использован скрипт, отбирающий только те последовательности, чей домен совпадает с вводимым:
python swisspfam_to_xls.py -z /srv/databases/pfam/swisspfam.gz -p PF00233 -o PF00233.xls
В полученную таблицу была добавлена сводная таблица, в которой содержится информация о принадлежности последовательности к архитектуре. При этом была добавлена колонка с таксономической принадлежностью в изначальный файл при помощи VLOOKUP (ВПР). Сама таксономия была получена при помощи скрипта. Для этого была создана таблица с информацией о таксономии из Uniprot:
python uniprot_to_taxonomy.py -i taxonomy.txt -o taxonomy.xls
Итоговую таблицу с листом filter можно скачать здесь. В листе filter содержится информация о выбранных последовательностях, которые относятся к одной из архитектур, при этом характерны для одного из подтаксонов - Ecdysozoa или Chordata.
Далее, при помощи скрипта было построено выравнивание только тех (!) последовательностей, которые были выбраны в таблице. В итоге, полученный fasta файл с выравниванием использовался программой Jalview.
После тщательного "осмотра", были удалены какие-то участки выравнивания. В итоге осталось 36 последовательностей. В целом выравнивание неплохое. Есть даже небольшие вертикальные блоки.
Проект jvp можно скачать здесь. *Окраска по ClustalX с порогом консервативности 10%.
Построение филогенетического дерева домена
Далее необходимо было построить дерево, используя полученное выше выравнивание. Для простоты ввели следующую легенду:
P_E - доменная архитектура, содержащая только PDEase_I; характрена для Ecdysozoa;
P_C - доменная архитектура, содержащая только PDEase_I; характрена для Chordata;
GP_E - доменная архитектура, содержащая PDEase_I и GAF(x2); характрена для Ecdysozoa;
GP_C - доменная архитектура, содержащая PDEase_I и GAF(x2); характрена для Chordata.
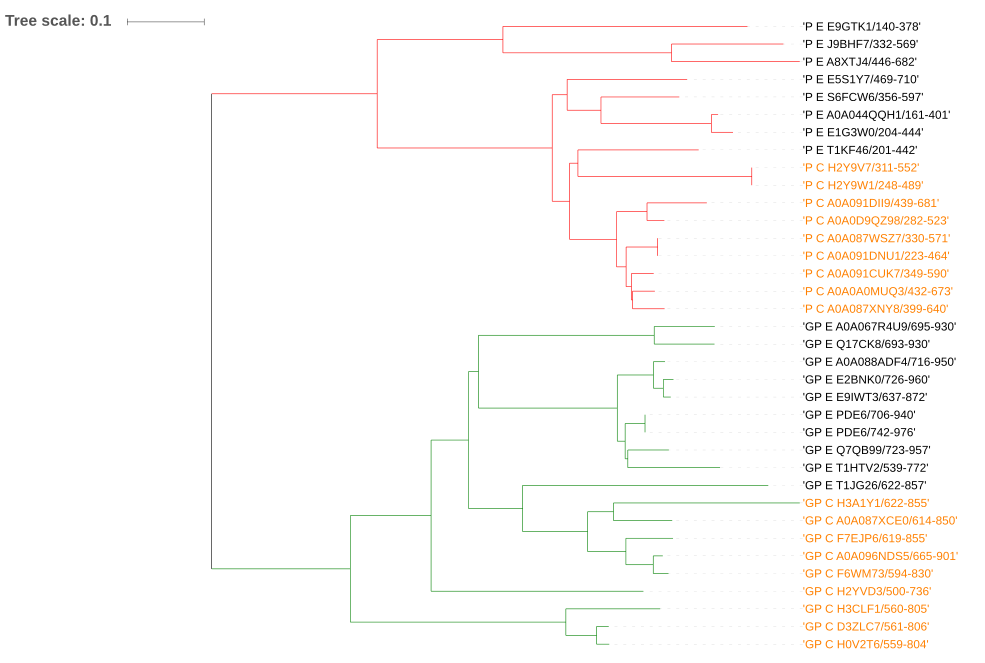
Дерево было построено с помощью MEGA алгоритмом Neighbour Joining без укоренения (!), раскрашено с помощью ITOL. На картинке красным отмечены представители доменной архитектуры PDEase_I, зеленым - PDEase_I + GAF(x2). Названия, выделенные оранжевым, отражают представителей таксона Chordata.
Сразу хочется отметить, что дерево разбивается на две клады, соответствующие архитектурным доменам (карсный и зеленый цвета). Чтобы проверить, что это действительно так, была использована программа retree, которая укореняет дерево в средняя точку. Результаты приведены ниже:
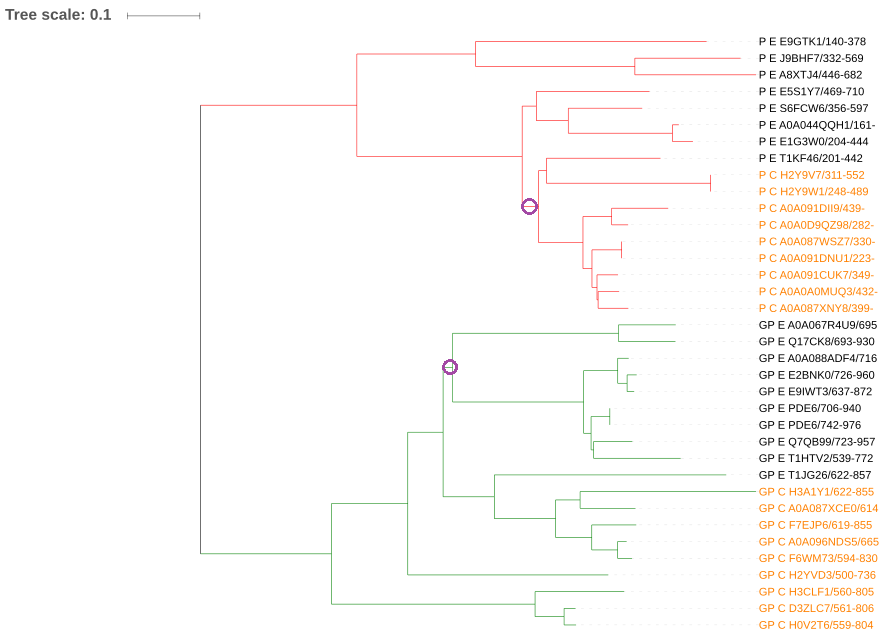
И действительно, видно, что дерево разбивается на прежние клады, соответствующие доменным архитектурам. Внутри архитектуры P (крансый цвет), можно видеть группирование клад по таксонам. Например клада, отмеченная сиреневым кружком, внутри доменной архитекутры P содержит в основном представителей из таксона Chordata. Единственной последовательностью, которая выбивается из этой клады, является последовательность с идентифифактором T1KF46.
Похожая картина наблюдается внутри доменной архитектуры GP (зеленый цвет). Также имеется клада (выделена сиреневым кружком), содержащая только представителей из таксона Ecdysozoa.
⌘
© Emir Radkevich, 2016