ДНК-белковые комплексы
Задание 1
В этом задании сравнивались реальная и предсказанная вторичные структуры тРНК. Сравнение проведено на примере PDBID 2DXI.
Реальная структура получена и проанализирована в Задании 3 предыдущего практикума при помощи find_pair.
Предсказание же делалось на основе fasta-последовательности тРНК посредством программы einverted и онлайн-сервиса RNAfold[1].
Программа einverted из пакета EMBOSS позволяет находить инвертированные повторы в структуре нуклеиновой кислоты. На вход подаётся fasta-последовательность и параметры расчёта Score. При использовании значений по умолчанию ничего не было найдено. Поэтому были использованы следующие:
Gap penalty [12]: 5 Minimum score threshold [50]: 20 Match score [3]: 5 Mismatch score [-4]: -1
На выходе программа einverted дала следующий результат. (Красным дополнительно показаны пары, совпадающие с таковыми в реальной структуре).
SEQUENCE: Score 59: 17/33 ( 51%) matches, 2 gaps
1 ggccccatcgtctagcg--gttaggacgcggccct 33
| ||||| | | || | ||||| |
71 ctggggtcccccttagcttgggggcaaagccggaa 37
Сервис RNAfold использует алгоритм Зукера, который основывается на сравнении свободной энергии структур. На вход так же подаётся fasta-последовательность.
Вывод содержит последовательность в записи формата dot-bracket, в которой "(" и ")" соответствуют 5'- и 3'-основаниям соответственно, а "." обозначает неспаренное основание.
GGCCCCAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGGUUCGAUUCCCCCUGGGGUCACCA (((((((.(((((........)))))(((((.......)))))....(((((.......))))))))))))....
Помимо текстовой записи мы получаем ещё и графическое изображение предсказанной вторичной структуры, которое может быть дополнительно раскрашено по вероятностям образования пар, а также точечный график c этими вероятностями.
Результат выдачи сервиса в такой форме можно видеть на Рис.1 и Рис.2. Дополнительно на Рис.2 показаны различные стебли и количество пар, совпадающих с таковыми в реальной структуре.Рис.1. Точечный график вероятностей образования пар
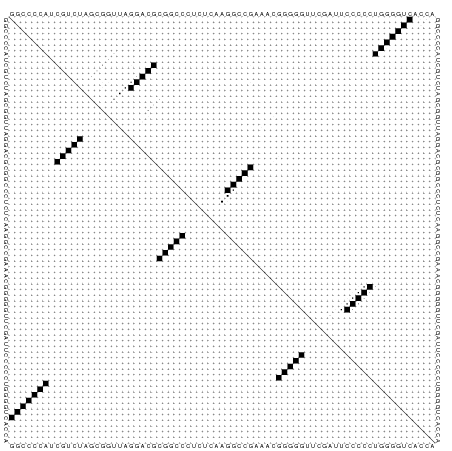
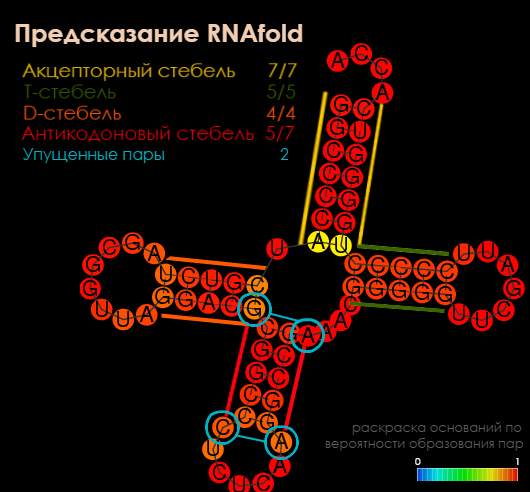
Рис.2. Предсказанная RNAfold вторичная структура c MFE
Результаты сравнения двух предсказанных структур с реальной приведены в Таблице 1.
| Участок структуры | Реальные позиции по результатам find_pair | Предсказано пар по einverted | Предсказано пар по алгоритму Зукера |
| Акцепторный стебель | 5'-501-507-3' 5'-566-572-3' 7 пар | 6/7 пропущена G-U пара | 7/7 |
| T-стебель | 5'-549-553-3' 5'-561-565-3' 5 пар | 0/5 | 5/5 |
| D-стебель | 5'-510-513-3' 5'-522-525-3' 4 пары | 0/4 | 4/4 имеется лишняя пара G-C, которая отсутствует в реальной структуре из-за неканонического взаимодействия G-А |
| Антикодоновый стебель | 5'-526-532-3' 5'-538-544-3' 7 пар | 5/7 пропущены G-A и C-A пары | 5/7 пропущены G-A и C-A пары |
| Общее число канонических пар нуклеотидов | 19 | 11 | 19 |
По результатам, представленным в таблице, можно сделать вывод о том, что основной проблемой обоих подходов было затруднение в учёте неканонических взаимодействий, имевших место в реальной структуре. einverted не построил ни одного из четырёх неканонических взаимодействий в стеблях. У RNAfold это вышло лучше: были учтены две G-U пары.
В целом можно сказать, что RNAfold справляется с поставленной задачей намного эффективнее (результат, описанный мною, это результат первой же выдачи программы). Более того, удобство работы с сервисом очевидно: не нужно перенастраивать параметры Score (более того, затруднительно будет проверить на адекватность выдачу einverted, когда вторичная структура будет действительно неизвестна); имеется графическое оформление результатов работы алгоритма.
Задание 2
В этом задании требовалось описать ДНК-белковые контакты различной природы в заданной структуре (PDBID 1BY4). От белка структуры для анализа была взята лишь одна B-цепь.
При написании скрипта использовались следующие соглашения:
- полярными считаются O, N
- неполярными — C, P, S
- полярный ДНК-белковый контакт — ситуация, в которой расстояние между полярным атомом белка и полярным атомом ДНК не превышает 3.5Å
- неполярный ДНК-белковый контакт — аналогично предыдущему пункту для неполярных, расстояние между которыми не превышает 4.5Å
Сперва был создан скрипт с перечнем определений нужных множеств атомов (Script#2a), таких как, к примеру, множество атомов кислорода из остатков фосфорной кислоты, множество углеродов в остатках дезоксирибозы, множество атомов основания, обращенных в большую/малую бороздку, множество полярных и неполярных атомов и т.д.
С ипользованием этого скрипта был написан второй, Script#2b, в котором с помощью команды within были определены множества атомов, вовлеченных в ДНК-белковый контакт, а затем (через group) — соответствующие множества остатков аминокислот или нуклеотидов. Так были получены участки белка и ДНК, на которых происходит образование комплекса.
Итоговый скрипт использовал определенные ранее множества атомов. Помимо этого потребовалось использовать команду, которая бы изобразила все возможные полярные и неполярные контакты, т.е. соединила бы атомы, находящиеся друг от друга на расстоянии не более чем 3.5 или 4.5Å:
measure RANGE 0 3.5 ALL (atom_expression1) (atom_expression2)
Команда выше отображает все измерения расстояний между атомами вида atom_expression1 и atom_expression2, которые находятся в интервале от 0 до 3.5Å. Для наглядности итоговых изображений числовые результаты измерений можно убрать командой
set measurements off
В итоге на изображении останутся только пунктирные линии, которые мы и будем рассматривать как ДНК-белковые контакты.
Запустить итоговый скрипт можно в апплете JSmol. Участки структуры, вовлечённые в контакт, изображены в проволочной модели, раскрашены в зелёный (ДНК) или малиновый (белок) цвет. Шариками с раскраской cpk показаны контактирующие атомы.
Апплет JSmol. Контактирующие регионы комплекса ДНК-белок
|
зелёный: участки ДНК
малиновый: участки белка Работа с апплетом:
Скрипты для скачивания: |
Количественный анализ полученных контактов приведён в Таблице 2.
Таблица 2. Контакты различных типов в комплексе 1BY4.pdb
| Контакты атомов белка с: | Полярные | Неполярные | Всего |
| Остатками 2'-дезоксирибозы | 0 | 13 | 13 |
| Остатками фосфорной кислоты | 20 | 13 | 33 |
| Остатками оснований со стороны большой бороздки | 3 | 6 | 9 |
| Остатками оснований со стороны малой бороздки | 0 | 0 | 0 |
Как видно из таблицы, со стороны малой бороздки белок никак не контактирует с ДНК, со стороны же большой преобладают неполярные контакты. Больше всего контактов, как полярных, так и неполярных, у белка образовалось с остатками фосфорной кислоты остова ДНК. Это можно объяснить хорошей пространственной доступностью атомов. А вот кислороды дезоксирибозы оказались недоступны для взаимодействий с полярными атомами белка. Остатки сахаров были намного более склонны к образованию неполярных контактов между углеродом сахара и неполярным атомом белка.
Получение популярной схемы ДНК-белковых контактов
Для анализа ДНК-белкового комплекса и получения графического изображения результатов использовалась программа nucplot. Для работы с ней требовалось перевести файл [pdb] в старый формат с помощью remediator:
remediator --pdb --old file.pdb > old_file.pdb
Синтаксис nucplot:
nucplot old_file.pdb
На выходе получали файл-изображение nucplot.ps, который для удобства нужно было конвертировать в [png]-формат:
convert PS:nucplot.ps PNG:nucplot.png
Выдачей программы convert послужили 4 файла: nucplot-0.png, ..., nucplot-4.png. Они приведены на Рис.2
Рис. 2. Изображение ДНК-белковых контактов в nucplot
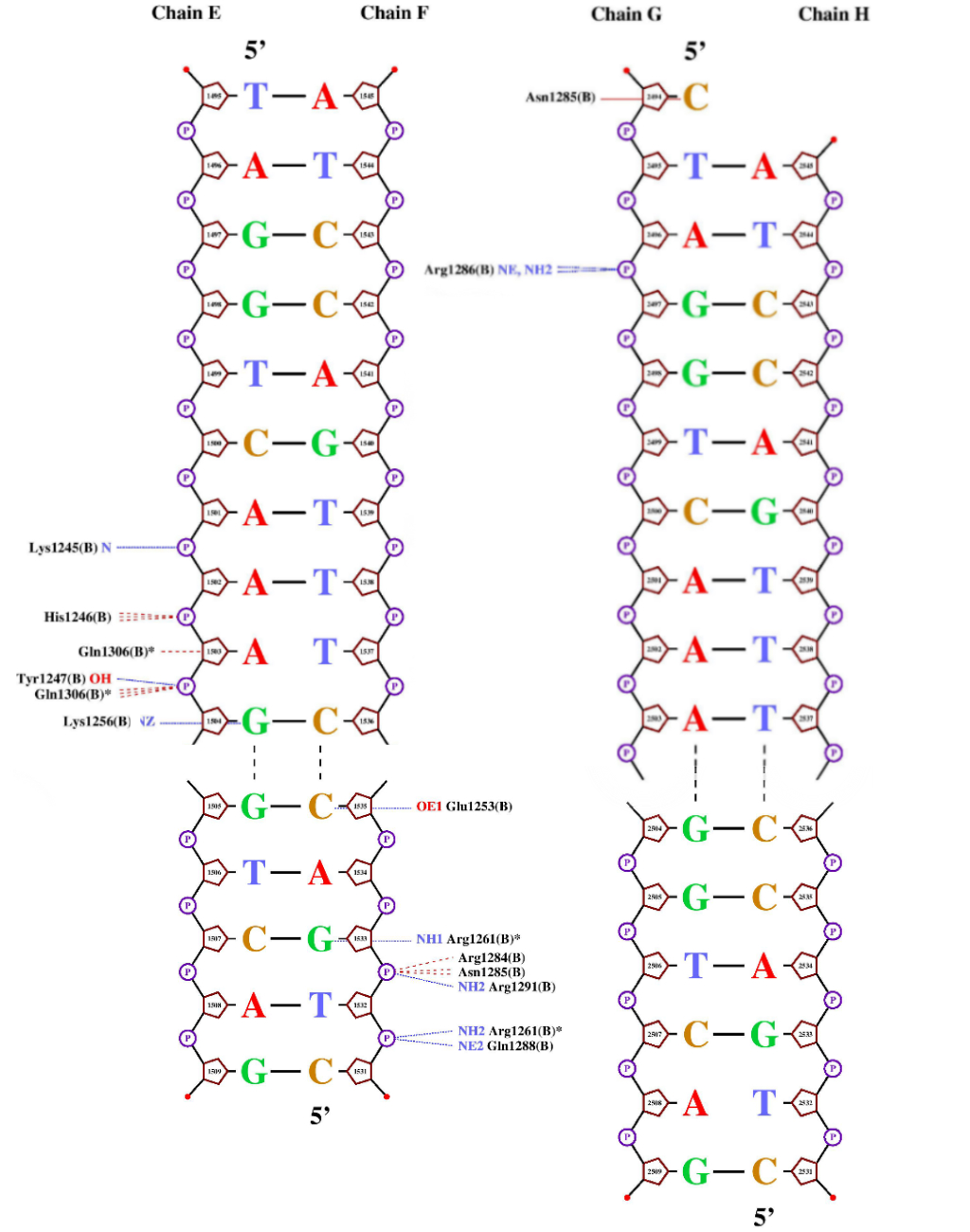
Таким образом очень удобно анализировать соотношение остатков аминокислот белка, участвующих в контакте c ДНК. Для получения более полного представления анализировались контакты ДНК не только с цепью В белка, но и целиком. Итоги приведены в Таблице 3.
Таблица 3. Вклад различных аминокислот в образование комплекса с ДНК
| Остаток аминокислоты | Участие в контактах ДНК и всего белка | Участие в контактах ДНК и цепи B белка |
| ARG | 31 | 6 |
| HIS | 15 | 3 |
| GLN | 12 | 5 |
| LYS | 9 | 2 |
| GLU | 8 | 1 |
| ASN | 6 | 3 |
| TYR | 2 | 1 |
| PHE | 2 | 0 |
| GLY | 1 | 0 |
| VAL | 1 | 0 |
Одной из причин того, что аргинин так часто участвует в контактах с ДНК, может быть его положительный заряд, который притягивается к отрицательно заряженному (заряд остатков фосфорной кислоты) остову спирали ДНК. Аналогичные рассуждения применимы к объяснению относительно высоких частот встречаемости лизина и гистидина.
После рассмотрения схемы nucplot можно предположить, что аргинин и глутамин — одни из важнейших остатков в распознавании ДНК. Дело в том, что, в отличие от других аминокислот, они способны к специфичному связыванию именно азотистых оснований (аргинин предпочтительно связывается с гуанином, а глутамин — с аденином), несмотря на стерические ограничения (расположение оснований внутри спирали). А гистидин, к примеру, хоть и вносит количественно большой вклад в ДНК-белковое взаимодействие, контактирует только с остатками фосфорной кислоты, доступ к которым никаких особых затруднений не вызывает. Следовательно, речь идет о неспецифическом связывании.
Механизм специфического узнавания гуанина остатком аргинина[2]. Аргинин обладает такими замечательными свойствами как (I)достаточная длина цепи (позволяет преодолевать стерические ограничения и подбираться вплотную в основаниям), (II)возможность образования не только монодентатных, но и бидентантых комплексов с гуанином. Здесь стоит отметить, что эта возможность резко повышает уровень специфичности узнавания аргинином гуанина. Это происходит потому, что обычно связи образуются с N7 или O6 атомами гуанина. Но, как известно, N7 атом есть и у аденина. Поэтому остатки, не способные обеспечить образование бидентатного комплекса и взаимодействующие только с N7, не различают между собой эти пуриновые основания.
Сказанное выше применимо и к глутамину, склонному к образованию бидентатных комплексов с аденином (N6, N7 атомы аденина).
На Рис. 3 и Рис. 4 проиллюстрированы контакты остатков аргинина с ДНК через связь с гуанином по N7-типу (с образованием монодентатного комплекса).
Рис. 3
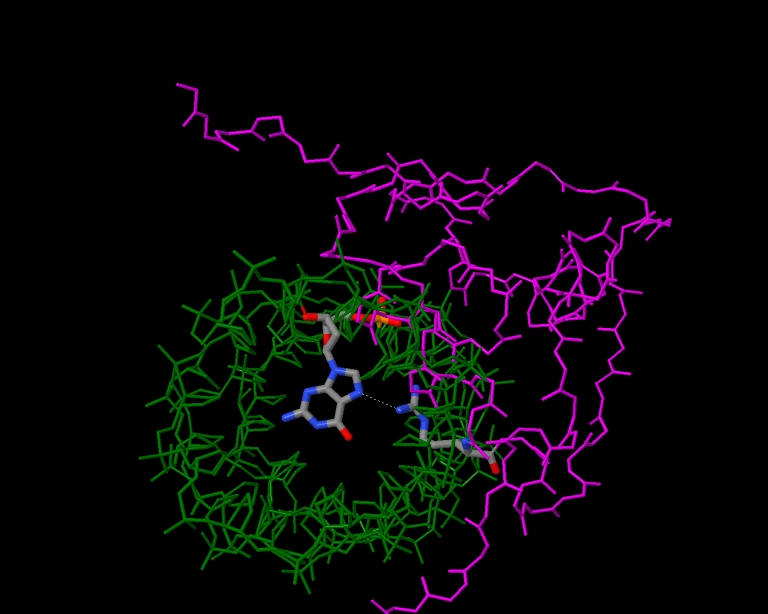
Вид сверху. Пунктирной линией показан контакт ДНК с аргинином. ДНК (зеленый) и белок (малиновый) показаны в проволочной модели. Боковые цепи скрыты. Основание [DG]1533 и остаток аминокислоты [ARG]1261 раскрашены по cpk.
Рис. 4
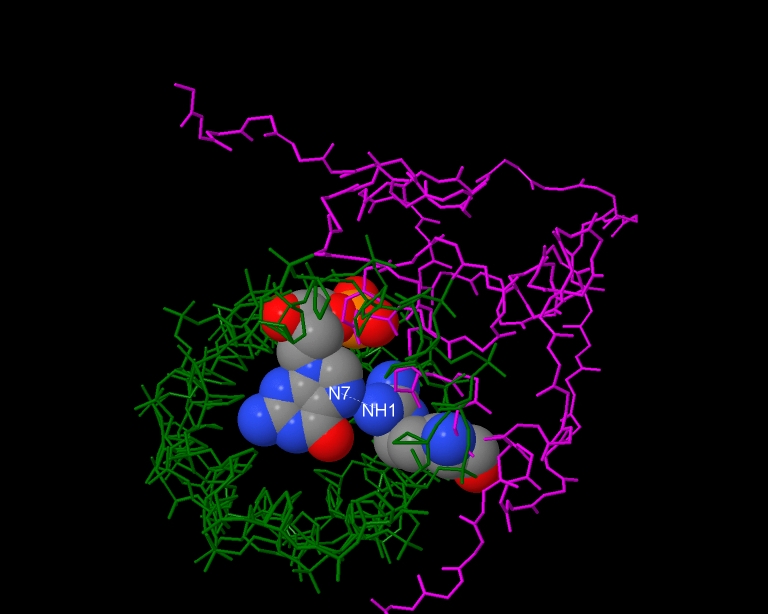
Вид сверху. Пунктирной линией показан контакт ДНК с аргинином. ДНК (зеленый) и белок (малиновый) показаны в проволочной модели. Боковые цепи скрыты. Основание [DG]1533 и остаток аминокислоты [ARG]1261 представлены с учётом ван-дер-ваальсовых радиусов и раскрашены по cpk.
На Рис. 5 и Рис. 6 показано взаимодействие глутамина с ДНК через остаток фосфорной кислоты.
Рис. 5
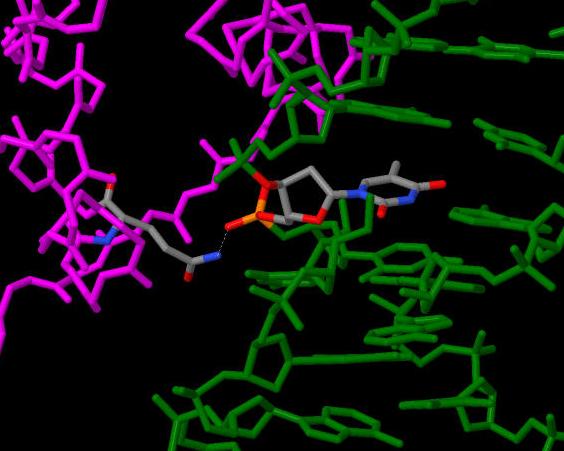
Пунктирной линией показан контакт ДНК с глутамином. ДНК (зеленый) и белок (малиновый) показаны в проволочной модели. Боковые цепи скрыты. Основание [DT]1532 и остаток аминокислоты [ARG]1288 раскрашены по cpk.
Рис. 6
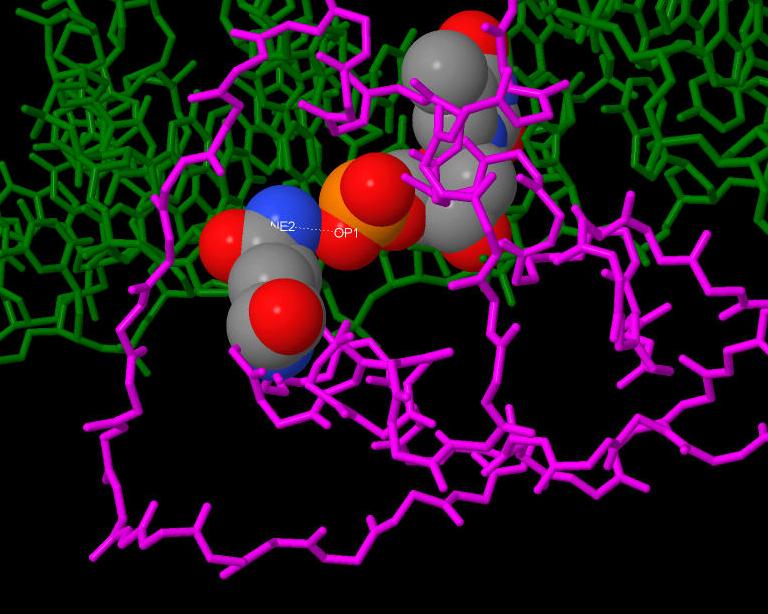
Вид сверху. Пунктирной линией показан контакт ДНК с аргинином. ДНК (зеленый) и белок (малиновый) показаны в проволочной модели. Боковые цепи скрыты. Основание [DG]1533 и остаток аминокислоты [GLU]1288 представлены с учётом ван-дер-ваальсовых радиусов и раскрашены по cpk.
[1] RNAfold online service
[2] Amino acid–base interactions: a three-dimensional analysis of protein–DNA interactions at an atomic level