А и В формы ДНК. Структура РНК
Задание 1
При помощи программы fiber из пакета 3DNA были получены файлы gatc_a.pdb, gatc_b.pdb, gatc_z.pdb, содержащие смоделированные структуры ДНК в А, В и Z формах соответственно.
А и В формы состояли из 5 повторений фрагмента GATC, Z форма — из 10 повторов GC.
Рис 1. Изображение форм ДНК

Задание 2
В этом задании полученные структуры сравнивались при помощи средств Jmol.
Упражнение 1
Были рассмотрены А и В формы ДНК, визуально найдены большая и малая бороздки. Затем для цитозина было определено, какие из атомов обращены к той или иной бороздке.
Рис 2. Выбранный цитозин в А и В формах.
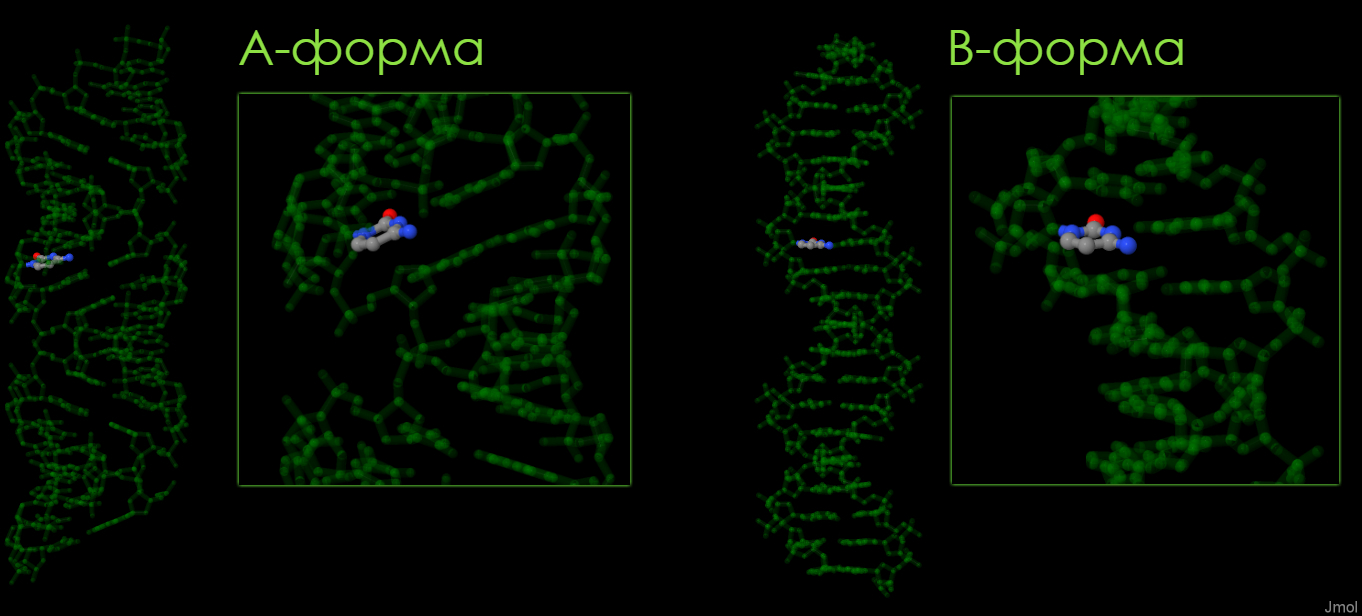
Результат можно видеть на Рис.3, где красным цветом обозначены атомы, обращенные к большой бороздке, синим — к малой. Изображение получено посредством MarvinSketch.
Рис 3. Цитозин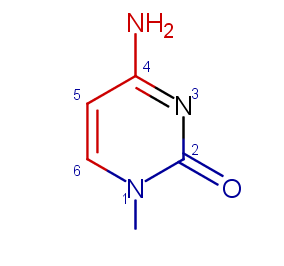 |
Резюме:
|
Упражнение 2
Измерение основных спиральных параметров разных форм ДНК было проведено в Jmol. Результаты представлены в Таблице 1.
Таблица 1
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали | 28.03 Å | 33.7 Å | 43.5 Å |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7.98 Å [G]29:B.P-[A]6:A.P | 17.21 Å [G]33:B.P-[G]5:A.P | 9.87 Å [G]17:A.P-[C]28:B.P |
| Ширина малой бороздки | 16.81 Å [T]15:A.P-[C]28:B.P | 11.69 Å [G]25:B.P-[C]20:A.P | 16.08 Å [C]12:A.P-[C]28:B.P |
Упражнение 3
После измерения торсионных углов (обозначены на схеме Рис.4) нуклеотида с цитозином данные сравнивались с содержащимися в презентации к лекции. По результатам была составлена Таблица 2.
Рис 4. Торсионные углы и их обозначения
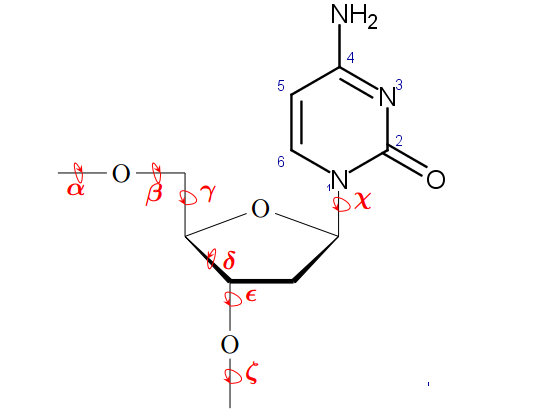
Таблица 2. Торсионные углы в А и В формах ДНК
| α | β | γ | δ | ε | ζ | χ | |
| A-форма (данные jmol) | 64.1° | 174.8° | 41.7° | 79.1° | -147.8° | -75.1° | -157.2° |
| A-форма (данные презентации) | 62° | 173° | 52° | 88/3 | 178° | -50° | -160° |
| В-форма (данные jmol) | 85.9° | 136.3° | 31.2° | 143.4° | -140.8° | -160.5° | -98° |
| В-форма (данные презентации) | 63° | 171° | 54° | 123/131 | 155° | -90° | -117° |
Задание 3
Работа с пакетом 3DNA, в частности, с программами find_pair и analyze.
Упражнение 1
Программы find_pair и analyze были объединены в конвейер, на вход которому подавался [pdb]-файл с определенной структурой.
find_pair -t file.pdb stdout | analyze
На выходе мы получали файл [out], содержащий информацию о структуре соединения.
Так, например, оттуда были извлечены данные о торсионных углах в А, В и Z-ДНК (из моделей, постороенных в Упр.1); в тРНК (PDBID=2dxi); в ДНК (PDBID=1by4). Затем данные для двух последних структур были проанализированы при помощи средств MS Excel c целью получения средних значений торсионных углов в реальных соединениях.
Файл [xlsx], в котором проводились расчёты, доступен для скачивания.
Итоговое сравнение торсионных углов приведено в Таблице 3.
Как видно, структура тРНК по торсионным углам наиболее близка к А-форме ДНК, нежели к иным.
Далее были определены самые "деформированные" нуклеотиды в структурах 2dxi и 1by4, то есть нуклеотиды, у которых значения торсионных углов сильно отличаются от средних. Они для наглядности также приведены в таблице.
Таблица 3. Средние значения и выбросы среди торсионных углов различных структур НК
| α | β | γ | δ | ε | ζ | χ | |
| Торсионные углы у смоделированных структур, ° | |||||||
| A-ДНК | -51,7 | 174,8 | 41,7 | 79,1 | -147,8 | -75,1 | -157,2 |
| В-ДНК | -29,9 | 136,4 | 31,1 | 143,4 | -140,8 | -160,5 | -98 |
| Z-ДНК (С) | -139,5 | -136,8 | 50,8 | 137,6 | -96,5 | 82 | -154,3 |
| Z-ДНК (G) | 52 | 179 | -173,8 | 94,9 | -103,6 | -64,8 | 58,7 |
| Значения торсионных углов у тРНК 2dxi, ° | |||||||
| Средние | -32,4 | 74,6 | 47,5 | 85,9 | -134,0 | -55,1 | -137,2 |
| Деформированный А41-нуклеотид | 126,5 | -104,8 | -160,7 | 100 | 88,2 | 88,6 | -129,5 |
| Значения торсионных углов у ДНК 1by4, ° | |||||||
| Средние | 10,6 | 16,6 | 12,3 | 143,0 | -98,0 | -78,4 | -109,2 |
| Деформированный G11-нуклеотид | -78,2 | 163,8 | 79,2 | 89,1 | -151,3 | -103,6 | -168,2 |
Упражнение 2
Определение водородных связей в структуре тРНК.
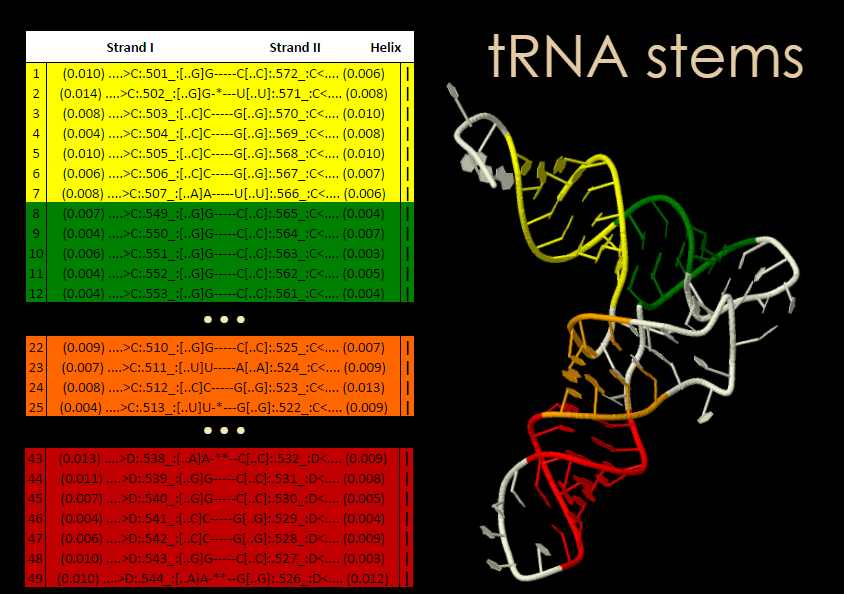
Информация о номерах нуклеотидах в стемах (стеблях) тРНК была извлечена из [out]-файла для структуры 2dxi. Для этого нужно было рассматривать те участки, на которых остатки обеих цепей идут согласно последовательности их номеров. Остальные же участки стемами не являются.
Получено 4 стема, каждый из которых окрашен в свой цвет (остальные же элементы структуры тРНК показаны в белом цвете). Соответствующие номера нуклеотидов можно увидеть на Рис. 5
- жёлтый: акцепторный стебель
- зелёный: T-стебель
- оранжевый: D-стебель
- красный: антикодоновый стебель
Рис 5. Стебли тРНК в структуре 2dxi
Наличие в структуре неканонических пар указано в файле [out]:
Strand I Strand II Helix 2 (0.014) ....>C:.502_:[..G]G-*---U[..U]:.571_:C<.... (0.008) | 14 (0.007) ....>C:.555_:[..U]U-**+-G[..G]:.518_:C<.... (0.008) x 15 (0.007) ....>C:.538_:[..A]A-**--C[..C]:.532_:C<.... (0.004) | 21 (0.006) ....>C:.544_:[..A]A-**--G[..G]:.526_:C<.... (0.008) | 25 (0.004) ....>C:.513_:[..U]U-*---G[..G]:.522_:C<.... (0.009) |
Стабилизирующие водородные связи могут быть определены при рассмотрении комплементарных пар, не входящих в структуру стеблей:
13 (0.005) ....>C:.554_:[..U]U-**--A[..A]:.558_:C<.... (0.003) | 14 (0.007) ....>C:.555_:[..U]U-**+-G[..G]:.518_:C<.... (0.008) x 15 (0.007) ....>C:.538_:[..A]A-**--C[..C]:.532_:C<.... (0.004) | 26 (0.007) ....>C:.514_:[..A]A-**--U[..U]:.508_:C<.... (0.007) | 27 (0.021) ....>C:.515_:[..G]G-**+-C[..C]:.548_:C<.... (0.011) x 28 (0.006) ....>C:.519_:[..G]G-----C[..C]:.556_:C<.... (0.002) +
Упражнение 3.
Нахождение возможных стекинг-взаимодействий.
В [out]-файле содержатся данные о площадях перекрывания для каждых двух последовательных пар оснований.
Одни из наибольших и наименьших площадей перекрывания у пар соответственно:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 2 GC/GU 7.07( 4.32) 0.00( 0.00) 0.00( 0.00) 6.67( 4.16) 13.74( 8.48) 14 UA/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Структура всех таких имеющихся пар записывается в файл stacking.pdb в виде отдельных моделей. Изображения были получены или посредством Jmol, или же программой stack2img, на вход которой подавалась вырезанная при помощи ex_str модель в формате [pdb]. (Затем полученный [ps]-файл был конвертирован в [png]-формат).
Выделение модели номер n в отдельный файл из stacking.pdb:
ex_str -n stacking.pdb step_n.pdb
Создание изображения по модели step_n.pdb с помошью stack2img:
stack2img -cdolt step_n.pdb step_n.ps
| Пары с максимальной площадью перекрывания GC/GU | |
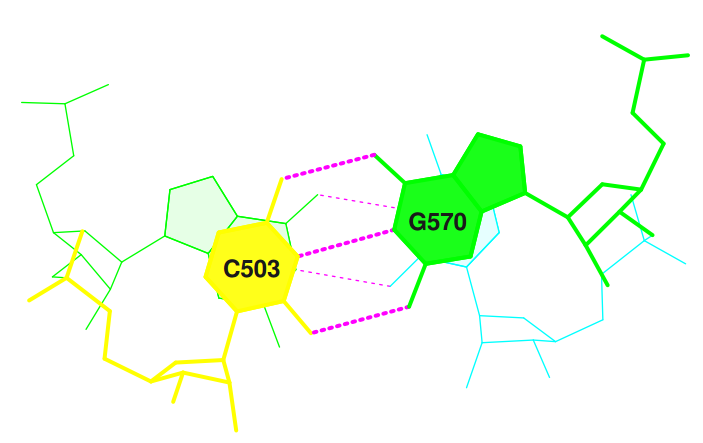 | 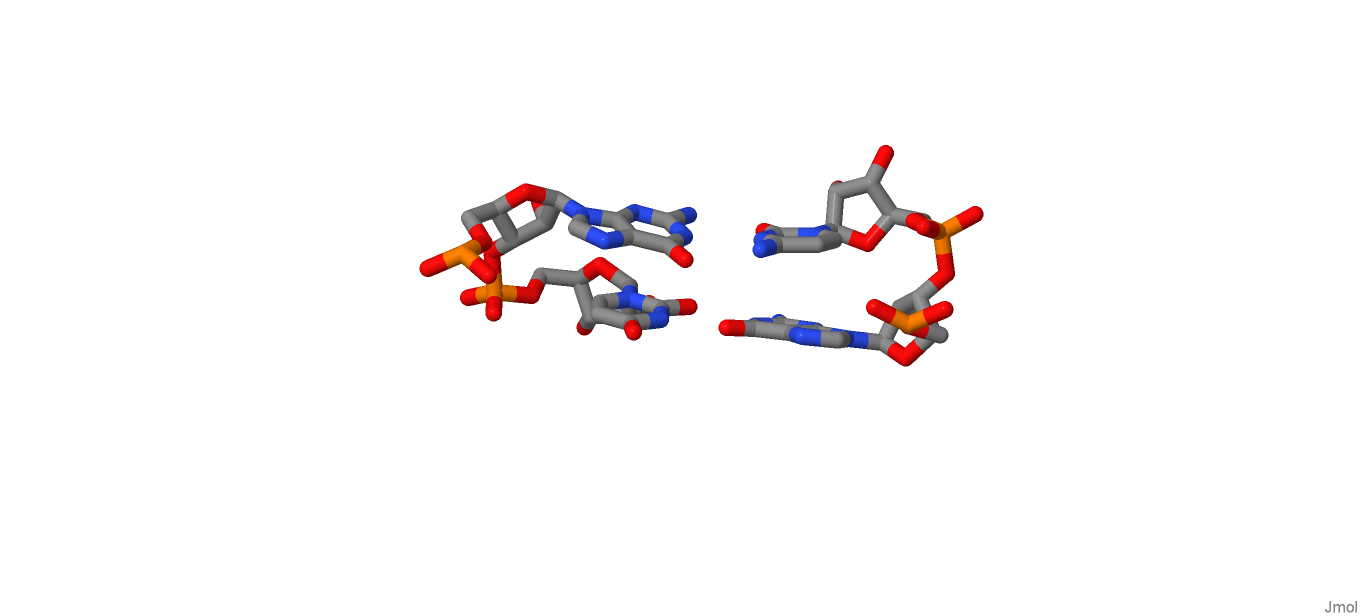 |
| stack2img | Jmol |
| Пары с минимальной площадью перекрывания UA/CG | |
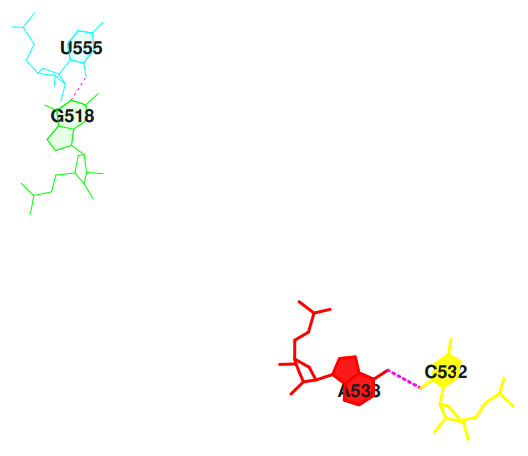 | 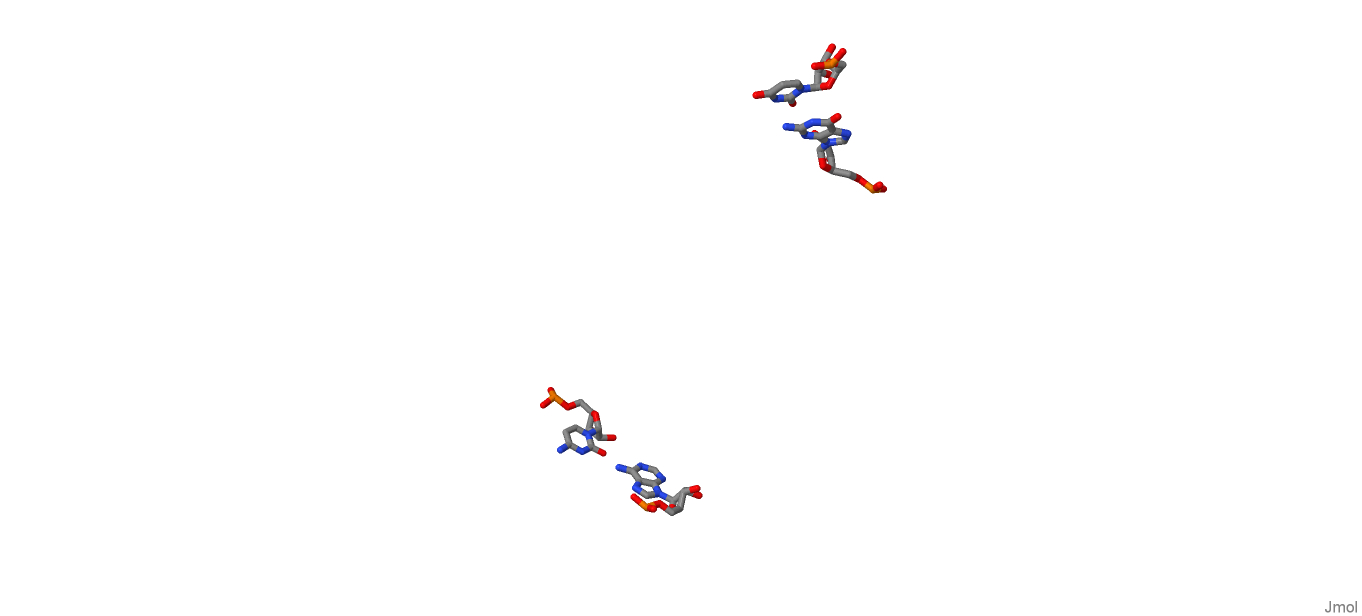 |
| stack2img | Jmol |