Укоренение в среднюю точку
Для реконструкции филогенетического древа было выбрано семейство белков RSMH — 16S рРНК-метилтрансферазы H; последовательности же соответствующих белков разных бактерий отдела фирмикут (см. Практикум 1) были получены из базы данных Uniprot, после чего выровнены посредством программы Jalview с использованием сервера Muscle (with defaults). Полученное выравнивание приведено на Рис. 1.
Рис. 1. Выравнивание белков семейства RSMH

На основании приведенного выравнивания в программе Jalview было реконструировано филогенетическое древо, для чего использовался метод Neighbor Joining Using % Identity. Результат представлен на Рис. 2, 3.
| Рис. 2. Jalview: NJ using % identity | 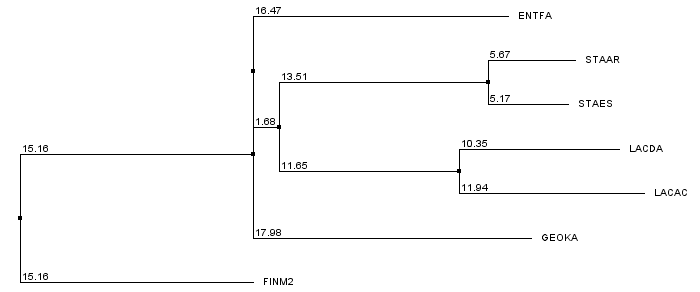 |
| Рис. 3. MEGA-изображение по newick-формату |
Переукоренение данного дерева было произведено при помощи программы retree из пакета PHYLIP. Для этого дерево из Jalview сохранялось в newick-формате в файле intree, который, находясь в одной директории с retree.exe, подается данной программе на вход, после чего возможно так называемое переукоренение в среднюю точку (клавиша M).
Этот метод укоренения основывается на поиске максимального расстояния между двумя листьями и размещении корня в середине данного пути. Укорененное дерево приведено на Рис. 4.
| Рис. 4. Укоренение в среднюю точку | |
| Рис. 5. Эталонное дерево | 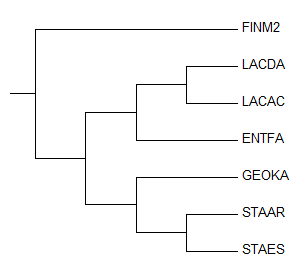 |
Укоренение произошло в ветвь {FINM2} против {GEOKA, LACAC, LACDA, STAAR, STAES, ENTFA}. Стоит отметить, что дерево, построенное Jalview изначально отличается от эталонного своей
неразрешенностью. Поэтому, строго говоря, укоренение произошло верно, но реконструированное дерево
в силу своей небинарности должно рассматриваться как множество возможных разрешений, и среди них имеется вариант, соответствующий эталонному бинарному дереву.