Отчет за практикум 9
Для выполнения заданий я выбрала домен Ig_5 (PF16681).
Число белков с этим доменом в выборках seed - 18, full - 294 и Uniprot - 647
Средняя длина домена - 78.6 aa
Cреднее сходство - 36 %
Cредний процент покрытия последовательности белка доменом (coverage) - 40.61 %
Затем я выбрала двухдоменную архитектуру, состоящую из Ig_5 и ITAM. Белков с данной архитектурой - 83.

Я скачала последовательности из выборки full в формате fasta (Erina_full-294.fasta), список AC белков архитектуры и создала файл с ее последовательностями. Затем я выровняла последовательности программой muscle3 и провела ревизию. В результате ревизии количество последовательностей было сокращено до 28. Результат - domain_align_r.fa.
Для создания HMM профиля я использовала следующие команды:
hmm2build hmm domain_align_r.fa
hmm2calibrate hmm
hmm2search --cpu=1 hmm Erina_full-294.fasta > result.txt
Получившиеся файлы: hmm, result.txt
Из получившегося в результате работы hmm2search файла (result.txt) я сделала таблицу с результатами поиска по профилю. Как мне удалось выяснить из документации, столбец Domain показывает номер домена из обнаруженных, seq-f и seq-t - начальную и конечную точку выравнивания в последовательности, hmm-f и hmm-t - начальную и конечную точку выравнивания в hmm профиле. Также в таблицу были добавлены колонки: отметка, совпадает ли доменная архитектура с выбранной; чувствительность при данном пороге; единица минус специфичность.
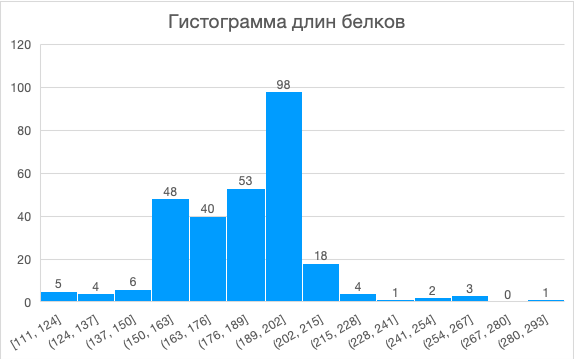
С помощью Excel я построила гистограмму распределения длин белков (Рис. 2). Получилось, что для семейства, содержащего выбранный домен наиболее характерна длина белков от 189 до 202.