Исследование ДНК-белковых взаимодействий в структуре комплекса 1RIO
1. Краткое описание структуры в файле 1RIO.pdb
В файле приведены координаты атомов ДНК (цепи U, T), белка сигма-фактора SigA (цепь H) (выделены из THERMUS AQUATICUS); N-концевого домена белка-репрессора CI (цепи A,B) (из ENTEROBACTERIA PHAGE LAMBDA), иона кальция (301, цепь А), (4с)-2-метил-2,4-пентандиола (201, цепь А) и молекул воды (176 атомов).
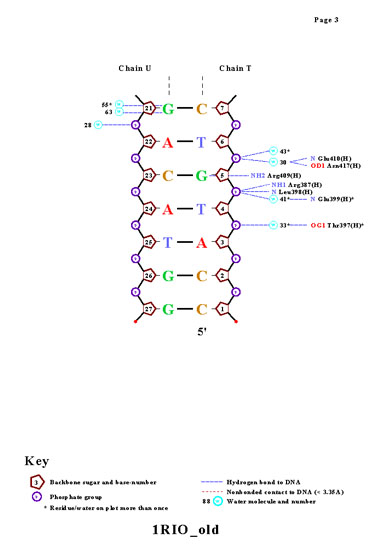
Для исследования были выбраны цепь H белка и цепи U, T, представляющие ДНК со следующей последовательностью:
цепь U [1] 5' - C-G-G-T-A-T-C-A-C-C-G-C-C-A-G-T-G-C-T-T-G-A-C-A-T- G-G - 3' [27]
цепь T [27] 3' - G-C-C-A-T-A-G-T-G-G-C-G-G-T-C-A-C-G-A-A-C-T-G-T-A-C-C - 5' [1],
где 1 и 27 - номера первого и последнего нуклеотида.
2. Функции белка, структура которого представлена в файле 1RIO.pdb
Сигма-фактор - это субъединица РНК-полимеразы, необходимая для инициации синтеза РНК бактерий, обеспечивает связывание РНК-полимеразы с промотором гена, отсоединяется по завершению транскрипции.
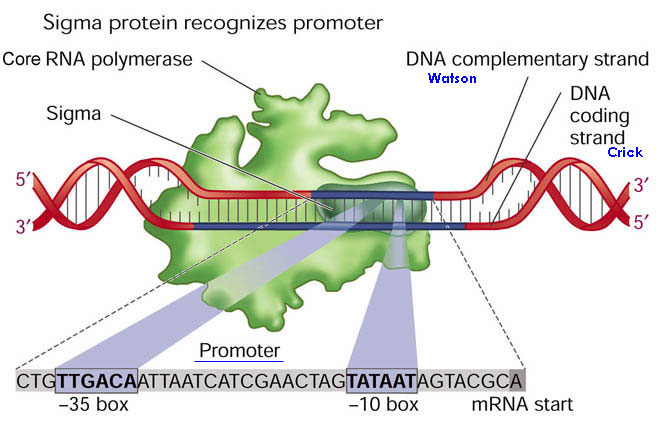
3. Исследование структуры ДНК
С помощью программ find_pair и analyze определила тип формы ДНК (B-ДНК):
Classification of each dinucleotide step in a right-handed nucleic acid
structure: A-like; B-like; TA-like; intermediate of A and B, or other
cases
step Xp Yp Zp XpH YpH ZpH Form
1 CG/CG -2.83 8.71 -0.42 -1.81 8.71 0.24 B
2 GG/CC -3.21 8.93 0.17 -4.65 8.78 1.61 B
3 GT/AC -3.03 9.04 0.40 -3.97 9.04 -0.30 B
4 TA/TA -2.74 9.08 0.42 -2.68 9.04 -0.93 B
5 AT/AT -3.02 9.40 -0.10 -5.46 9.13 2.19 B
6 TC/GA -1.54 9.33 0.11 -0.38 9.24 -1.31 B
7 CA/TG -3.26 9.03 -0.15 -5.44 8.73 2.30 B
8 AC/GT -2.78 8.92 1.15 -5.55 8.75 2.10
9 CC/GG -3.21 9.21 0.94 -8.77 8.44 3.83
10 CG/CG -3.13 8.73 -0.75 0.55 8.13 -3.37
11 GC/GC -2.91 9.00 -0.83 -1.58 8.91 -1.51 B
12 CC/GG -3.16 9.06 -1.15 -3.04 9.03 1.36 B
13 CA/TG -2.18 8.49 -0.90 0.81 8.32 -1.91
14 AG/CT -3.01 9.03 -0.45 -4.25 8.85 1.89 B
15 GT/AC -3.06 9.03 -0.02 -5.56 8.92 1.38 B
16 TG/CA -2.72 8.78 -0.65 -1.34 8.80 -0.32 B
17 GC/GC -3.46 9.31 -0.32 -4.20 9.31 -0.21 B
18 CT/AG -2.82 8.71 0.77 -5.10 8.17 3.16
19 TT/AA -3.20 8.85 -0.30 -3.28 8.86 -0.13 B
20 TG/CA -2.95 9.03 -0.37 -3.41 8.87 1.74 B
21 GA/TC -3.45 9.25 -0.46 -4.35 9.26 0.34 B
22 AC/GT -3.62 9.23 0.05 -7.24 9.03 1.92 B
23 CA/TG -3.11 8.99 -0.01 -4.78 8.74 2.10 B
24 AT/AT -3.55 8.95 -1.04 -4.96 9.01 0.30 B
25 TG/CA -2.02 8.59 -0.82 0.62 8.41 -1.94
26 GG/CC -3.06 9.18 -1.01 -5.68 9.08 1.70
Cредние значения торсионных углов для внутренних нуклеотидов - dna.xlsx
Cамыми "кривыми" мне показались, нуклеотиды, выделенные красным в excel-файле - t6:t и a22:u, образующие комплементарную пару в ДНК.
По моему мнению, связывание с белком должно приводить к деформации ДНК. В данном случае, понаблюдав структуру в RasMol, получаю:
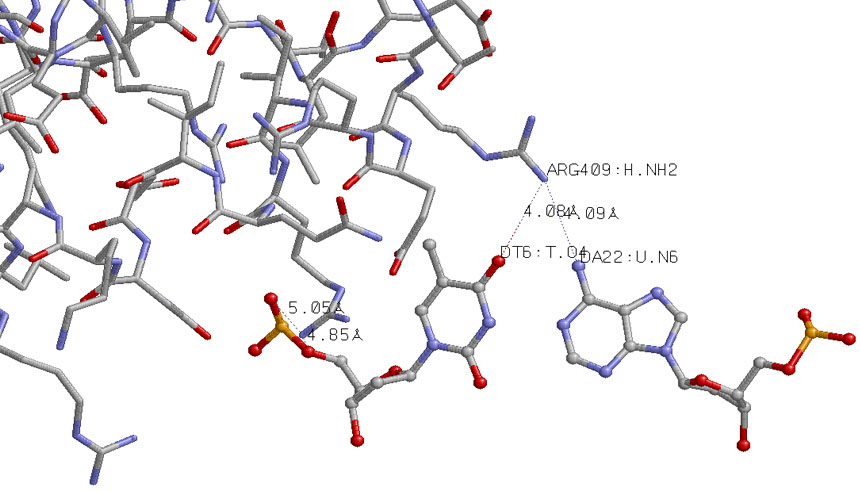
Расстояния велики для полярных взаимодействий, вероятно, непосредственные контакты между данными нуклеотидами и белком отствутвуют. Arg409, указанный на рисунке, скорее всего взаимодейсвует с g5:t - соседним нуклеотидом.
4. Исследование природы ДНК-белковых контактов
Скрипт с определениями заданных множеств и с последовательными изображениями структуры - ex1.spt (вспомнить, как с помощью команды define RasMol задавать множества атомов).
Определяю ДНК-белковые контакты с помощью следующих множеств:
define set1 *.O?* and dna
echo set1 - oxygen in 2'-desoxyriboses
define set2 *.O?P and dna
echo set2 - oxygen in phosphates
define set3 nitrogen and dna
echo set3 - nitrogen in bases
define set4 *.C?* and dna
echo set4 - carbon in 2'-desoxyriboses
define set5 *.P and dna
echo set5 - phosphorus
define pol oxygen, nitrogen
echo pol - polar atoms
define nonpol carbon, sulphur, phosphorus
echo nonpol - nonpolar atoms
define polmajg G?.O6, T?.O4, *.N7, A?.N1, G?.N1, C?.N4, *.N6 and dna
echo major groove's polar atoms
define nonpolmajg *.C5, A?.C6, G?.C6, *.C8, (*.C4 and pyrimidine) and dna
echo major groove's nonpolar atoms
define polming C?.O2, C?.N1, T?.O2, *.N3, G?.N2 and dna
echo minor groove's polar atoms
define nonpolming *.C2, *.C1, (*.C4 and purine) and dna
echo minor groove's nonpolar atoms
Скрипт для описания ДНК-белковых контактов - ex2.spt.
Таблица. Контакты разного типа в комплексе 1RIO.pdb
Контакты атомов белка (цепь H) с |
Полярные |
Неполярные |
Всего |
остатками 2'-дезоксирибозы |
0 |
4 |
4 |
остатками фосфорной кислоты |
8 |
11 |
19 |
остатками азотистых оснований со стороны большой бороздки |
2 |
4 |
6 |
остатками азотистых оснований со стороны малой бороздки |
0 |
0 |
0 |
В данном случае число неполярных взаимодействий превышает число полярных более чем в два раза. Это связано с тем, что полярных атомов меньше, и большинство из них скрыто внутри молекул. Не обнаружено взаимодействий между белком и остатками азотистых оснований со стороны малой бороздки.
5. Получение популярной схемы ДНК-белковых контактов с помощью nucplot
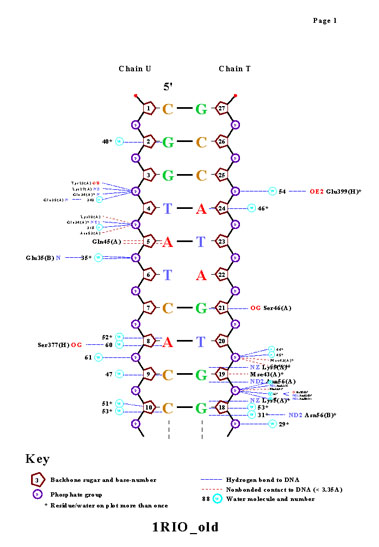
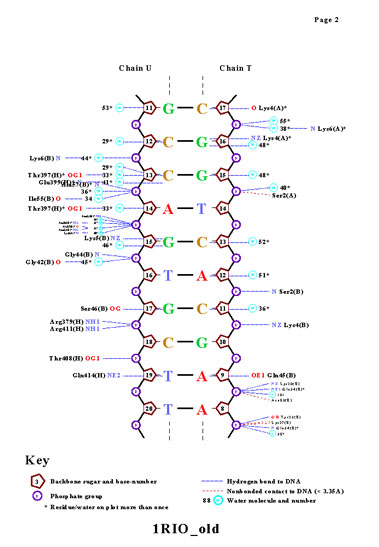
На схеме изображены контакты ДНК со всем белком, в таблице приведены данные только для H цепи (как было указано в задании), поэтому представлено гораздо больше контактов. Преобладают водородные связи, наибольшее число контактов ДНК с белком приходится на фосфатные группы, приведено довольно много взаимодействий с водой.
Также видно, что пара "кривых" нуклеотидов t6:t и a22:u, описываемых в пункте 3, не взаимодействует с молекулами белка или воды (за исключением фосфата), в отличие от соседних пар. Этим можно объяснить полученное ранее отклонение от средних значений торсионных углов для внутренних нуклеотидов.
Glu399 и Thr397 - аминокислотные остатки с наибольшим числом указанных на схеме контактов - 3. Но они координируют молекулы воды, а не напрямую взаимодействуют с атомами ДНК:
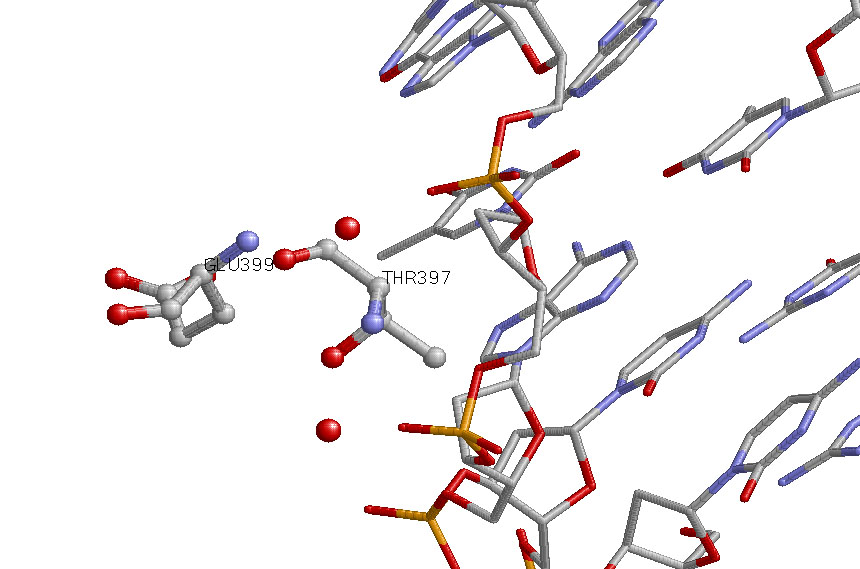
6. Возможные распознающие контакты
Аминокислотные остатки наиболее важные для распознавания последовательности ДНК:
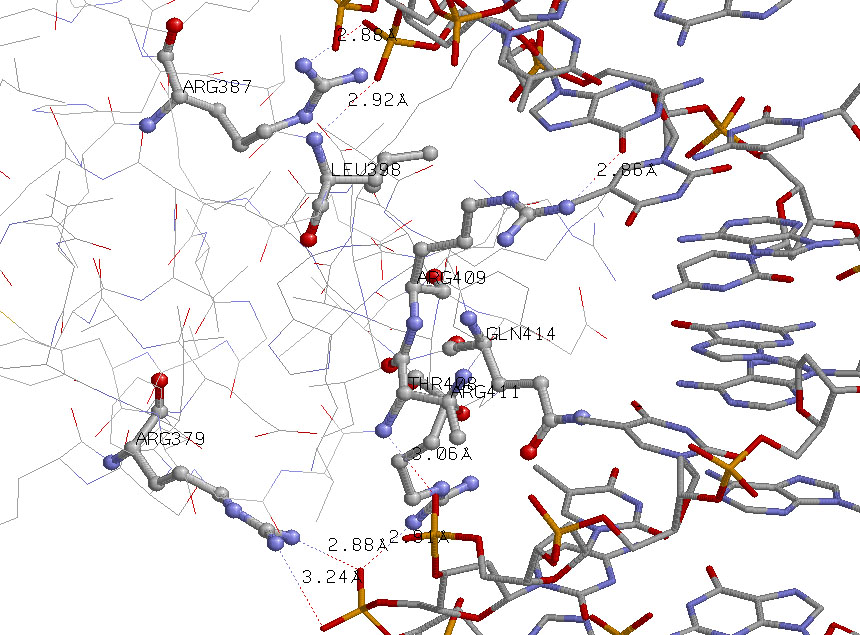
Выделенные а.о. непосредственно взаимодействуют с фосфатами или азотистыми основаниями. Для остальных остатков цепи H вовсе не характерны прямые взаимодействия с ДНК, поэтому данные а.о. наиболее важны для распознавания. Но только два а.о. взаимодействуют с атомами азотистых оснований - Arg409 и Gln414. Из них меньшая длина связи характерна для первого а.о., поэтому его в данном случае я считаю наиболее важным:
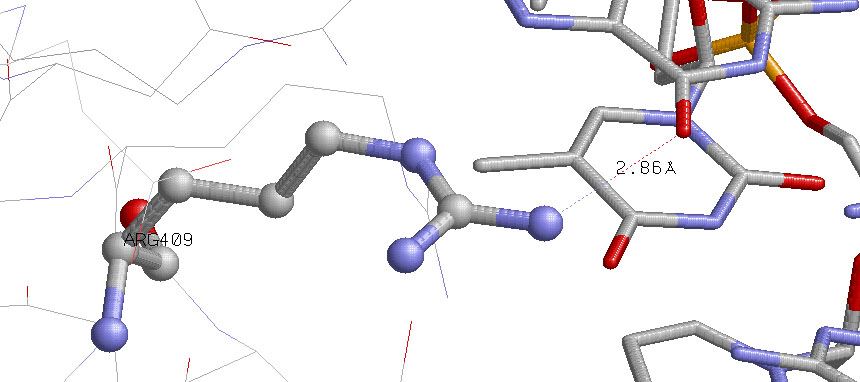
7. Характеристика ДНК-связывающего домена (UniProt ID Q9EZJ8_THEAQ)
С помощью инструментов Pfam определена доменная структура цепи H белка из исследуемого комплекса (UniProt entry Q9EZJ8_THEAQ):

Домены:
Sigma70_r1_2 (PF00140), 93-129;
Sigma70_r2 (PF04542), 202-272 - наиболее консервативный домен, включает спираль для узнавания промотера (-10) и фрагмент, необходимый для узнавания РНК-полимеразой;
Sigma70_r3 (PF04539), 281-360 - учавствует в связывании с РНК-полимеразой;
Sigma70_r4 (PF04545), 372-425 - учавствует в связывании с промотером (-35) с помощью мотива спираль-поворот-спираль.
© Eugenia Prokhorova 2011