Пояснения: первая последовательность - самая лучшая находка, вторая и третья - прямая и обратная прочтенная последовательность, две последних - другие найденные последовательности от родственных видов.
Моя последовательность кодирует субъединицу I цитохром оксидазы (ген CO1) в митохондриях вида Ophiopholis aculeata (офиура , смотри фотографии).




Данный организм относится к отряду Ophiurida (Настоящие офиуры, с простыми неветвящимися лучами).
Офиуры, или змеехвостки ( Ophiuroidea по латыни) — класс донных морских животных из типа иглокожих. Своё название получили за своеобразный способ передвижения: когда они ползут по дну, их «руки» извиваются, как змеи. Офиуры — самый разнообразный класс иглокожих, включающий около 2 тысяч видов, большинство из которых — обитатели тропических вод (в России встречается 120 видов). Известны в ископаемом состоянии с раннего ордовика.
Офиуры обитают на морском дне повсеместно, на глубине до 6—8 км. Большинство встречается на глубине более 500 м; населяют и коралловые рифы. Ползают по дну посредством изгибания лучей или зарываются в грунт. У некоторых офиур руки ветвятся, и скопление питающихся офиур напоминает настоящий ковёр из щупалец. В нём застревают или активно захватываются мелкие животные (черви, ракообразные, медузы) и взвешенные частицы пищи (планктон). Офиуры по характеру питания являются слизисто-ресничными фильтраторами; имеются также офиуры-детритофаги и трупоеды. У офиур развита способность к регенерации лучей, но если отсечь все лучи от диска, животное умрёт. Офиуры часто образуют массовые поселения и служат пищей рыбам. Некоторые офиуры живут на водорослях, губках, кораллах и морских ежах.
Офиуры обычно раздельнополы, но имеются и виды-гермафродиты. Лишь немногие способны размножаться и бесполым путём — разделением надвое, с последующим восстановлением недостающих частей. Развитие происходит в большинстве случаев с метаморфозом, при этом образуется свободноплавающая личинка — офиоплутеус. Реже развитие прямое.
Информация взята со всем известного сайта.
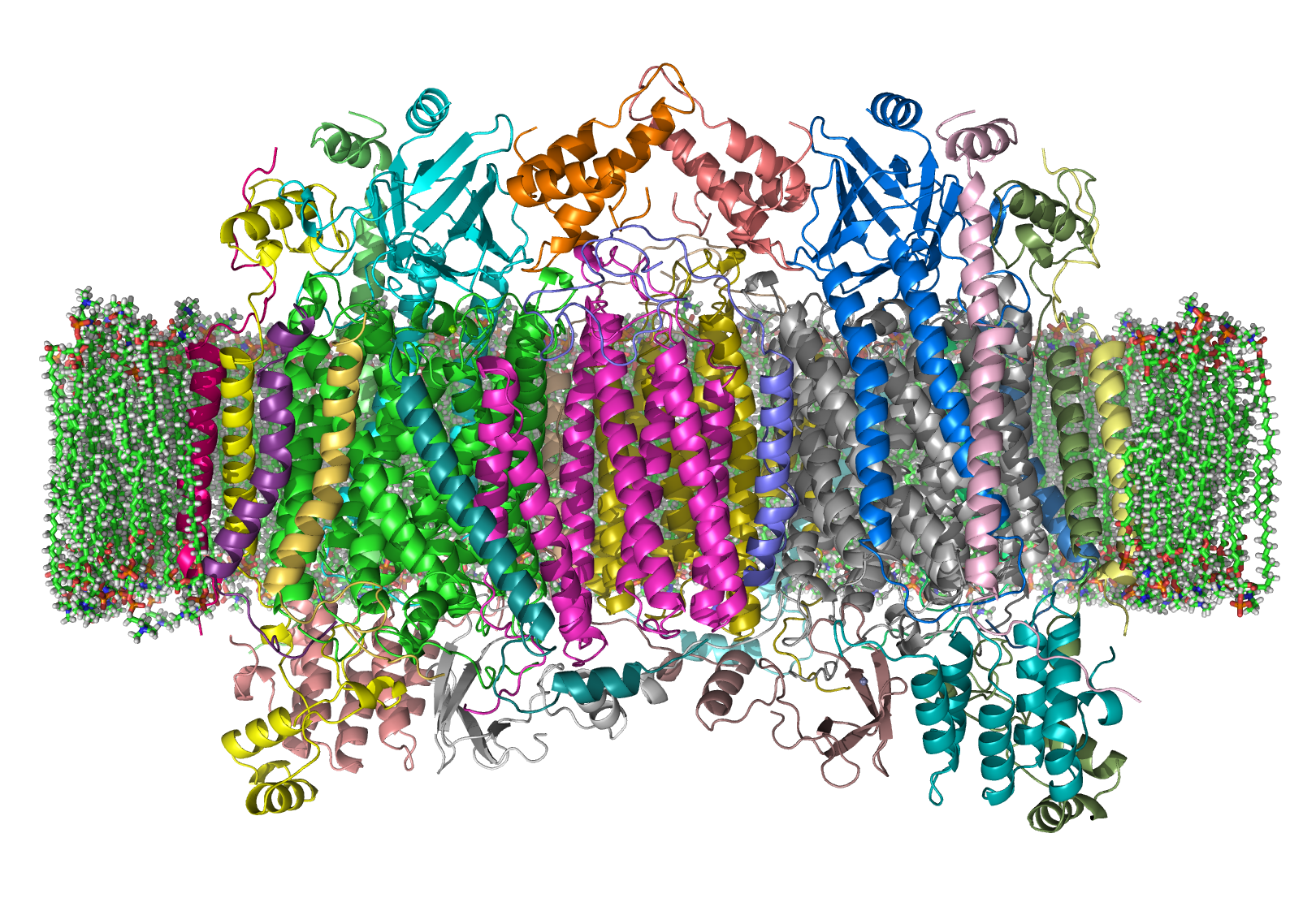
Цитохром с-оксидаза (или комплекс IV, или цитохромоксидаза) - чрезвычайно важный белок, являющийся терминальной оксидазой цепи переноса электронов в митохондриях. Она катализирует окисление кислорода (донор электронов - цитохром а, продукт реакции - вода).
Цитохромоксидаза вносит свой вклад в создание протонного градиента для синтеза АТФ и является частью пути окислительного фосфорилирования. Кроме того, этот мультибелковый комплекс играет ключевую роль в регуляции активности всей дыхательной цепи и производстве энергии эукариотической клеткой.
Субъединица I cвязывает гем и гем а3, содержит Cu и имеет протонные каналы, гомологична бактериальной.
Последовательность участка гена субъединицы I цитохром с-оксидазы (длиной порядка 600 нуклеотидов) широко используется в проектах, связанных с ДНК-баркодированием — определением принадлежности организма к тому или иному таксону на основе коротких маркеров в его ДНК.
Сравнение списков находок 3-я разными алгоритмами blast
Я использовал уже знакомую последовательность.
Для поиска я решил ограничиться одним родом (Ophiopholis).
| Параметры запуска BLAST (по умолчаниию для каждого алгоритма): | |||||||
|---|---|---|---|---|---|---|---|
| Database | Max Target Sequences | Expect Threshold | Word Size | Max matches | Match/Mismatch Scores | Gap Costs | |
| megablast | Nucleotide collection (nr/nt) | 100 | 10 | 28 | 0 | 1, -2 | Linear |
| discontiguous megablast | Nucleotide collection (nr/nt) | 1000 | 10 | 11 | 0 | 2, -3 | Existence:5, Extention: 2 |
| blastn | Nucleotide collection (nr/nt) | 1000 | 10 | 11 | 0 | 2, -3 | Existence:5, Extention: 2 |
| Результаты сравнения алгоритмов: | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Алгоритм | Число находок | Score лучшей находки | Score худшей находки | E-value лучшей находки | E-value худшей находки | Ident лучшей находки | Ident худшей находки | Query cover лучшей находки | Query cover худшей находки |
| MEGABLAST | 49 | 1173 | 564 | 0.0 | 1e-162 | 99% | 94% | 96% | 82% |
| DISCONTIGUOUS MEGABLAST | 65 | 1148 | 432 | 0.0 | 4e-123 | 99% | 80% | 96% | 68% |
| BLASTN | 65 | 1148 | 432 | 0.0 | 4e-123 | 99% | 80% | 96% | 68% |
Можно сделать вывод, что алгоритмы BLASTN и DISCONTIGUOUS MEGABLAST при сходных вводных данных дают близкое число находок сходного качества.
MEGABLAST отсеивает большее число находок, выдаваемые им последовательности ближе к исходной. Он предъявяет относительно высокие требования к E-value, а Query cover не может быть ниже 28 нуклеотидов, поэтому этот алгоритм может не найти короткие последовательности.
DISCONTIGUOUS MEGABLAST действует аналогично MEGABLAST, но быстрее.
BLASTN лучше всего использовать для поиска схожих нуклеотидных последовательностей с целью определения таксономии и функции.
Пример последовательности, найденный только МEGABLAST.
Пример последовательности, найденный только DISCONTIGUOUS MEGABLAST и BLASTN.
Наличие гомологов трех белков в геноме одного организма
Возьмем сборку генома организма X5 (Amoboaphelidium protococarum)
Для начала необходимо создать локальную базу данных (команда makeblastdb -in X5.fasta -dbtype nucl ).
Затем запустим алгоритм TBLASTN, который по последовательности аминокислот будет искать гомологи нуклеиновых кислот (команда tblastn -query xxxx.fasta -db X5.fasta > xxxx.out).
TERT_HUMAN
Теломераза — фермент, добавляющий особые повторяющиеся последовательности ДНК (TTAGGG у позвоночных) к 3'-концу цепи ДНК на участках теломер, которые располагаются на концах хромосом в эукариотических клетках.
Теломераза состоит из теломеразной обратной транскриптазы (TERT), теломеразной РНК (hTR или TERC), и дискерина (по две молекулы каждого из этих веществ). Две субъединицы фермента кодируются двумя различными генами.
TERT — это обратная транскриптаза, то есть фермент, создающий одноцепочечную ДНК на основе шаблонной одноцепочечной РНК.
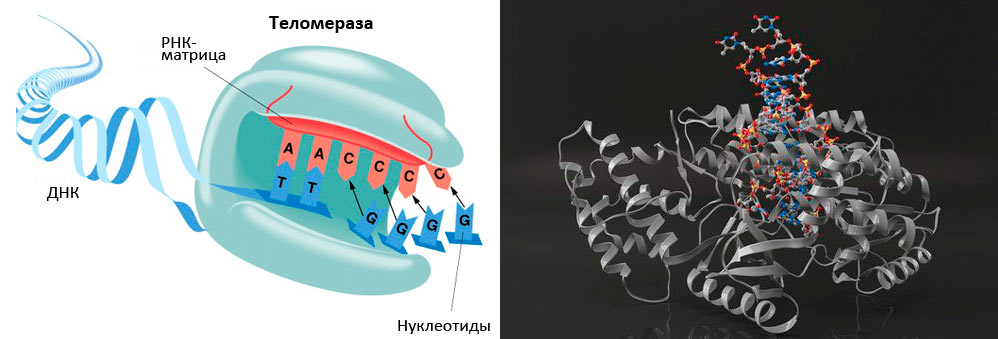
Теломераза экспрессируется в стволовых, половых и некоторых других типах клеток организма, которым необходимо постоянно делиться для функционирования определённых тканей (например, клетки эпителия кишечника). Обычные соматические клетки организма лишены теломеразной активности. Клетки 85 % раковых опухолей обладают теломеразной активностью, поэтому считается, что активация теломеразы является одним из событий на пути клетки к злокачественному перерождению.
Результаты tblastn по базе данных X5.fasta для TERT_HUMAN:
Для лучшей нахоки сходство составляет 27% при длине 576 нуклеотидов. К тому же при длине теломеразной обратной транскриптазы в 1132 аминокислотных остатка мы видим очень малый процент перекрывания (50.2 %). Хоть в данном выравнивании и присутствуют консервативные участки, но утверждать о гомологии из-за этого нельзя
Скорее всего данный организм имеет обратную транскриптазу, но она очень сильно отличается от человеческого аналога (гомологичны лишь несколько консервативных участков).
RPB1_HUMAN
Перед нами субъединица эукариотической ДНК-зависимой РНК-полимеразы II.
Мне не удалось найти картинку только первой субъединицы, поэтому я представлю изображение всего фермента.
РНК-полимераза II катализирует транскрипцию ДНК, синтезирует предшественников мРНК и большинство мяРНК и микроРНК. Эта полимераза представляет собой комплекс массой 550 кДа и состоящий из 12 субъединиц. Является наиболее изученным типом РНК-полимеразы.
RPB1 самая большая субъединица РНК-полимеразы II. Её С-концевой домен, объединяющий до 52 гептапептидных повторов (YSPTSPS), которые необходимы для полимеразной активности. В сочетании с рядом других субъединиц полимеразы, она образует ДНК-связывающий домен полимеразы, в котором матрица ДНК транскрибируется в РНК.
Результаты tblastn по базе данных X5.fasta для RPB1_HUMAN:
Человеческий белок имеет длину 1970 нуклеотидов. Для лучшей находки длиной 1547 сходство составляет 52%, процент покрытия 79%. Находка является гомологом исходного человеского белка.
Гистон H4 серой крысы (Rattus norvegicus)
Гистоны — обширный класс ядерных белков, выполняющих две основные функции: они участвуют в упаковке нитей ДНК в ядре и в эпигенетической регуляции таких ядерных процессов, как транскрипция, репликация и репарация. Существует пять различных типов гистонов H1/Н5, H2A, H2B, H3, H4. Гистоны H2A, H2B, H3, H4, называемых коровыми гистонами, формируют нуклеосому, представляющую собой белковую глобулу, вокруг которой накручена нить ДНК. Гистон H1/H5, называемый линкерным гистоном, связывается с внешней стороной нуклеосомы, фиксируя на ней нить ДНК.
Благодаря высокому содержанию лизина и аргинина гистоны проявляют сильно оснoвные свойства. Гистоны непосредственно контактируют с ДНК и способны нейтрализовать отрицательный заряд фосфатных групп ДНК за счёт положительных зарядов аминокислотных остатков.
Последовательность аминокислот в этих белках является консервативной и практически не различается в организмах различных таксонов. Гистоны присутствуют в ядрах эукариотических клеток; у бактерий гистонов нет, но они выявлены у архей группы Euryarchaea
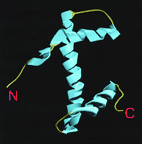
Общая структура для гистонов H2A, H2B, H3, H4. Три центральные а-спирали образующие фолд домен и свободные концевые участки подвергаемые различным модификациям.
Результаты tblastn по базе данных X5.fasta для H4:
Длина крысиного гистона 899 нуклеотидов. Программа нашла несколько гомологичных находок, почти все они имеют сходство 94%.
Можно утверждать, что Amoboaphelidium имеет гистоны, гомологичные гистонам позвоночных.
Ген белка, закодированный в одном скэффолде ''Amoboaphelidium''
Для начала нужно получить информацию а длинах котингов. Это можно сделать при помощи команды infoseq X5.fasta -only -name -length
Результат - список котингов. Нужно выбрать один контиг длины порядка десятков тысяч пар нуклеотидов. Интроны у амебоафилидум короткие, так что ген может поместиться в одном таком контиге
Чтобы получить последовательность, я применил команду seqret X5.fasta: scaffold-698 -out scA.fasta. Ccылка на результат.
Далее я провел поиск в BLAST по алгоритму BLASTN
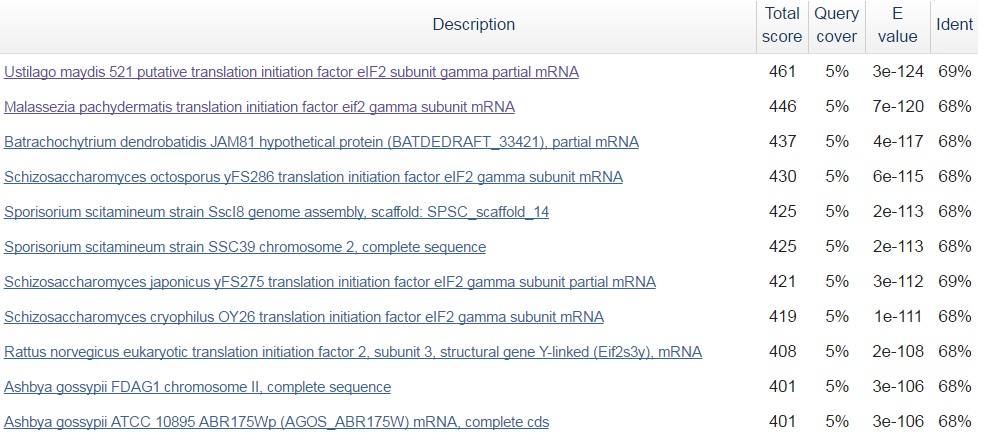
Мы можем наблюдать множество последовательностей схожей длины c покрытием 5%.
Cудя о всему этот ген кодирует гамма цепь фактора инициации трансляции eIF2
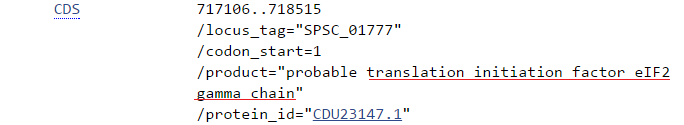
eIF2 является одним из важнейших факторов инициации трансляции и присутствует у всех эукарит. Его функция - взаимодействие с инициаторной тРНК.
Этот ген находится в самом начале нашего скэффолда, его координаты 1462-2793. Данные были получены с помощью BLASTX.
Alignment statistics for match #1
Score Expect Method Identities Positives Gaps
Frame
629 bits(1621) 0.0 Compositional matrix adjust. 322/446(72%) 364/446(81%) 3/446(0%)
-1
Query 2793 VDVSKLTPLSPEVISN*ATINIGTIGHVAHGKSTVVKAISGV*TVRHKTELERNITIKLG 2614
+DV+KLTPLS EVIS ATINIGTIGHVAHGKSTVVKAISGV TVR K ELERNITIKLG
Sbjct 23 LDVTKLTPLSHEVISRQATINIGTIGHVAHGKSTVVKAISGVHTVRFKNELERNITIKLG 82
Query 2613 YANAKIYECDQ--CPRPLKYKSFRSDKEVQFKCEEPNCGGTMHLKRHVSFVDCPGHDILM 2440
YANAKIY+ D CPRP Y+S S +F + P G L RHVSFVDCPGHDILM
Sbjct 83 YANAKIYKLDDPSCPRPECYRSCGSSTPDEFPTDIPGTKGNFKLVRHVSFVDCPGHDILM 142
Query 2439 ATMLNGAAVMDAAMLLIAGNESCP*P*TSEHLAAIEIMKLNHVIILQNKIDLISE**AAE 2260
ATMLNGAAVMDAA+LLIAGNESCP P TSEHLAAIEIMKL H++ILQNKIDL+ E A E
Sbjct 143 ATMLNGAAVMDAALLLIAGNESCPQPQTSEHLAAIEIMKLKHILILQNKIDLVKESQAKE 202
Query 2259 *YKSILKFINGTVAQKAPIVPISA*MKYNIDAVNEYICKKVPVPVRDFTAPARLIVIRSF 2080
Y+ IL F+ GTVA+ API+PISA +KYNI+ V EYI KK+PVP RDFT+ RLIVIRSF
Sbjct 203 QYEQILAFVQGTVAEGAPIIPISAQLKYNIEVVCEYIVKKIPVPPRDFTSEPRLIVIRSF 262
Query 2079 DVNKPGTEY*DLKGGVAGGSIL*GCLKLGDEIEVRPGIVSKDSEGKFRVRPILSRIVSLH 1900
DVNKPG E DLKGGVAGGSIL G LK+G EIEVRPGIVSKDSEGK +PI S+IVSL
Sbjct 263 DVNKPGCEVDDLKGGVAGGSILKGVLKVGQEIEVRPGIVSKDSEGKLMCKPIFSKIVSLF 322
Query 1899 TE*NHLQFAVPGGLIGVGTQIDPTVCRSDRLVGQVLGARGTLPEIYTELEITYFLLRQLL 1720
E N LQ+A PGGLIGVGT+IDPT+CR+DR+VGQVLGA G LPEI+TELEI+YFLLR+LL
Sbjct 323 AEHNDLQYAAPGGLIGVGTKIDPTLCRADRMVGQVLGAVGALPEIFTELEISYFLLRRLL 382
Query 1719 GVRSTTENK*LTKVTKLAKNETLMVNIGSTTAGCKVLNVKADMAKIYLTVPCCTEIGEKV 1540
GVR+ + K KV KL+KNE LMVNIGS + G +V VKAD+ KI LT P CTE+GEK+
Sbjct 383 GVRTEGDKK-AAKVQKLSKNEVLMVNIGSLSTGGRVSAVKADLGKIVLTNPVCTEVGEKI 441
Query 1539 ALSRRIDRHWRLIGWGKITRGVTIEP 1462
ALSRR+++HWRLIGWG+I RGVTI+P
Sbjct 442 ALSRRVEKHWRLIGWGQIRRGVTIKP 467
В данном случае subject - мой белок (гамма-цепь elF4, ссылка на NCBI)
Query - скэффолд 698.
© Борисов Евгений 2016