Результаты поиска гомологов представлены в следующей таблице:
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession | O34559 | 1NC5_A | YP_006631111.1 |
| E-value | 0.0 | 0.0 | 0.0 |
| Вес (в битах) | 780 | 780 | 780 |
| Процент идентичности | 100% | 100% | 100% |
2. Число находок с E-value < 10–10 |
3 | 5 | 100 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 3 | 5 | 97 |
| Accession | O31521.1 | 3PMM_A | ZP_07997326.1 |
| E-value | 1*10-20 | 2*10-12
| 1*10-88 |
|
| Вес (в битах) | 94.7 | 68.6 | 284 |
| % идентичности | 27% | 25% | 40% |
| % сходства | 45% | 42% | |
| Длина выравнивания | 344 | 382 | 399 |
| Координаты выравнивания (от-до, в запросе и в находке) | Query: 39-281; Sbjct: 29-262 | Query: 124-363; Sbjct: 148-376 | Query: 38-367; Sbjct: 58-397 |
| Число гэпов | 21 | 21 | 10 |
- С помощью программы BLASTP удалось найти исходный белок во всех базах данных.
- Число явных гомологов в разных базах данных принимает разное значение. Так, например, в банке Swiss-Prot было найдено 3 гомолога. Это прежде всего связано с тем, что банк Swiss-Prot состоит только из хорошо изученных белков. В банке PDB число явных гомологов равно 5. Их не так много, так как пространственная структура известна не для всех белков. Наибольшее количество гомологов найдено в базе данных nr. Это объясняется тем, что база nr включает все белковые последовательности из всевозможных источников.
- В базе данных Swiss-Prot найдено 4 гомолога, в PDB - 12, а в nr - 100. Число находок было лимитировано значением E-value.
Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
Гомологи были найдены в царстве Eukaryota. Поиск проводился по базе данных RefSeq, так как , как уже было сказано выше, в базе Swiss-Prot содержатся только три гомолога белка URHG2_BACSU.
Результаты поиска представлены в следующей таблице:
| Поиск по RefSeq | |
|---|---|
| Номер находки в списке описаний | 6 |
| Accession | XP_003026001.1 |
| E-value | 8-61 |
| Вес (в битах) | 207 |
| % идентичности | 36% |
| % сходства | 56% |
| Длина выравнивания | 328 |
| Координаты выравнивания (от-до, в запросе и в находке) | Query: 55-365; Sbjct: 1-328 |
| Число гэпов | 17 |
С помощью программы BLASTP были выровнены две последовательности белков O34559 и XP_003026001.1. Карты локального сходства приведены на рис.1 и рис.2 с порогом E-value=10 и E-value=0.01 соответственно. Как видно из рисунков, карты совсем не отличаются.
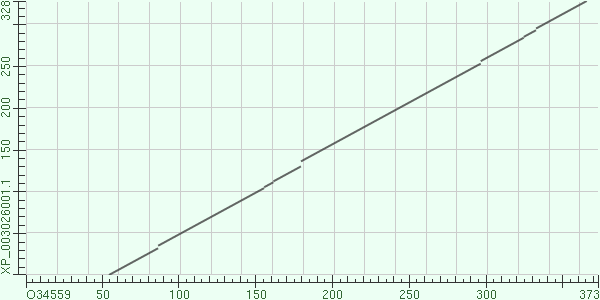
Рис.1. Карта локального сходства с порогом E-value=10
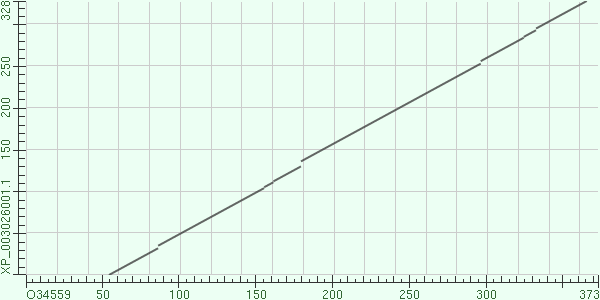
Рис.2. Карта локального сходства с порогом E-value=0.01