Составление репрезентативной выборки гомологов вашего белка с помощью BLAST
Поиск гомологов изучаемого белка проводился с помощью программы BLASTP по базе данных RefSeq.
Для поиска по прокариотам были изменены некоторые параметры. Во-первых, поиск проводился по всем организмам, кроме Eukaryota и Firmicutes. Во-вторых, было увеличено максимальное количество хитов до 1000 с порогом E-value=1. Однако, в результате этого запроса было получено 976 хитов, из которых еще было исключено 42, так как их E-value был больше 10-5. Таким образом, мы имеем 934 гомолога. Далее по всем этим белкам была получена выдача GenBank. К сожалению, GenBank отказался обработать все 934 гомолога, поэтому пришлось их разделить на две части. Я не получила единого дерева, пришлось довольствоваться двумя, представленными на рис.1 и рис.2. Из всех гомологов случайным образом было отобрано 27, каждый из которых принадлежит бактериям разных филумов.
Поиск по эукариотам проводился по всем организмам из царства Eukaryota с максимальным количеством хитов равным 1000 и с E-value=1. В результате запроса был получен 91 хит. Далее я получила распределение хитов по таксонам эукариот в виде дерева, которое представлено на рис.3. Параметры поиска представлены в таб.1. Из всех белков эукариот было отобрано 13 гомологов, принадлежащих разным царствам.
Далее было выяснено, что гены гомологов белка URHG2_BACSU локализированы в ядре.
Рис.1. |
Рис.2. |
Рис.3. |
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
|---|---|---|---|---|---|
| По прокариотам | BLASTP | RefSeq | все, кроме Firmicutes и Eukaryota | 1 | 1000 |
| По эукариотам | BLASTP | RefSeq | только Eukaryota | 1 | 1000 |
Таб.1. Параметры поиска хитов в программе BLASTP.
Отчет о встречаемости выбранных белков представлен в табл.2.
Табл.2. Встречаемость гомологов белка URHG2_BACSU в различных таксонах.
| Домен | Филум/Царство | Название организма | Количество белков |
|---|---|---|---|
| Archaea | Euryarchaeota | Haloferax sp. ATCC BAA-644 | 1 |
| Bacteria | Acidobacteria | Granulicella tundricola MP5ACTX9 | 1 |
| Actinobacteria | Amycolatopsis vancoresmycina | 1 | |
| Streptomyces sp. AA4 | 1 | ||
| Amycolatopsis decaplanina | 1 | ||
| Bacteroidales | Prevotella saccharolytica | 1 | |
| Bacteroides fluxus | 1 | ||
| Niabella soli | 1 | ||
| Mucilaginibacter paludis | 1 | ||
| Galbibacter sp. ck-I2-15 | 1 | ||
| Joostella marina | 1 | ||
| Galbibacter sp. ck-I2-15 | 1 | ||
| Pontibacter sp. BAB1700 | 1 | ||
| Echinicola vietnamensis DSM 17526 | 1 | ||
| Bacteroides sp. 1_1_14 | 1 | ||
| Fusobacteria | Fusobacterium mortiferum | 1 | |
| Sebaldella termitidis ATCC 33386 | 1 | ||
| Firmicutes | Bacillus subtilis strain 168 | 1 | |
| Ignavibacteriae | Melioribacter roseus P3M-2 | 1 | |
| Lentisphaerae | Lentisphaera araneosa | 1 | |
| Proteobacteria | Brenneria sp. EniD312 | 1 | |
| Pantoea sp. Sc1 | 1 | ||
| Novosphingobium nitrogenifigens | 1 | ||
| Rhizobium sp. CF080 | 1 | ||
| Spirochaetes | Treponema saccharophilum | 1 | |
| Spirochaeta thermophila DSM 6578 | 1 | ||
| Verrucomicrobia | Verrucomicrobiae bacterium DG1235 | 1 | |
| Opitutaceae bacterium TAV1 | 1 | ||
| Eukaryotes | Fungi | Aspergillus oryzae RIB40 | 1 |
| Leptosphaeria maculans JN3 | 1 | ||
| Thielavia terrestris NRRL 81 | 1 | ||
| Fusarium graminearum PH-1 | 1 | ||
| Magnaporthe oryzae 70-15 | 1 | ||
| Verticillium albo-atrum VaMs.102 | 1 | ||
| Debaryomyces hansenii CBS767 | 1 | ||
| Schizophyllum commune H4-8 | 1 | ||
| Cryptococcus gattii WM276 | 1 | ||
| Stramenopiles | Phytophthora infestans T30-4 | 1 | |
| Viridiplantae | Physcomitrella patens subsp. patens | 1 | |
| Cucumis sativus | 1 | ||
| Ricinus communis | 1 |
Множественное выравнивание гомологов белка URHG2_BACSU
С помощью программы Muscle было построено множественное выравнивание гомологов для эукариот и прокариот. Результаты представлены на рис.4 и в файле project.jar.
Рис.4. Множественное выравнивание гомологов белка URHG2_BACSU среди прокариот и эукариот.
Для построения выравнивания использовалась цветовая схема CLustalX, основанная на свойствах аминокислот. Значение cut-off равно 10%, это значит, что те участки выравнивания, для которых процент идентичности ниже 10, не окрашиваются. Последовательность белка URHG2_BACSU была проассоциирована с соответствующим PDB-файлом для нанесения информации из структуры на выравнивание. Таким образом были созданы три новые строки аннотации: на строке SECONDARY отображается втричная структура белка, на BLOCKS символом "В" показаны те участки выравнивания, которые похожи на блоки, а на строке LIGAND символом "L" указаны остатки белка, связывающие единственный лиганд UCD.
Результаты анализа множественного выравнивания гомологов белка URHG2_BACSU
Данное выравнивание можно назвать достаточно консервативными. Хуже всего выровнены концевые участки белков. Это связано с тем, что белок представляет собой глобулу, концевые участки которого выдаются наружу, а значит, вероятность того, что они могут претерпеть различные изменения выше, чем , например, у участков внутри глобулы. Следовательно, более консервативные участки расположены в центре глобулы.
Как и следовало ожидать, элементы вторичной структуры взаимосвязаны с участками консервативности. Такую особенность можно обосновать тем, что функции белка напрямую связаны со вторичной структурой. Так как участки, отвечающие элементам вторичной структуры, консервативны, то представленные гомологи имеют схожие функции.
На элементы вторичной структуры приходится мало столбцов-гэпов, по сравнению с другими участками белков.
Изучая выравнивание, можно заметить, что среди аминокислот, связывающих лиганд есть как консервативные, так и неконсервативные. С лигандом связываются следующие аминокислоты: His40, Tyr41, His42, Ser334, Lys133, Tyr136. Из них консервативными являются Tyr41 и Tyr136. Рассмотрим каждую из этих аминокислот:
- His40. Можно заметить, что в колонке выравнивания, соответствующей этой аминокислоте, в основном встречаются гидрофильные отрицательно заряженные аминокислоты. В большинстве случаев у эукариот это глутамат.
- Tyr41. Как было сказано ранее, это достаточно консервативная амнокислота. Если замена все-таки происходит, то чаще всего на триптофан, который так же, как и тирозин, является гидрофобной ароматической аминокислотой.
- His42. В колонке, соответствующей этой аминокислоте, встречаются аминокислоты, свойства которых очень отличаются. Однако у эукариот гистидин чаще всего заменен на треонин.
- Lys133. У выбранных прокариот лизин остается консервативным, что нельзя сказать об эукариотах, у которых в этом положении отсутствуют аминокислоты. Возможно эти белки утратили способность связывать лиганд UCD.
- Tyr136. Эта аминокислота в большинстве случаев консервативна.
- Ser334. В этом положении чаще всего втречаются пролин и глицин. Обе аминокислоты гидрофильны.
Таким образом, нельзя сказать, что все аминокилоты, связывающие лиганд, консервативны. Это может значить то, что приведенные гомологи утратили способность связывать лиганд UCD.
На рис.5 представлена пространственная структура белка URHG2_BACSU. Сам белок представлен в виде cartoons, остатки связывающие лиганд выделены в виде wireframe и окрашены в те же цвета, что и во множественном выравнивании, а лиганд представлен в виде cpk с соответствующей окраской.
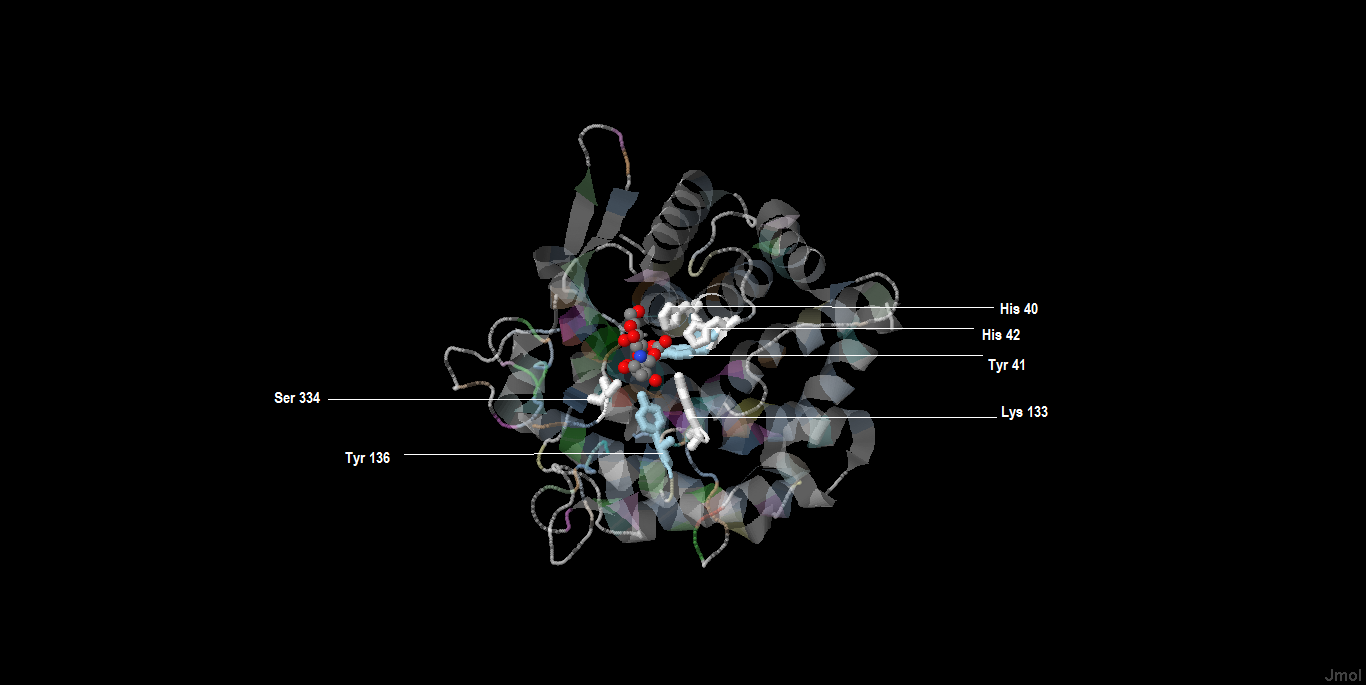
Рис.5. Пространственная структура URHG2_BACSU.
Дата последнего обновления: 27. 05. 2013
