А- и В-формы ДНК
А-, В- и Z-формы ДНК были построены с помощью инструментов пакета 3DNA. Последовательность одной из нитей ДНК представляет собой 5 раз повторенную последовательность "GATC". Структуры дуплекса в форме А, В и Z сохранены в файлах gatc-a.pdb, gatc-b.pdb и gatc-z.pdb соответственно.
На представленных ниже рисунках изображена А-форма ДНК. Цветом выделены различные атомы и химические группировки молекулы.
Рис.1. А-форма дуплекса ДНК. Желтым цветом выделен сахарофосфатный остов
Рис.2. А-форма дуплекса ДНК. Голубым цветом выделены все нуклеотиды.
Рис.3. А-форма ДНК. Фиолетовым цветом выделены все аденины.
Рис.4. А-форма ДНК. Голубым цветом выделены все гуанины. Желтым цветом выделены N7 атомы во всех гуанинах.
Проверка данных структур ДНК и РНК на наличие разрывов
Далее на сайте PDB были получены структуры 1O0C (рис.5) и 1P47 (рис.7), содержащие РНК и ДНК соответственно. Выбранные структуры сохранены в файлах 1O0C.pdb и 1P47.pdb. На рис.6 и рис.8 представлены изображения только нуклеиновых кислот в проволочной модели. Внимательно рассмотрев структуры, можно заметить, что аденозинмонофосфат на рис.9 отделен от остальной структуры 1O0C, то есть РНК имеет разрыв. В файлах 1O0C.txt и 1P47.txt сохранены координаты атомов РНК и ДНК соответственно.
Рис.5. Кристаллическая структура L-глутамата и AMPCPP, связанная с глутаминовой аминоацил-тРНК-синтетазой (PDB ID: 1O0C)
Рис.6. Нуклеиновая кислота в 1O0C
Рис.9. Оторвавшийся аденозинмонофосфат в структуре 1O0C
Рис.7. Кристаллическая структура тандемных молекул Zif268, связанных с ДНК (PDB ID: 1P47)
Рис.8. Нуклеиновая кислота в 1P47
Сравнение структур 3-х форм ДНК с помощью средств JMol
Как известно, в двойной спирали ДНК различают большую и малую бороздку. С помощью программы Jmol откроем полученные ранее файлы gatc-b.pdb, gatc-a.pdb и gatc-z.pdb и визуально определим бороздки. Результаты представлены на рис.10, рис.11 и рис.12.
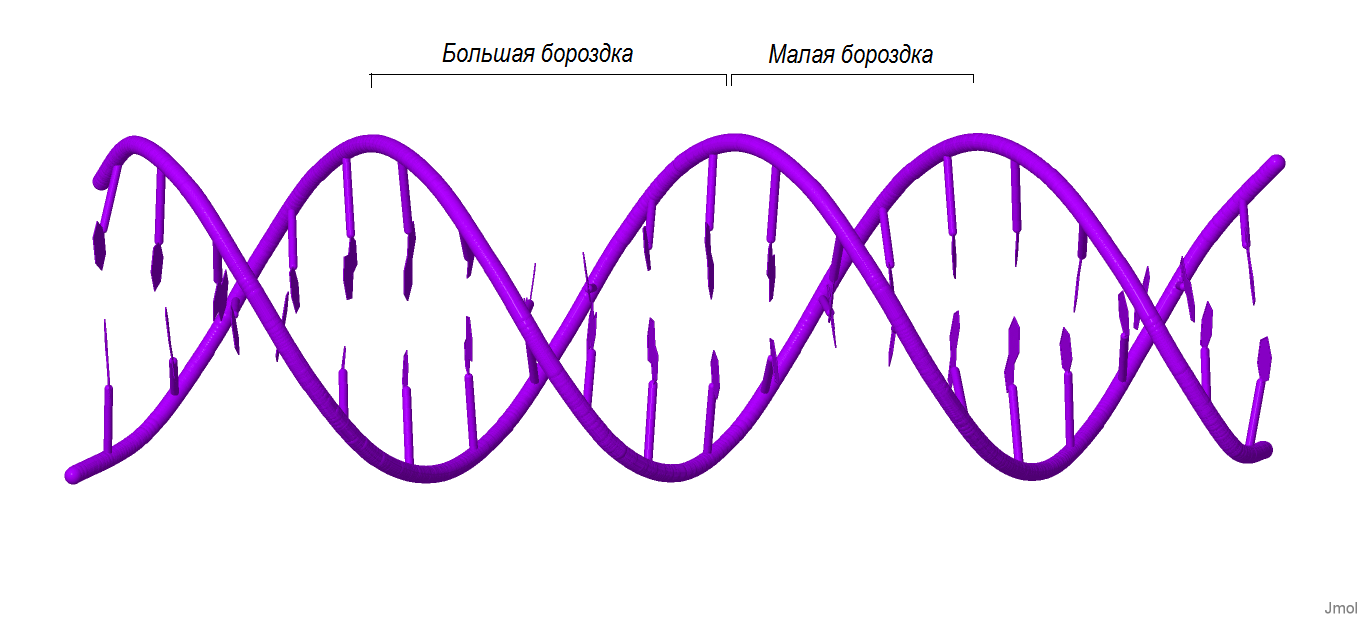
| Рис.10. B-форма ДНК |
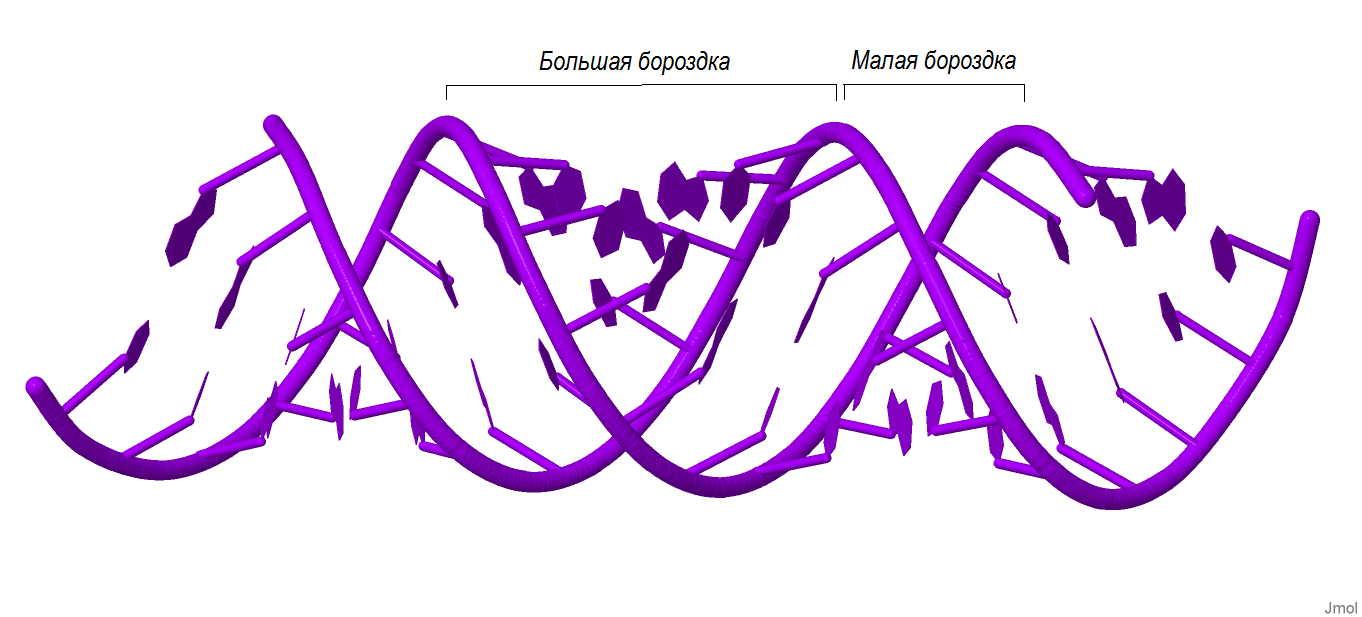
| Рис.11. A-форма ДНК |
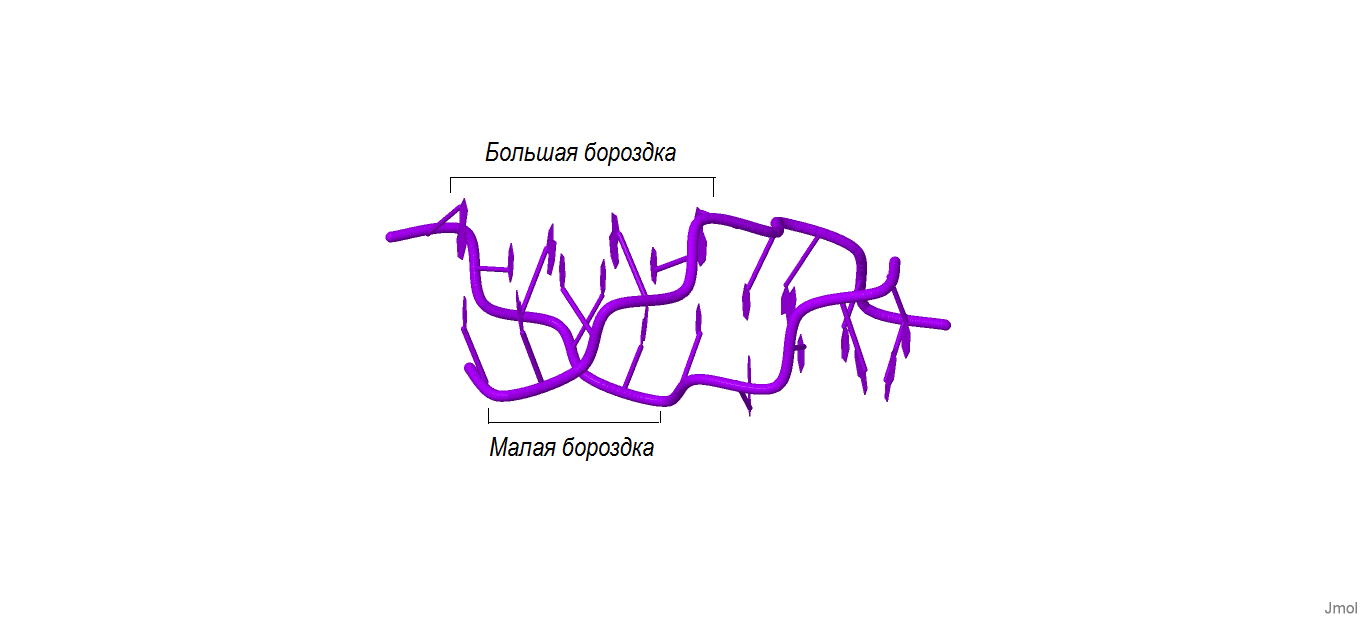
| Рис.12. Z-форма ДНК |
В В-форме ДНК выберем тимин T11. Выделим атомы, обращенные в сторону большой и в сторону малой бороздки. Красным цветом выделены атомы, смотрящие в сторону большой бороздки, синим - в сторону малой (рис.13).
С помощью ChemSketch получим изображение основания(рис.14). Выделим красным цветом атомы, смотрящие в сторону большой бороздки, синим в сторону малой.
Рис.13.Тимин в структуре B-формы ДНК
Рис.14. Структурная формула тимина
Рис.15. А-форма ДНК с выделенным Т11. Красным цветом окрашены атомы C4, O4, C5, C5M, C6, синим - N1, C2, O2, серым - N3.
| А-форма | В-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Â) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Â) | 16.97 ([A]26:B.P #515 - [A]18:A.P #315) | 17.21 ([G]5:A.P #83 - [G]33:B.P #657) | 17.6 ([G]5:A.P #83 - [G]15:B.P #288) |
| Ширина малой бороздки (Â) | 7.98 ([A]6:A.P #105 - [G]29:B.P #575) | 11.69 ([C]32:B.P #638 - [G]13:A.P #247) | 7.2 ([G]19:B.P #370 - [G]5:A.P #83) |
Сравнение торсионных углов в структурах А- и В-форм ДНК
С помощью программы Jmol были измерены торсионные углы тимидилового нуклеотида. Ниже в таблицах представлены результаты сравнения торсионных углов Т11 из А- и В-форм ДНК со значениями из таблицы.
Табл.1. Торсионные углы для Т11 и B-формы ДНК
| Угол | Значения для Т11 | Значения из презентации |
| α | 85.9 | 63 |
| β | 136.4 | 171 |
| γ | 31.1 | 54 |
| δ | 143.4 | 123/131 |
| ε | 105.9 | 155 |
| ξ | -44.7 | -90 |
| χ | -98.0 | -117 |
Табл.2. Торсионные углы для Т11 и A-формы ДНК
| Угол | Значения для Т11 | Значения из презентации |
| α | 64.1 | 62 |
| β | 174.8 | 173 |
| γ | 41.7 | 52 |
| δ | 79.1 | 88/3 |
| ε | 100.4 | 178 |
| ξ | -75.1 | -50 |
| χ | -157.2 | -160 |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Определим значения торсионных углов в структурах А-, В- и Z-форм ДНК (файлы gatc-b.pdb, gatc-а.pdb, gatc-z.pdb). Для этого используем команды пакета 3DNA.
remediator --old gatc-b.pdb > gatc-b_old.pdb find_pair -t gatc-b_old.pdb stdout | analyze
Таким образом были получены файлы gatc-a_old.out, gatc-b_old.out, gatc-z_old.out. Если внимательно изучить таблицы с информацией о торсионных углах, можно заметить, что между формами А и В сильные различия в торсионных углах δ, ξ и χ. Что касается формы Z, она отличается от форм А и В почти по всем торсионным углам.
Табл.3. Среднее значение торсионных углов для структуры тРНК 1О0С.
| alpha | beta | gamma | delta | epsilon | epsilon | chi | |
| Strand 1 | -59.8 | 145.3 | 67.7 | 81.8 | -137.2 | -62.1 | -161.6 |
| Strand 2 | -65.8 | 164.2 | 57.2 | 83.6 | -146.8 | -71.4 | -166.7 |
Таким образом, мы видим, что заданная структура тРНК по своим значениям торсионных углов похода на А-форму ДНК.
Рис.16. Рассчет отклонений значений торсионных углов для первой цепи ДНК. Оранжевым цветом выделены углы тех оснований первой цепи, для которых по данному углу наблюдается наибольшее отклонение. Красныйм цветом выделены краевые нуклеотиды.Фиолетовым цветом выделено самое большое суммарное отклонение для нуклеотидов.
Рис.17.Рассчет отклонений значений торсионных углов для второй цепи ДНК. Оранжевым цветом выделены углы тех оснований первой цепи, для которых по данному углу наблюдается наибольшее отклонение. Красныйм цветом выделены краевые нуклеотиды.Фиолетовым цветом выделено самое большое суммарное отклонение для нуклеотидов.
Из рис.16 и рис.17 видно, что самый деформированный нуклеотид по сумме отклонений C17 из второй цепи.
Рис.18. Схема расположения стеблей. голубым цветом обозначен акцепторный стебель, желтым - Т-стебель, серым - антикодоновый стебель, розовым - D-стебель.
В том же файле найдем информацию о неканонических парах.Результаты представлены на рис.19.
Рис.19. Расположение неканонических пар. Неканонические пары выделены зеленым цветом.
Определим, есть дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру (для этого следует рассмотреть комплементарные пары, не имеющие отношения к стеблям). Результаты представлены на рис.20.
Рис.20. Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру. Пары выделены серым цветом.
В полученном ранее файле 1O0C_old.out найдем информацию о последовательном перекрывании пар оснований. На рис.21 видно, что что 11-ая пара имеет максимальное перекрываение - 11.35 квадратных ангстрем.
Рис.20. Данные о перекрывании пар оснований, полученные командой find_pair и analyze. Серым цветом выделена пара с максимальным перекрыванием.
Информацию о пространственной структуре интересующей нас пары можно получить из файла stacking.pdb, который генерируется программой find_pair, использованной нами ранее. В этом файле есть pdb модели всех перекрывающихся попарно пар. Вычленим координаты нужной нам пары в файл 11pair.pdb. Визуализируем пару с помощью Jmol(рис.22) и staking2img(рис.22).
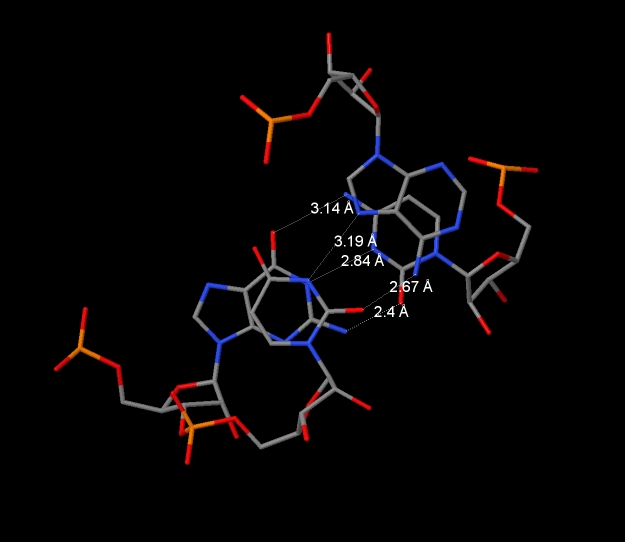
Рис.21. 11-ая пара с максимальным перекрыванием в Jmol.
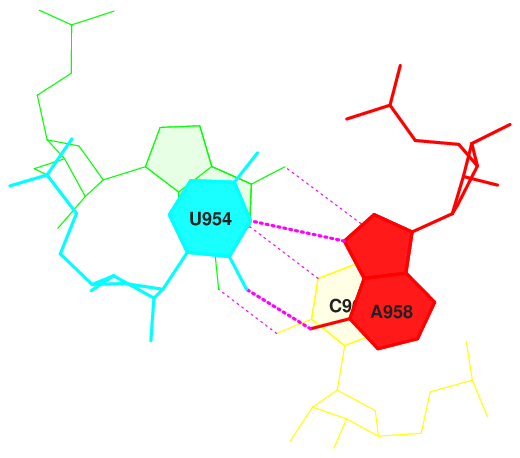
Рис.22. 11-ая пара с максимальным перекрыванием в Jmol.